
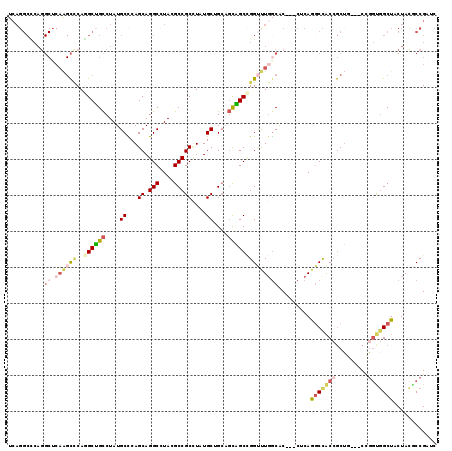
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,878,131 – 12,878,241 |
| Length | 110 |
| Max. P | 0.633000 |

| Location | 12,878,131 – 12,878,241 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 77.22 |
| Mean single sequence MFE | -44.45 |
| Consensus MFE | -25.67 |
| Energy contribution | -27.07 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.546306 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
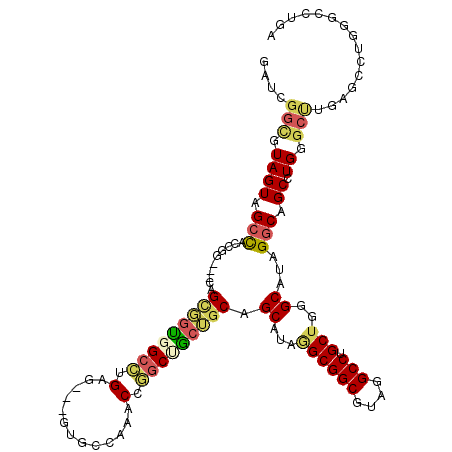
>2R_DroMel_CAF1 12878131 110 + 20766785 UCAGGCGCAGGCUCAAGCCCAUGCUGUCUAUGCCCAGCAGGCCUACGCCGCCUAUGCCG---CAGCCGGUUUGGCAC---CUCAGGCCACCGCUA---CAGGUAGCUACUACGCCGAUC ...((((.((((.((((((..(((((........)))))(((....((.((....)).)---).)))))))))))..---....(((.(((....---..))).))).)).)))).... ( -39.50) >DroPse_CAF1 1302 119 + 1 CCAGGCCCAGGCUCAGGCACAGGCGGCCUAUGCUCAGCAGGCUUAUGCCGCAUAUGCGGCCGCUGCAGGACUCACGCACACACAGCCCACGGCAGCCGCUGGCGGCUACUAUGCCGAUC ...(((....)))..((((.((..((((...((.((((.((((...(((((....))))).((((..((.((...........))))..)))))))))))))))))).)).)))).... ( -47.10) >DroSec_CAF1 1272 113 + 1 UCAGGCCCAGGCUCAAGCCCAGGCUGCCUAUGCCCAGCAGGCCUACGCCGCCUAUGCUGCAGCAGCCGGUUUGGCAC---CUCAGGCCACCGCUA---CAGGUGGCUACUACGCCGAUC ...(((..((((.((((((..((((((...(((..(((((((.......)))..)))))))))))))))))))))..---....(((((((....---..))))))).))..))).... ( -49.40) >DroEre_CAF1 1359 113 + 1 UCAGGCCCAGGCGCAAGCCCAGGCUGCCUAUGCCCAGCAGGCCUACGCCGCCUAUGCUGCAGUGGCCGGUUUGGCAC---CUCAGGCCACCGCUG---CCAGUGGCUACUACACCGAUC ...((((..(((....))).((((.((.((.(((.....))).)).)).))))..((((((((((..(((((((...---.))))))).))))))---.))))))))............ ( -47.40) >DroYak_CAF1 1368 113 + 1 UCAGGCUCAGGCUCAAGCUCAGGCUGCCUAUGCCCAGCAGGCCUACGCCGCUUAUGCUGCAGCAGCCGGUUUGGCGC---CGCAGGCCACCGCUG---CCGGUGGCUACUACACCGAUC ...((....((((((((((..((((((....((..(((.(((....))))))...))....))))))))))))).))---)...((((((((...---.))))))))......)).... ( -48.40) >DroAna_CAF1 1310 96 + 1 -----------------UCCAAGCGGCCUAUGCUCAGCAGGCCUACGCCGCUUAUGCCGCAGCUGCUGGCCUGGCAC---CACAAGCUGCCACUG---CCGCCGGCUACUACCCCGAUC -----------------.....(((((.((.....(((.(((....)))))))).)))))(((((.((((.(((((.---(....).)))))..)---))).)))))............ ( -34.90) >consensus UCAGGCCCAGGCUCAAGCCCAGGCUGCCUAUGCCCAGCAGGCCUACGCCGCCUAUGCUGCAGCAGCCGGUUUGGCAC___CUCAGGCCACCGCUG___CCGGUGGCUACUACGCCGAUC ..........((.((((((..((((((....((...((.(((....)))))....))....)))))))))))))).........(((((((.........)))))))............ (-25.67 = -27.07 + 1.39)


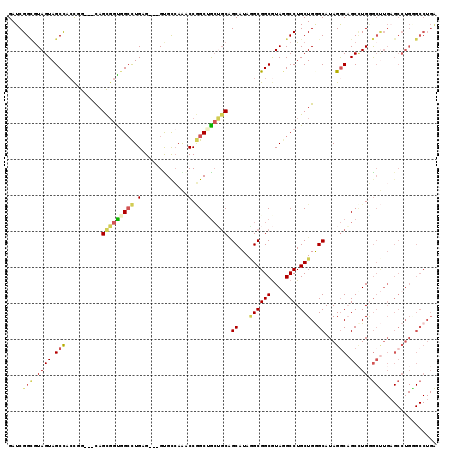
| Location | 12,878,131 – 12,878,241 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 77.22 |
| Mean single sequence MFE | -53.73 |
| Consensus MFE | -28.26 |
| Energy contribution | -28.77 |
| Covariance contribution | 0.51 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.633000 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12878131 110 - 20766785 GAUCGGCGUAGUAGCUACCUG---UAGCGGUGGCCUGAG---GUGCCAAACCGGCUG---CGGCAUAGGCGGCGUAGGCCUGCUGGGCAUAGACAGCAUGGGCUUGAGCCUGCGCCUGA ....(((((((..((..((((---(.((.(..(((...(---((.....))))))..---).)))))))..)).(((((((((((........))))..)))))))...)))))))... ( -53.60) >DroPse_CAF1 1302 119 - 1 GAUCGGCAUAGUAGCCGCCAGCGGCUGCCGUGGGCUGUGUGUGCGUGAGUCCUGCAGCGGCCGCAUAUGCGGCAUAAGCCUGCUGAGCAUAGGCCGCCUGUGCCUGAGCCUGGGCCUGG ..(((((((((((((((....))))))).((((.(((((((..(....)..).(((((.(((((....)))))....).))))...)))))).)))).)))))).))((....)).... ( -56.90) >DroSec_CAF1 1272 113 - 1 GAUCGGCGUAGUAGCCACCUG---UAGCGGUGGCCUGAG---GUGCCAAACCGGCUGCUGCAGCAUAGGCGGCGUAGGCCUGCUGGGCAUAGGCAGCCUGGGCUUGAGCCUGGGCCUGA ....(((......))).((((---(.((((..(((...(---((.....))))))..)))).((...((((((....))).)))..)))))))(((((..(((....)))..)).))). ( -54.80) >DroEre_CAF1 1359 113 - 1 GAUCGGUGUAGUAGCCACUGG---CAGCGGUGGCCUGAG---GUGCCAAACCGGCCACUGCAGCAUAGGCGGCGUAGGCCUGCUGGGCAUAGGCAGCCUGGGCUUGCGCCUGGGCCUGA ...(((((.......)))))(---(.(((((((((...(---((.....)))))))))))).)).((((((((((((((((...((((.......)))))))))))))))...))))). ( -59.10) >DroYak_CAF1 1368 113 - 1 GAUCGGUGUAGUAGCCACCGG---CAGCGGUGGCCUGCG---GCGCCAAACCGGCUGCUGCAGCAUAAGCGGCGUAGGCCUGCUGGGCAUAGGCAGCCUGAGCUUGAGCCUGAGCCUGA ....(((((.((((((((((.---...)))))).)))).---))))).....(((((((((.......))))).((((((.(((((((.......)))).)))..).)))))))))... ( -54.50) >DroAna_CAF1 1310 96 - 1 GAUCGGGGUAGUAGCCGGCGG---CAGUGGCAGCUUGUG---GUGCCAGGCCAGCAGCUGCGGCAUAAGCGGCGUAGGCCUGCUGAGCAUAGGCCGCUUGGA----------------- .......((.(((((..((((---(..(((((.(....)---.))))).))).)).))))).)).((((((((.((.(((....).)).)).))))))))..----------------- ( -43.50) >consensus GAUCGGCGUAGUAGCCACCGG___CAGCGGUGGCCUGAG___GUGCCAAACCGGCUGCUGCAGCAUAGGCGGCGUAGGCCUGCUGGGCAUAGGCAGCCUGGGCUUGAGCCUGGGCCUGA ....(((.((((.(((..........(((((((((.(.............).))))))))).((...((((((....))).)))..))...))).)).)).)))............... (-28.26 = -28.77 + 0.51)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:06:59 2006