
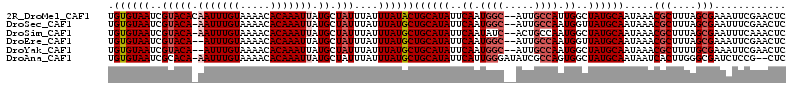
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,869,984 – 12,870,095 |
| Length | 111 |
| Max. P | 0.843380 |

| Location | 12,869,984 – 12,870,095 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 91.59 |
| Mean single sequence MFE | -24.67 |
| Consensus MFE | -20.39 |
| Energy contribution | -21.42 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.843380 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12869984 111 + 20766785 UGUGUAAUCGUACACAAUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUACUGCAUAUUCAAUGGC--AUUGCCAUUGGCUAUGCAAUAAACGCUUUAGCGAAAUUCGAACUC .(((((....)))))(((((((.....))))))).(((((....(((((..((((((..(((((((--...)))))))..))))))))))).....)))))............ ( -30.20) >DroSec_CAF1 3113 110 + 1 UGUGUAAUCGUACA-AAUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAAUGGC--AUUGCCAAUGGUUAUGCAAUAAACGCUUUAGCGAAUUUCGAACUC (((((.....((((-....))))..))))).....(((((....(((((..((((((..((.((((--...)))).))..))))))))))).....)))))............ ( -24.20) >DroSim_CAF1 2971 110 + 1 UGUGUAAUCGUACA-AAUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAAUAUC--ACUGCCAAUGGCUAUGCAAUAAACGCUUUAGCGAAUUUCAAACUC (((((.....((((-....))))..))))).....(((((....(((((..((((((..((.....--........))..))))))))))).....)))))............ ( -16.32) >DroEre_CAF1 3422 109 + 1 UGUGUAAUCGUACA--AUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAAUGGC--AUUGCCAAUGGUUAUGCAAUAAACGCUUUAGCGAAAUUCGAACUC (((((.....(((.--....)))..)))))((((.(((((....(((((..((((((..((.((((--...)))).))..))))))))))).....))))).))))....... ( -24.10) >DroYak_CAF1 3059 109 + 1 UGUGUAAUCGUACA--AUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAAUGGC--AUUGCCAAUGGCUAUGCAAUAAACGCUUUUGCGAAAUUCGAACUC .((((((..(((.(--((((((.....))))))).))).......))))))((((((..((.((((--...)))).))..)))))).....(((....)))............ ( -24.30) >DroAna_CAF1 3980 110 + 1 UGUGUAAUCGCACA-AAUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAUUGGGAUAUCGCCAGUGGCUAUGCAAUAAUCACUUGGGCGAUCUCCG--CUC .(((.....(((..-(((((((.....))))))).))).............((((((..(((((((....).))))))..)))))).....)))..(((((.....))--))) ( -28.90) >consensus UGUGUAAUCGUACA_AAUUUGUAAAACACAAAUUAUGCUAUUUAUUUAUGCUGCAUAUUCAAUGGC__AUUGCCAAUGGCUAUGCAAUAAACGCUUUAGCGAAAUUCGAACUC .((((((..(((((.(((((((.....))))))).)).)))....))))))((((((..((.((((.....)))).))..)))))).....(((....)))............ (-20.39 = -21.42 + 1.03)
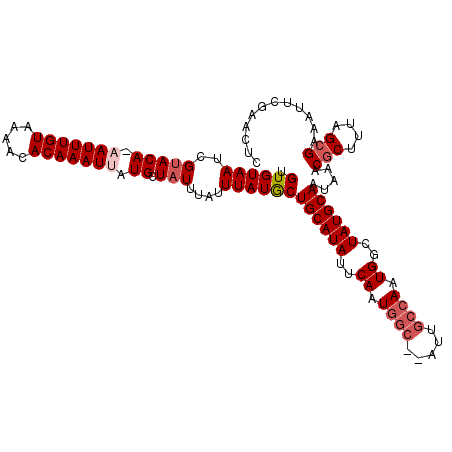
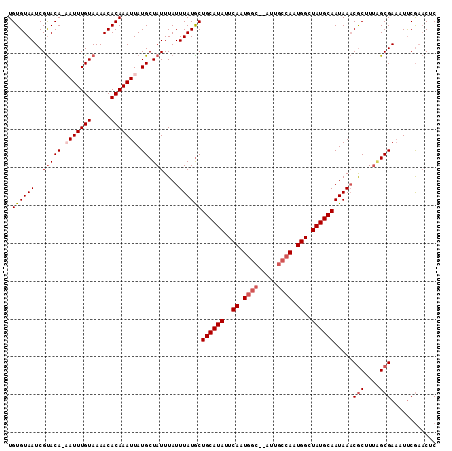
| Location | 12,869,984 – 12,870,095 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 91.59 |
| Mean single sequence MFE | -26.27 |
| Consensus MFE | -21.33 |
| Energy contribution | -21.75 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.752973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12869984 111 - 20766785 GAGUUCGAAUUUCGCUAAAGCGUUUAUUGCAUAGCCAAUGGCAAU--GCCAUUGAAUAUGCAGUAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAUUGUGUACGAUUACACA ....(((.....(((....)))..(((((((((..(((((((...--)))))))..)))))))))..........((((((((((((...)))))))))))).)))....... ( -34.90) >DroSec_CAF1 3113 110 - 1 GAGUUCGAAAUUCGCUAAAGCGUUUAUUGCAUAACCAUUGGCAAU--GCCAUUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAUU-UGUACGAUUACACA ((((.....))))((((....((((((((((((..((.((((...--)))).))..))))))..))))))...))))......(((((..((((....-)))).....))))) ( -23.70) >DroSim_CAF1 2971 110 - 1 GAGUUUGAAAUUCGCUAAAGCGUUUAUUGCAUAGCCAUUGGCAGU--GAUAUUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAUU-UGUACGAUUACACA (((((((....(((((...(((.....)))...(((...))))))--))............(((((((((..........)))))))))...))))))-)............. ( -18.50) >DroEre_CAF1 3422 109 - 1 GAGUUCGAAUUUCGCUAAAGCGUUUAUUGCAUAACCAUUGGCAAU--GCCAUUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAU--UGUACGAUUACACA ....(((......((((....((((((((((((..((.((((...--)))).))..))))))..))))))...))))((((((((((...)))))))--))).)))....... ( -26.60) >DroYak_CAF1 3059 109 - 1 GAGUUCGAAUUUCGCAAAAGCGUUUAUUGCAUAGCCAUUGGCAAU--GCCAUUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAU--UGUACGAUUACACA ....(((......((......((((((((((((..((.((((...--)))).))..))))))..)))))).....))((((((((((...)))))))--))).)))....... ( -24.70) >DroAna_CAF1 3980 110 - 1 GAG--CGGAGAUCGCCCAAGUGAUUAUUGCAUAGCCACUGGCGAUAUCCCAAUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAUU-UGUGCGAUUACACA (.(--((.....))).)..((((((..((((((..((.(((.(....)))).))..))))))((((((((....(((((.....)))))......)))-)))))))))))... ( -29.20) >consensus GAGUUCGAAUUUCGCUAAAGCGUUUAUUGCAUAGCCAUUGGCAAU__GCCAUUGAAUAUGCAGCAUAAAUAAAUAGCAUAAUUUGUGUUUUACAAAUU_UGUACGAUUACACA ....(((.....(((....)))....(((((((..((.((((.....)))).))..)))))))............(((.((((((((...)))))))).))).)))....... (-21.33 = -21.75 + 0.42)

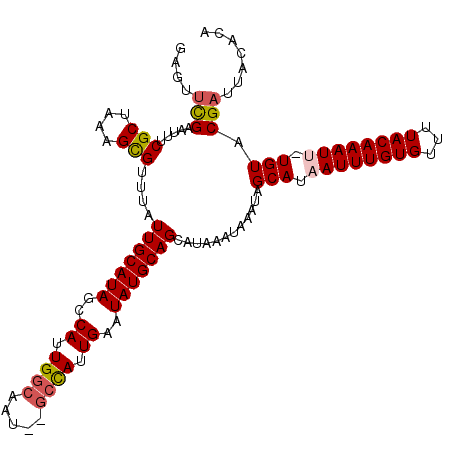
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:06:52 2006