
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,584,494 – 12,584,608 |
| Length | 114 |
| Max. P | 0.957837 |

| Location | 12,584,494 – 12,584,608 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.11 |
| Mean single sequence MFE | -51.64 |
| Consensus MFE | -35.58 |
| Energy contribution | -35.67 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.71 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.924971 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
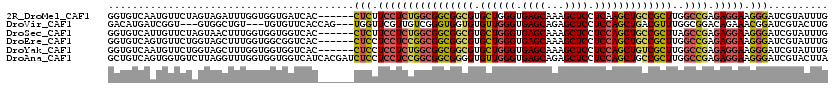
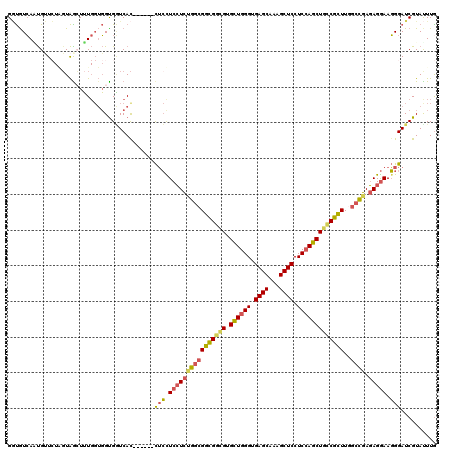
>2R_DroMel_CAF1 12584494 114 + 20766785 GGUGUCAAUGUUCUAGUAGAUUUGGUGGUGAUCAC------CUCUUCCUCUGGCGGCGGCGUGCUGGGUGAGCAAAGCUCCUCAAGCUGCCGCUUGGCCGAGAGGAAGGGAUCGUAUUUG (((((((.....((((.....))))...))).)))------)(((((((((((((((((((.((((((.((((...))))))).))))))))))..))).)))))))))........... ( -51.00) >DroVir_CAF1 14064 111 + 1 GACAUGAUCGGU---GUGGCUGU---UGUGUUCACCAG---UGGUUCGUUGUCGGGUGGUGUGUUGGGUGAGCAGAGCUCCUCCAGCUGACGUUUGGCGGACAGAAACGGAUCGUACUUG ...(((((((((---(.(((...---...)))))))..---((.(((.((((((..((....((((((.((((...)))).))))))...))..))))))...))).))))))))..... ( -36.10) >DroSec_CAF1 9639 114 + 1 GGUGUCAUUGUUCUAGUAACUUUGGUGGUGGUCAC------CUCUUCCUCUGGCGGCGGCGUGCUGGGUGAGCAAAGCUCCUCCAGCUGCCGCUUAGCCGAGAGGAAGGGAUCGUAUUUG ((((((((..((..((...))..))..)))).)))------)(((((((((((((((((((.((((((.((((...)))).)))))))))))))..))).)))))))))........... ( -57.00) >DroEre_CAF1 9665 114 + 1 GGUGUCAGUGUUCUGGUAGCUUUGGUGGCGGUCAC------CUCCUCCUCCGGCGGCGGCGUGCUGGGUGAGCAAAGCUCCUCCAGCUGCCGCUUGGCCGAGAGGAAGGGAUCGUAUUUG (.(..(((....)))..).).......((((((.(------((..((((((((((((((((.((((((.((((...)))).)))))))))))))..)))).))))))))))))))..... ( -59.70) >DroYak_CAF1 9642 114 + 1 GGUGUCAAUGUUCUGGUAGCUUUGGUGGUGGUCAC------CUCCUCCUCUGGCGGCGGCGUGCUGGGUGAGCAAAGCUCCUCCAGCUGUCGCUUGGCCGAGAGGAAGGGAUCGUAUUUG (((((((.....(..(.....)..)...))).)))------)(((((((((((((((((((.((((((.((((...)))).)))))))))))))..))).))))).)))).......... ( -50.50) >DroAna_CAF1 9976 120 + 1 GCUGUCAGUGGUGUCUUAGGUUUGGUGGUGGUCAUCACGAUCUCCUCCUCCGGCGGCGGGGUGUUGGGUGAGCAGAGCUCCUCCAGCUGCCGCUUGGCCGAGAGGAAGGGAUCGUACUUA .......((((((.((.............)).))))))((((((.((((((((((((((.(.((((((.((((...)))).))))))).)))))..)))).))))).))))))....... ( -55.52) >consensus GGUGUCAAUGUUCUAGUAGCUUUGGUGGUGGUCAC______CUCCUCCUCUGGCGGCGGCGUGCUGGGUGAGCAAAGCUCCUCCAGCUGCCGCUUGGCCGAGAGGAAGGGAUCGUAUUUG .........................................(((.((((((((((((((((.((((((.((((...)))).)))))))))))))..)))).))))).))).......... (-35.58 = -35.67 + 0.09)

| Location | 12,584,494 – 12,584,608 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.11 |
| Mean single sequence MFE | -42.03 |
| Consensus MFE | -30.11 |
| Energy contribution | -31.78 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.16 |
| Mean z-score | -3.43 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.957837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
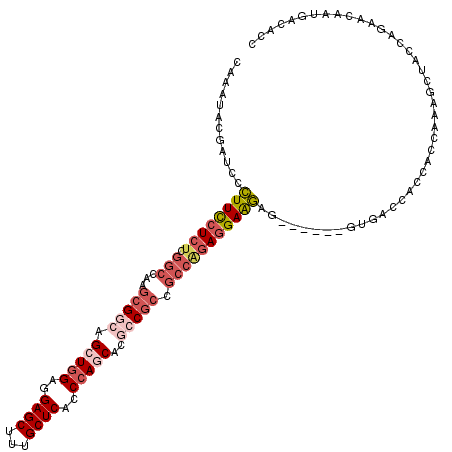
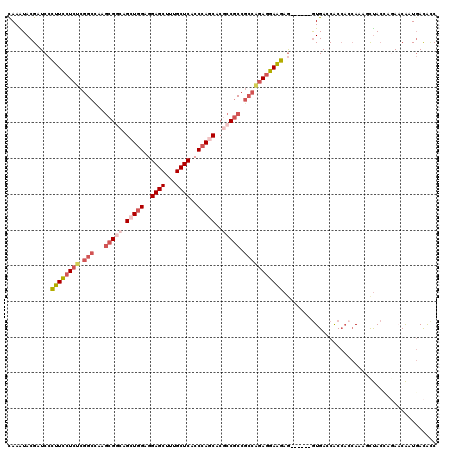
>2R_DroMel_CAF1 12584494 114 - 20766785 CAAAUACGAUCCCUUCCUCUCGGCCAAGCGGCAGCUUGAGGAGCUUUGCUCACCCAGCACGCCGCCGCCAGAGGAAGAG------GUGAUCACCACCAAAUCUACUAGAACAUUGACACC ......((((..((((((((.(((...(((((.(((.(..((((...))))..).)))..))))).))))))))))).(------(((.....))))..............))))..... ( -43.00) >DroVir_CAF1 14064 111 - 1 CAAGUACGAUCCGUUUCUGUCCGCCAAACGUCAGCUGGAGGAGCUCUGCUCACCCAACACACCACCCGACAACGAACCA---CUGGUGAACACA---ACAGCCAC---ACCGAUCAUGUC .......((((.((..((((.............(.(((..((((...))))..))).).(((((...............---.)))))......---))))....---)).))))..... ( -21.59) >DroSec_CAF1 9639 114 - 1 CAAAUACGAUCCCUUCCUCUCGGCUAAGCGGCAGCUGGAGGAGCUUUGCUCACCCAGCACGCCGCCGCCAGAGGAAGAG------GUGACCACCACCAAAGUUACUAGAACAAUGACACC ............((((((((.(((...(((((.(((((..((((...))))..)))))..))))).))))))))))).(------(((.....))))...((((.........))))... ( -48.90) >DroEre_CAF1 9665 114 - 1 CAAAUACGAUCCCUUCCUCUCGGCCAAGCGGCAGCUGGAGGAGCUUUGCUCACCCAGCACGCCGCCGCCGGAGGAGGAG------GUGACCGCCACCAAAGCUACCAGAACACUGACACC ............(((((((.((((...(((((.(((((..((((...))))..)))))..))))).))))))))))).(------(((.....))))........(((....)))..... ( -51.10) >DroYak_CAF1 9642 114 - 1 CAAAUACGAUCCCUUCCUCUCGGCCAAGCGACAGCUGGAGGAGCUUUGCUCACCCAGCACGCCGCCGCCAGAGGAGGAG------GUGACCACCACCAAAGCUACCAGAACAUUGACACC ......((((..((((((((((((...(((...(((((..((((...))))..))))).))).))))..)))))))).(------(((.....))))..............))))..... ( -42.80) >DroAna_CAF1 9976 120 - 1 UAAGUACGAUCCCUUCCUCUCGGCCAAGCGGCAGCUGGAGGAGCUCUGCUCACCCAACACCCCGCCGCCGGAGGAGGAGAUCGUGAUGACCACCACCAAACCUAAGACACCACUGACAGC ....(((((((.(((((((.((((...((((..(.(((..((((...))))..))).)...)))).))))))))))).)))))))................................... ( -44.80) >consensus CAAAUACGAUCCCUUCCUCUCGGCCAAGCGGCAGCUGGAGGAGCUUUGCUCACCCAGCACGCCGCCGCCAGAGGAAGAG______GUGACCACCACCAAAGCUACCAGAACAAUGACACC ............((((((((.(((...(((((.(((((..((((...))))..)))))..))))).)))))))))))........................................... (-30.11 = -31.78 + 1.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:04:38 2006