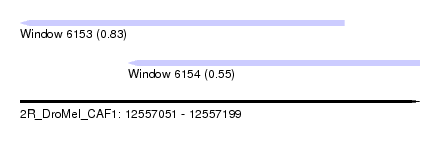
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,557,051 – 12,557,199 |
| Length | 148 |
| Max. P | 0.826384 |

| Location | 12,557,051 – 12,557,171 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 71.40 |
| Mean single sequence MFE | -48.88 |
| Consensus MFE | -24.41 |
| Energy contribution | -24.80 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.42 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.826384 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12557051 120 - 20766785 UACUAUUAUUGGUCGCUGGAGCCGGUGUAACCGUGGCUAUAGGCAGCAUUGGGGCCAAUGGUGUGGUAGUCAAUGUGGCUGUCGCAGCCAAUGGCUGUACUGUAUCAGUGCUCAACUCCA .((((....))))....(((((((((((..((.........))..)))))))(((((.(((((((..((((.....))))..)))).))).)))))((((((...))))))....)))). ( -45.40) >DroPse_CAF1 18875 120 - 1 UGCUGUUGCUUGUUGCAGGUGCGGGCGUGACUGUGGCUAUUGGCAGCAUCGGUGCCAAUGGUGUCGUUGUCACAGUCGCGGUGGCGGCCAUUGGCUGCGGGCACUCGGACGCCAGCUCCA .((((((((.....))))((.((((.(((((((((((...(((((.(((.(....).))).)))))..)))))))))))(.(.((((((...)))))).).).)))).))..)))).... ( -56.50) >DroWil_CAF1 38963 117 - 1 UGC---UGCUGGCAAUAGGCGCCGGCGUCACUGUAGCAAUGGGCAAAAGAGGCGCCAAUGGCGUUGUUGUCAUUGUUGCUGUGGCUGCCAUUGGCUGAACGCGAUCCGGUGUUAGAUCCA .((---.((((((.......))))))(((((.((((((((((.(((....((((((...)))))).))))))))))))).))))).))...(((...(((((......)))))....))) ( -47.00) >DroYak_CAF1 18510 120 - 1 UACUAUUAUUGGUCGCUGGAGCUGGUGUAACUGUGGCUAUAGGCAACAUUGGUGCCAAGGGAGUGGUAGUCAAUGUGGCUGUCGCAGCCAAUGGCUGUACUGUAUCGGUGCUCAACUCCA .........(((((((.(..((....))..).)))))))..((((.......))))...(((((...(((((...((((((...)))))).)))))((((((...))))))...))))). ( -40.00) >DroAna_CAF1 17499 111 - 1 UGC---------UCGCUGGAGCUGGGGUGACGGUGGCUAUUGGUAGCAUUGGAGCCAGAGGGGUUGUGGUAACCGUGGCAGUAGCUGCCAUCGGCUGUGGCACUUCGGAGGUCAGCUCCA ...---------.....(((((((((((..(((((.(((....))))))))..))).((((.((..(((...(((((((((...)))))).))))))..)).)))).....)))))))). ( -47.90) >DroPer_CAF1 18880 120 - 1 UGCUGUUGCUUGUUGCAGGUGCGGGCGUGACUGUGGCUAUUGGCAGCAUCGGUGCCAAUGGUGUCGUUGUCACAGUCGCGGUGGCGGCCAUUGGCUGCGGGCACUCGGACGCCAGCUCCA .((((((((.....))))((.((((.(((((((((((...(((((.(((.(....).))).)))))..)))))))))))(.(.((((((...)))))).).).)))).))..)))).... ( -56.50) >consensus UGCU_UUGCUGGUCGCAGGAGCCGGCGUGACUGUGGCUAUUGGCAGCAUUGGUGCCAAUGGUGUCGUUGUCACUGUGGCUGUGGCAGCCAUUGGCUGUACGCAAUCGGAGGUCAGCUCCA ...........(((((((..((....))..)))))))................(((((((((((((((((.......)))))))).)))))))))......................... (-24.41 = -24.80 + 0.39)

| Location | 12,557,091 – 12,557,199 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 82.84 |
| Mean single sequence MFE | -34.75 |
| Consensus MFE | -22.62 |
| Energy contribution | -23.27 |
| Covariance contribution | 0.65 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.549719 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12557091 108 - 20766785 AACUGAACUCCAGUAAGAGAGCUAACGUUACUAUUAUUGGUCGCUGGAGCCGGUGUAACCGUGGCUAUAGGCAGCAUUGGGGCCAAUGGUGUGGUAGUCAAUGUGGCU .((((..(((((((.....(((....)))((((....)))).))))))).))))((.((..(((((((..(((.(((((....))))).))).)))))))..)).)). ( -36.50) >DroVir_CAF1 23186 99 - 1 AGCUGAGCACC---------GCUGUCAUAGUUAUUACUUGUGGUUGGGGCUGGUGCUACAGUAGCUAUUGGGAGAAGCGGUGCCAGAGGCGUUGUAGUAACAAUUGCA ..(((.(((((---------(((.((.((((..(((((.(((((..........))))))))))..)))).))..)))))))))))..(((((((....)))).))). ( -35.00) >DroSec_CAF1 18487 108 - 1 AACUGAACUCCAGUCAGAGAGCUAACGUUACUAUUAUUAGUCGCUGGAGCCGGUGUAACCGUGGCUAUAGGCAACAUUGGGGCCAAAGGUGUGGUAGUCAAUGUGGCU ..((((.(....)))))..(((((.((((((((((((....(((((....)))))..(((.(((((...(....).....)))))..))))))))))).))))))))) ( -32.10) >DroSim_CAF1 18461 108 - 1 AACUGAACUCCAGUAAGAGAGCUAACGUUACUAUUAUUAGUCGCUGGAGCUGGUGUAACCGUGGCUAUAGGCAACAUUGGGGCCAAAGGUGUGGUAGUCAAUGUGGCU .((((.....)))).....(((((.(((((((((((((((((((.(..((....))..).)))))))..(((..(...)..)))......)))))))).))))))))) ( -30.50) >DroEre_CAF1 18296 108 - 1 AGCUGAAUUCCAGUAAUAGAGCUAACGUUACUAUUAUUGGUCGCUGGAGCCGGUGUAACCGUGGCUAUAGGCAACAUUGGGGCCAAGGGAGUGGUAGUCAAUGUGGCU ((((.....(((((((((((((....))).))))))))))..(((..((((((.....))..))))...))).(((((((.((((......))))..))))))))))) ( -39.30) >DroYak_CAF1 18550 108 - 1 AACUGAAUUCCAGUAAUAGAGCUAACGUUACUAUUAUUGGUCGCUGGAGCUGGUGUAACUGUGGCUAUAGGCAACAUUGGUGCCAAGGGAGUGGUAGUCAAUGUGGCU ..(((.....((((((((((((....))).)))))))))(((((.(..((....))..).)))))..)))((.((((((((((((......))))).))))))).)). ( -35.10) >consensus AACUGAACUCCAGUAAGAGAGCUAACGUUACUAUUAUUGGUCGCUGGAGCCGGUGUAACCGUGGCUAUAGGCAACAUUGGGGCCAAAGGUGUGGUAGUCAAUGUGGCU .((((.....))))......((((.((((((((((((...((..(((..(((((((..((.........))..)))))))..)))..)).)))))))).)))))))). (-22.62 = -23.27 + 0.65)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:04:30 2006