
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
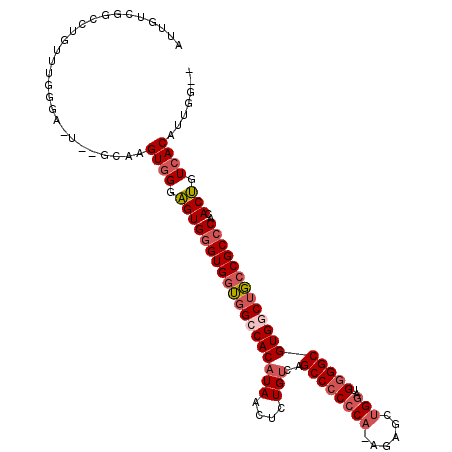
| Location | 12,313,171 – 12,313,281 |
| Length | 110 |
| Max. P | 0.939635 |

| Location | 12,313,171 – 12,313,281 |
|---|---|
| Length | 110 |
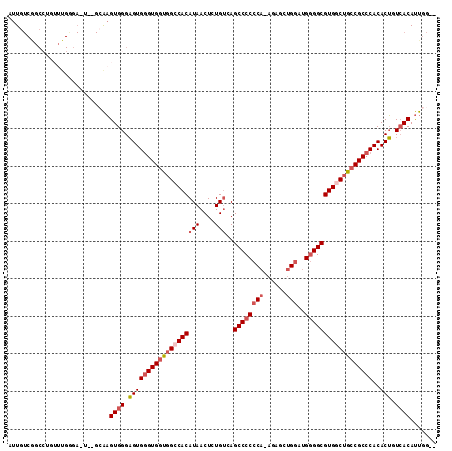
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.71 |
| Mean single sequence MFE | -46.60 |
| Consensus MFE | -35.45 |
| Energy contribution | -36.65 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939635 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
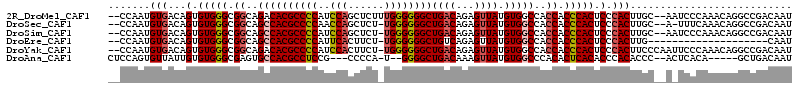
>2R_DroMel_CAF1 12313171 110 + 20766785 AUUGUCGGCCUGUUUGGGAUU--GCAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGUCAGCCCCCCAAAGAGCUGGAUGGGGCGUGUCUGCCGCCCACACUGUCACAUUGG-- ..((.(((((..((((.....--.))))..)).((((((((..(.((((((....)))..((((((((......)))..)))))))).)..)))))))).))).))......-- ( -45.90) >DroSec_CAF1 11930 108 + 1 AUUGUCGGCCUGUUUGAAA-U--GCAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGUCAGCCCCCCA-AGAGCUGGUUGGGGCGUGGCUGCCGCCCACACUGUCACAUUGG-- ..((.(((((..((((...-.--.))))..)).((((((((..((((((((....)))..((((((((-.....)))..))))))))))..)))))))).))).))......-- ( -52.50) >DroSim_CAF1 10058 109 + 1 AUUGUCGGCCUGUUUGGGAUU--GCAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGUCAGCCCCCCA-AGAGCUGGAUGGGGCGUGGCUGCCGCCCACACUGUCACAUUGG-- ..((.(((((..((((.....--.))))..)).((((((((..((((((((....)))..((((((((-.....)))..))))))))))..)))))))).))).))......-- ( -52.80) >DroEre_CAF1 10280 91 + 1 AUUG--------------------CAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGACAGCCCCCCA-AGAAGUGAAUGGGGCGUGGCUGCCGCCCACACUGUCACAUUGG-- ....--------------------(((((((.(((((((((..(((((.((....))...(((((.((-.....))...))))))))))..))))))..))).)))).))).-- ( -42.40) >DroYak_CAF1 10272 111 + 1 AUUGUCGGCCUGUUUGGGAAUUGGGAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGUCAGCCCCCCA-AGAAGUGGAUGGGGCGUGUCUGCCGCCCACACUGUCACAUUGG-- ..((.(((((..(((..........)))..)).((((((((..(.((((((....)))..((((((((-.....)))..)))))))).)..)))))))).))).))......-- ( -41.60) >DroAna_CAF1 9802 101 + 1 AUUGUCAGC-----UGUGAGU--GGGUGUGGGUGUGAGUGUGGGCCACAUAACUUUGUCAGCCCC--A-UGGGG---CGGAGGCGUGGCACUCGCCCACACAAUAACACUGGAG ....((((.-----(((....--..((((((((..((((((.(((((((......)))..((((.--.-..)))---)...))).).))))))))))))))....))))))).. ( -44.40) >consensus AUUGUCGGCCUGUUUGGGA_U__GCAAGUGGGAGUGGGUGGUGGCCACAUAACUCUGUCAGCCCCCCA_AGAGCUGGAUGGGGCGUGGCUGCCGCCCACACUGUCACAUUGG__ ...........................((((.(((((((((((((((((((....)))..((((((((......)))..))))))))))))))))))..))).))))....... (-35.45 = -36.65 + 1.20)

| Location | 12,313,171 – 12,313,281 |
|---|---|
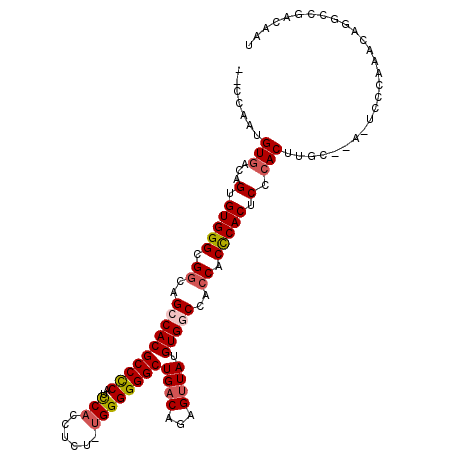
| Length | 110 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.71 |
| Mean single sequence MFE | -34.70 |
| Consensus MFE | -28.40 |
| Energy contribution | -29.10 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.677657 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12313171 110 - 20766785 --CCAAUGUGACAGUGUGGGCGGCAGACACGCCCCAUCCAGCUCUUUGGGGGGCUGACAGAGUUAUGUGGCCACCACCCACUCCCACUUGC--AAUCCCAAACAGGCCGACAAU --((((.((....(((.(((((.......))))))))...))...))))(((..((...((((...((((.......))))....)))).)--)..)))............... ( -32.30) >DroSec_CAF1 11930 108 - 1 --CCAAUGUGACAGUGUGGGCGGCAGCCACGCCCCAACCAGCUCU-UGGGGGGCUGACAGAGUUAUGUGGCCACCACCCACUCCCACUUGC--A-UUUCAAACAGGCCGACAAU --..((((..(..(.(((((.((..((((((((((..((((...)-))))))))((((...)))).)))))..)).))))).)....)..)--)-))................. ( -37.00) >DroSim_CAF1 10058 109 - 1 --CCAAUGUGACAGUGUGGGCGGCAGCCACGCCCCAUCCAGCUCU-UGGGGGGCUGACAGAGUUAUGUGGCCACCACCCACUCCCACUUGC--AAUCCCAAACAGGCCGACAAU --.(((.(((...(.(((((.((..((((((((((..((((...)-))))))))((((...)))).)))))..)).))))).).)))))).--..................... ( -36.40) >DroEre_CAF1 10280 91 - 1 --CCAAUGUGACAGUGUGGGCGGCAGCCACGCCCCAUUCACUUCU-UGGGGGGCUGUCAGAGUUAUGUGGCCACCACCCACUCCCACUUG--------------------CAAU --......((.(((.((((..(((((((...(((((.........-)))))))))))).((((...((((...))))..)))))))))))--------------------)).. ( -34.80) >DroYak_CAF1 10272 111 - 1 --CCAAUGUGACAGUGUGGGCGGCAGACACGCCCCAUCCACUUCU-UGGGGGGCUGACAGAGUUAUGUGGCCACCACCCACUCCCACUUCCCAAUUCCCAAACAGGCCGACAAU --((...(((...(((.(((((.......)))))))).)))...(-(((((((.((...((((...((((...))))..)))).))))))))))..........))........ ( -32.30) >DroAna_CAF1 9802 101 - 1 CUCCAGUGUUAUUGUGUGGGCGAGUGCCACGCCUCCG---CCCCA-U--GGGGCUGACAAAGUUAUGUGGCCCACACUCACACCCACACCC--ACUCACA-----GCUGACAAU .....(((.....(((((((.((((((((((.....(---(((..-.--.))))((((...))))))))))....))))...)))))))..--...))).-----......... ( -35.40) >consensus __CCAAUGUGACAGUGUGGGCGGCAGCCACGCCCCAUCCACCUCU_UGGGGGGCUGACAGAGUUAUGUGGCCACCACCCACUCCCACUUGC__A_UCCCAAACAGGCCGACAAU .......(((...(.(((((.((..((((((((((..(((......))))))))((((...)))).)))))..)).))))).).)))........................... (-28.40 = -29.10 + 0.70)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:02:29 2006