
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,286,514 – 12,286,616 |
| Length | 102 |
| Max. P | 0.971826 |

| Location | 12,286,514 – 12,286,616 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.37 |
| Mean single sequence MFE | -38.15 |
| Consensus MFE | -19.09 |
| Energy contribution | -21.28 |
| Covariance contribution | 2.20 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.50 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925738 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
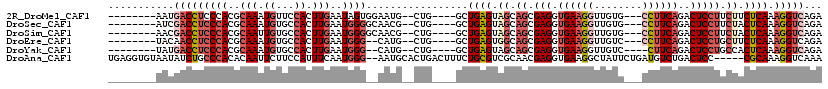
>2R_DroMel_CAF1 12286514 102 + 20766785 --------AAUGACCUCCCACGCAAAUGUUCCACUUGAAUAGUGGAAUG--CUG----GCUGAGUAGCAGCGAGGUGAAGGUUGUG---CCUUCAGACUCCUUCUUCUCAAAGGUCAGA --------..((((((.(((.(((....(((((((.....)))))))))--)))----).((((.((.((.(((.((((((.....---))))))..))))).)).)))).)))))).. ( -40.30) >DroSec_CAF1 150094 102 + 1 --------AUCGACCUCCCACGCAAAUGUGCCACUUGAAUGGGGCAACG--CUG----GCUGAGUAGCAGCGAGGUGAAGGUUGUG---CCUUCAGACUCCUUCUACUCAAAGGUCAGA --------...(((((.(((.((...(((.(((......))).)))..)--)))----).(((((((.((.(((.((((((.....---))))))..))))).))))))).)))))... ( -41.20) >DroSim_CAF1 143869 102 + 1 --------AACGACCUCCCACGCAAUUGUGCCACUUGAAUGGGGCAACG--CUG----GCUGAGUAGCAGCGAGGUGAAGGUUGUG---CCUUCAGACUCCUUCUACUCAAAGGUCAGA --------...(((((.(((.((..((((.(((......))).)))).)--)))----).(((((((.((.(((.((((((.....---))))))..))))).))))))).)))))... ( -42.00) >DroEre_CAF1 156448 100 + 1 --------UACAACCUCCCACGCAAAUGUGCCACUUGAAUGGG--CAUG--CUG----GCUGAGUGGCAGCGAGGUGAAGGUUGUC---CCUUCAGACUCCUGCUUCUCAAAGGUCAGA --------....((((.(((.(((....((((.(......)))--))))--)))----).((((.(((((.(((.((((((.....---))))))..)))))))).)))).)))).... ( -37.20) >DroYak_CAF1 152120 99 + 1 --------UAUGACCUCCCACGCAAAUGUGCCACUUGAAUGGG--CAUG--CUG----GCUGAGUAGCAGCGAGGUGAAGGUUGUC----CUUCAGACUCCUGCCACUCAAAGGUCAGA --------..((((((.(((.(((....((((.(......)))--))))--)))----).(((((.((((.(((.((((((....)----)))))..))))))).))))).)))))).. ( -43.00) >DroAna_CAF1 141135 112 + 1 UGAGGUGUAAUAUCUGCCCACACAAUUCUUCCAUUUCAAUGGG--AAUGCACUGACUUUCUGCGUCGCAACGAGGUGAAGGCUAUUCUGAUGUCUGACUCC-----CGCAAAGGUCAAA ...((((((.....((......))....((((((....)))))--).))))))((((((.((((((((......))))((((.........))))......-----))))))))))... ( -25.20) >consensus ________AACGACCUCCCACGCAAAUGUGCCACUUGAAUGGG__AAUG__CUG____GCUGAGUAGCAGCGAGGUGAAGGUUGUG___CCUUCAGACUCCUUCUACUCAAAGGUCAGA ...........(((((((((..(((.((...)).)))..)))).................((((.((.((.(((.((((((........))))))..))))).)).)))).)))))... (-19.09 = -21.28 + 2.20)
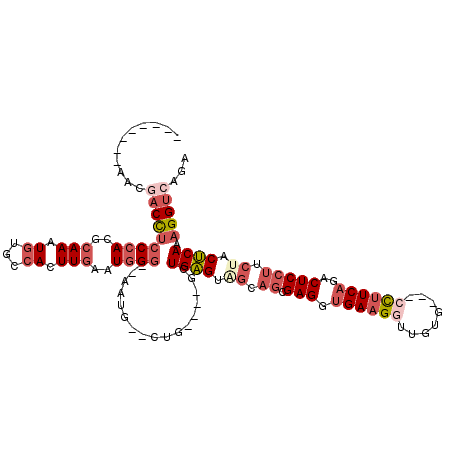
| Location | 12,286,514 – 12,286,616 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.37 |
| Mean single sequence MFE | -38.45 |
| Consensus MFE | -24.41 |
| Energy contribution | -26.13 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971826 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
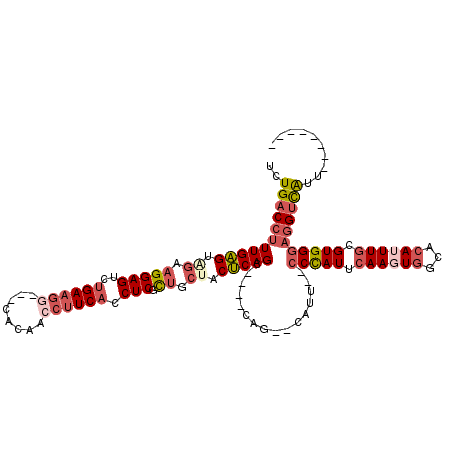
>2R_DroMel_CAF1 12286514 102 - 20766785 UCUGACCUUUGAGAAGAAGGAGUCUGAAGG---CACAACCUUCACCUCGCUGCUACUCAGC----CAG--CAUUCCACUAUUCAAGUGGAACAUUUGCGUGGGAGGUCAUU-------- ..(((((((((((.((.(((((..((((((---.....)))))).))).)).)).)))).(----(((--(((((((((.....)))))))....))).))))))))))..-------- ( -39.70) >DroSec_CAF1 150094 102 - 1 UCUGACCUUUGAGUAGAAGGAGUCUGAAGG---CACAACCUUCACCUCGCUGCUACUCAGC----CAG--CGUUGCCCCAUUCAAGUGGCACAUUUGCGUGGGAGGUCGAU-------- ..((((((((((((((.(((((..((((((---.....)))))).))).)).)))))))).----...--......(((((.((((((...)))))).)))))))))))..-------- ( -40.60) >DroSim_CAF1 143869 102 - 1 UCUGACCUUUGAGUAGAAGGAGUCUGAAGG---CACAACCUUCACCUCGCUGCUACUCAGC----CAG--CGUUGCCCCAUUCAAGUGGCACAAUUGCGUGGGAGGUCGUU-------- ...(((((((((((((.(((((..((((((---.....)))))).))).)).))))))).(----(((--(((((..((((....))))..))).))).)))))))))...-------- ( -41.10) >DroEre_CAF1 156448 100 - 1 UCUGACCUUUGAGAAGCAGGAGUCUGAAGG---GACAACCUUCACCUCGCUGCCACUCAGC----CAG--CAUG--CCCAUUCAAGUGGCACAUUUGCGUGGGAGGUUGUA-------- ...((((((((((..(((((((..((((((---.....)))))).))).))))..))))).----...--....--(((((.((((((...)))))).))))))))))...-------- ( -39.00) >DroYak_CAF1 152120 99 - 1 UCUGACCUUUGAGUGGCAGGAGUCUGAAG----GACAACCUUCACCUCGCUGCUACUCAGC----CAG--CAUG--CCCAUUCAAGUGGCACAUUUGCGUGGGAGGUCAUA-------- ..((((((((((((((((((((..(((((----(....)))))).))).))))))))))).----...--....--(((((.((((((...)))))).)))))))))))..-------- ( -47.30) >DroAna_CAF1 141135 112 - 1 UUUGACCUUUGCG-----GGAGUCAGACAUCAGAAUAGCCUUCACCUCGUUGCGACGCAGAAAGUCAGUGCAUU--CCCAUUGAAAUGGAAGAAUUGUGUGGGCAGAUAUUACACCUCA (((((..(((((.-----...).))))..)))))...((((.(((((..((((...))))..))((((((....--..))))))............))).))))............... ( -23.00) >consensus UCUGACCUUUGAGUAGAAGGAGUCUGAAGG___CACAACCUUCACCUCGCUGCUACUCAGC____CAG__CAUU__CCCAUUCAAGUGGCACAUUUGCGUGGGAGGUCAUU________ ..(((((((((((.((.(((((..((((((........)))))).))).)).)).)))))................(((((.((((((...)))))).))))))))))).......... (-24.41 = -26.13 + 1.73)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:02:18 2006