
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 12,073,554 – 12,073,766 |
| Length | 212 |
| Max. P | 0.987044 |

| Location | 12,073,554 – 12,073,652 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 79.21 |
| Mean single sequence MFE | -19.32 |
| Consensus MFE | -19.45 |
| Energy contribution | -19.37 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.24 |
| Mean z-score | -3.00 |
| Structure conservation index | 1.01 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.987044 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
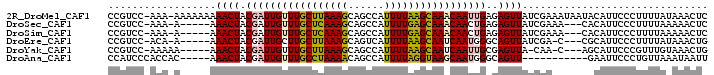
>2R_DroMel_CAF1 12073554 98 - 20766785 CCGUCC-AAA-AAAAAAAAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAACAAUUGAGAGUUAUCGAAAUAAUACAUUCCCUUUUAUAAACUC ......-...-.......((((.(((((((((((((((((......)))))))))))))))))..))))............................... ( -21.00) >DroSec_CAF1 69096 90 - 1 CCGUCC-AAA-A-----AAACUACGAUUGUUUGCUCAAAGCAGCCAUUUUGAGCAAACAACUGAGAGUUAUCGAAA---CACAUUCCCUUUUAAAAACUC ......-...-.-----......((.((((((((((((((......)))))))))))))).)).(((((.(..(((---.........)))..).))))) ( -19.70) >DroSim_CAF1 69966 90 - 1 CCGUCC-AAA-A-----AAACUACGAUUGUUUGCUCAAAGCAGCCAUUUUGAGCAAACAACUGAGAGUUAUCGAAA---CACAUUCCCUUUUAAAAACUC ......-...-.-----......((.((((((((((((((......)))))))))))))).)).(((((.(..(((---.........)))..).))))) ( -19.70) >DroEre_CAF1 69292 89 - 1 CCGUCC-ACA-A-----AAACUACGAUUGCUUGCUUAAAGCAGUCAUUUUAAGCAAUCAAUGGGCAGUUAUCGA-C---CGCAUUCCCUUUUAUAAACUG ..(((.-...-.-----.((((.(.((((.((((((((((......)))))))))).)))).)..))))...))-)---..................... ( -16.70) >DroYak_CAF1 70980 89 - 1 CCGUCC-AAAAA-----AAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAUCAAUUGCGAGUUA-CAA-C---AGCAUUCCCGUUUGUAAACUG ......-.....-----......((((((.((((((((((......)))))))))).))))))..(((((-(((-.---.((......)))))).)))). ( -17.80) >DroAna_CAF1 64859 84 - 1 CCAUCCCACCAC-----AAACUACGAUUGUUUGCCUAAAACAGCCAUUUUAGGUAAGCAAUGGGCAGUU-----------GAAUUCCCUGUUAAAUAAUU ............-----.((((.(.(((((((((((((((......))))))))))))))).)..))))-----------.................... ( -21.00) >consensus CCGUCC_AAA_A_____AAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAACAAUUGAGAGUUAUCGA_A___CACAUUCCCUUUUAAAAACUC ..................((((.(((((((((((((((((......)))))))))))))))))..))))............................... (-19.45 = -19.37 + -0.08)
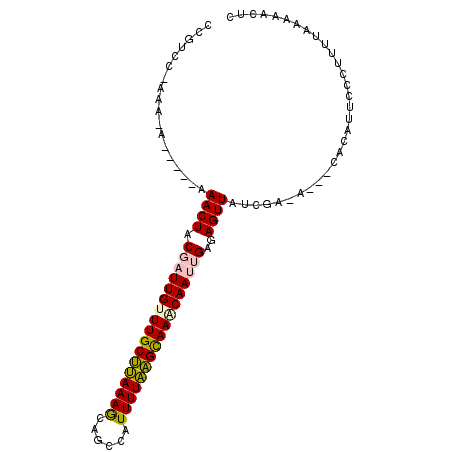
| Location | 12,073,585 – 12,073,692 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 86.36 |
| Mean single sequence MFE | -22.53 |
| Consensus MFE | -20.94 |
| Energy contribution | -20.78 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.967010 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12073585 107 - 20766785 AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUAACCCCGUCC-AAA-AAAAAAAAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAACAAUUGAGAGUU .....(((....((((....))))....)))...............-...-.......((((.(((((((((((((((((......)))))))))))))))))..)))) ( -24.00) >DroSec_CAF1 69124 102 - 1 AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAGUUAACCCCGUCC-AAA-A-----AAACUACGAUUGUUUGCUCAAAGCAGCCAUUUUGAGCAAACAACUGAGAGUU .....(((....((((....))))....)))...............-...-.-----.((((.((.((((((((((((((......)))))))))))))).))..)))) ( -22.20) >DroSim_CAF1 69994 102 - 1 AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAGUUAACCCCGUCC-AAA-A-----AAACUACGAUUGUUUGCUCAAAGCAGCCAUUUUGAGCAAACAACUGAGAGUU .....(((....((((....))))....)))...............-...-.-----.((((.((.((((((((((((((......)))))))))))))).))..)))) ( -22.20) >DroEre_CAF1 69319 102 - 1 AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUUACCCCGUCC-ACA-A-----AAACUACGAUUGCUUGCUUAAAGCAGUCAUUUUAAGCAAUCAAUGGGCAGUU ...........(((...(((((......(((...........))).-...-.-----))))).(.((((.((((((((((......)))))))))).)))).))))... ( -20.10) >DroYak_CAF1 71006 103 - 1 AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUUACCCCGUCC-AAAAA-----AAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAUCAAUUGCGAGUU .....(((....((((....))))....)))...............-.....-----.((((.((((((.((((((((((......)))))))))).))))))..)))) ( -19.90) >DroAna_CAF1 64879 102 - 1 AUUCUGCCAUAUGCAAAGGUUUAUUUUAAAAUUAU-AA-CCCAUCCCACCAC-----AAACUACGAUUGUUUGCCUAAAACAGCCAUUUUAGGUAAGCAAUGGGCAGUU ...(((((.........((.((((.........))-))-.))..........-----........(((((((((((((((......))))))))))))))).))))).. ( -26.80) >consensus AUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUAACCCCGUCC_AAA_A_____AAACUACGAUUGUUUGCUUAAAGCAGCCAUUUUAAGCAAACAAUUGAGAGUU ..(((((.....)))))((((((...))))))..........................((((.(((((((((((((((((......)))))))))))))))))..)))) (-20.94 = -20.78 + -0.16)

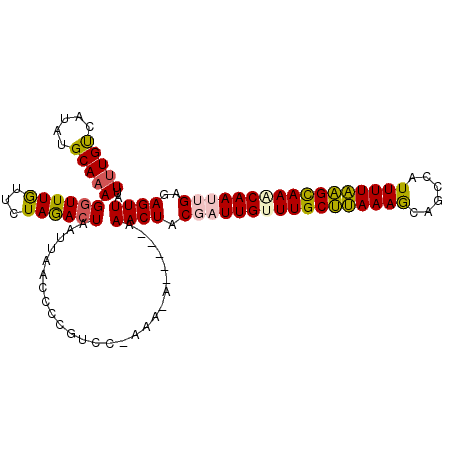
| Location | 12,073,652 – 12,073,766 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.69 |
| Mean single sequence MFE | -33.10 |
| Consensus MFE | -23.84 |
| Energy contribution | -23.65 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.74 |
| SVM RNA-class probability | 0.974951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12073652 114 + 20766785 GGUUAAUUAGUCUAGAACAAACCUUUGCAUAUGACAAAAUCCCCUCAUGGCUGUUUGAGCGGAUUUUGUAGUUCAACUCUGGCAG------CUUUUGCUUCAGUUCCAUGAGAUAUCCUA ((.((.........((((.........(....)(((((((((.((((........)))).))))))))).))))..((((((.((------((........))))))).))).)).)).. ( -27.10) >DroPse_CAF1 123283 103 + 1 GG---ACUACUGGAAAUCAAUC--------AACACAAAAUCCCUUCAUGGCUGUUUGAAGGGAUUUUGUGGUUCCACUGUGGCAG------CGUAUGCUUCAGUUCGAUGAGAGAUCCUC ..---......(((..((.(((--------..(((((((((((((((........))))))))))))))).....((((.((((.------....)))).))))..)))..))..))).. ( -39.20) >DroEre_CAF1 69381 114 + 1 GGUAAAUUAGUCUAGAACAAACCUUUGCAUAUGACAAAAUCCCCCCAUGGCUGUUUGAGGGGAUUUUGUAGUUCAACUCUGGCAG------CUUUUGCUUCAGUUCCAUGAGAUAUCCUA ((((..........((((.........(....)(((((((((((.((........)).))))))))))).))))..((((((.((------((........))))))).))).))))... ( -29.60) >DroYak_CAF1 71069 114 + 1 GGUAAAUUAGUCUAGAACAAACCUUUGCAUAUGACAAAAUCCCAACAUGGCUGUUUGUGGGGAUUUUGUAGUUCAACUCUGGCAG------CUUUUGCUUCAGUUCCAUGAGUUAUCCUA ((............((((.........(....)((((((((((.(((........))).)))))))))).))))((((((((.((------((........))))))).)))))..)).. ( -29.30) >DroAna_CAF1 64943 118 + 1 G-UU-AUAAUUUUAAAAUAAACCUUUGCAUAUGGCAGAAUCGUCUCAUGGCUGUUUGAGGCGAUUCUGUUGAUCAACUGUGGCAGCAUUUGCUUUUGCUUCAGUUCCAUAAGAUAUCCUA .-..-........................((((((((((((((((((........)))))))))))))......(((((.((((((....))...)))).)))))))))).......... ( -34.20) >DroPer_CAF1 120946 103 + 1 GG---ACUACUGGAAAUCAAUC--------AACACAAAAUCCCUUCAUGGCUGUUUGAAGGGAUUUUGUGGUUCCACUGUGGCAG------CGUAUGCUUCAGUUCGAUGAGAGAUCCUC ..---......(((..((.(((--------..(((((((((((((((........))))))))))))))).....((((.((((.------....)))).))))..)))..))..))).. ( -39.20) >consensus GGU__AUUAGUCUAAAACAAACCUUUGCAUAUGACAAAAUCCCCUCAUGGCUGUUUGAGGGGAUUUUGUAGUUCAACUCUGGCAG______CUUUUGCUUCAGUUCCAUGAGAUAUCCUA .................................((((((((((((((........)))))))))))))).....((((..(((((.........)))))..))))............... (-23.84 = -23.65 + -0.19)


| Location | 12,073,652 – 12,073,766 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.69 |
| Mean single sequence MFE | -32.37 |
| Consensus MFE | -22.16 |
| Energy contribution | -21.72 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.29 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.92 |
| SVM RNA-class probability | 0.982612 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 12073652 114 - 20766785 UAGGAUAUCUCAUGGAACUGAAGCAAAAG------CUGCCAGAGUUGAACUACAAAAUCCGCUCAAACAGCCAUGAGGGGAUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUAACC (((((((..((......(((.(((....)------))..))).((......(((((((((.((((........)))).))))))))).....))...))..)))))))............ ( -29.50) >DroPse_CAF1 123283 103 - 1 GAGGAUCUCUCAUCGAACUGAAGCAUACG------CUGCCACAGUGGAACCACAAAAUCCCUUCAAACAGCCAUGAAGGGAUUUUGUGUU--------GAUUGAUUUCCAGUAGU---CC ..(((..((..(((((((((..(((....------.)))..)))).....(((((((((((((((........)))))))))))))))))--------))).))..)))......---.. ( -36.30) >DroEre_CAF1 69381 114 - 1 UAGGAUAUCUCAUGGAACUGAAGCAAAAG------CUGCCAGAGUUGAACUACAAAAUCCCCUCAAACAGCCAUGGGGGGAUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUUACC (((((((..((......(((.(((....)------))..))).((......((((((((((((((........)))))))))))))).....))...))..)))))))............ ( -33.50) >DroYak_CAF1 71069 114 - 1 UAGGAUAACUCAUGGAACUGAAGCAAAAG------CUGCCAGAGUUGAACUACAAAAUCCCCACAAACAGCCAUGUUGGGAUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAUUUACC (((((((((((.(((......(((....)------)).))))))))(((((((((((((((.(((........))).))))))))))..........)))))))))))............ ( -29.60) >DroAna_CAF1 64943 118 - 1 UAGGAUAUCUUAUGGAACUGAAGCAAAAGCAAAUGCUGCCACAGUUGAUCAACAGAAUCGCCUCAAACAGCCAUGAGACGAUUCUGCCAUAUGCAAAGGUUUAUUUUAAAAUUAU-AA-C ((((((((((((((((((((..(((...((....)))))..)))))......((((((((.((((........)))).))))))))))))).....))))..)))))).......-..-. ( -29.00) >DroPer_CAF1 120946 103 - 1 GAGGAUCUCUCAUCGAACUGAAGCAUACG------CUGCCACAGUGGAACCACAAAAUCCCUUCAAACAGCCAUGAAGGGAUUUUGUGUU--------GAUUGAUUUCCAGUAGU---CC ..(((..((..(((((((((..(((....------.)))..)))).....(((((((((((((((........)))))))))))))))))--------))).))..)))......---.. ( -36.30) >consensus UAGGAUAUCUCAUGGAACUGAAGCAAAAG______CUGCCACAGUUGAACUACAAAAUCCCCUCAAACAGCCAUGAGGGGAUUUUGUCAUAUGCAAAGGUUUGUUCUAGACUAAU__ACC (((((((........((((...(((...........)))...)))).....((((((((((((((........))))))))))))))..............)))))))............ (-22.16 = -21.72 + -0.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:00:33 2006