
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,824,584 – 11,824,697 |
| Length | 113 |
| Max. P | 0.882727 |

| Location | 11,824,584 – 11,824,678 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 79.86 |
| Mean single sequence MFE | -39.35 |
| Consensus MFE | -20.76 |
| Energy contribution | -23.65 |
| Covariance contribution | 2.89 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.572444 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
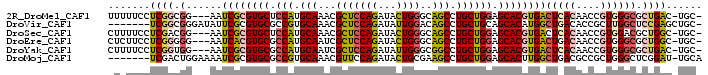
>2R_DroMel_CAF1 11824584 94 + 20766785 -GCA-GUCAGCGCCCACGGUUGUGAGUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGAGCACGCGAUU---CCGCCGAGGAAAAA -((.-....))..((.((((.(.(((((.(((((((((((((((.((((((...)))).)))))))).))))))))).))))---)))))).))..... ( -48.50) >DroVir_CAF1 31718 91 + 1 -GCAGCUCGGAGCCAAGCGGUGUCAGCCAUGUGCUGCAGCAGGCUGUCCAAUAUCUGGAGCGUUUGCACGGCGCACGCGAAUAUCCCGCCGA------- -((.(((.(....).)))(((((..((..(((((((..((((((..((((.....))))..)))))).))))))).))..)))))..))...------- ( -32.00) >DroSec_CAF1 15136 94 + 1 -GCA-GCCAGCGUCCACGGUUGUGAGUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGAGCACGCGAUU---CCGUCGAGGAAAAG -(((-(((.........))))))(((((.(((((((((((((((.((((((...)))).)))))))).))))))))).))))---)............. ( -45.00) >DroEre_CAF1 14970 94 + 1 -GCA-GCCAGCGCCCACGGUUGUCAGUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGAUUGCAUGGCGCACGUGAUU---CCCCCGAGGAAGAG -((.-....))..((.(((..(..((((((((((.(((((((..(((((((...)))).))).)))).))).))))))))))---..)))).))..... ( -39.90) >DroYak_CAF1 15280 94 + 1 -GCA-GUCAGCGCCCACGGUUGUGAGUCACGUGCUCCAGCAGGCCGCCCAAUAUCUGGAGCGAUUGCAUGGCGCACGCGAUU---CCACCGAGGAAAAG -((.-....))..((.((((.(.(((((.(((((.(((((((..((((((.....))).))).)))).))).))))).))))---)))))).))..... ( -42.30) >DroMoj_CAF1 38849 91 + 1 UGCA-AUCCGAGCCCAGCGGCGUCAGCCAAGUGCUCCAGCAGGCUUCGCAGUAUCUGGAACGUUUGCACGGCGCACGCGAUUUUCCAGUCGA------- ....-...........(((((....)))..((((.((.(((((((((.(((...)))))).))))))..)).))))))(((......)))..------- ( -28.40) >consensus _GCA_GUCAGCGCCCACGGUUGUCAGUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGCGCACGCGAUU___CCGCCGAGGAAAAG .............((.(((.....((((.(((((.(((((((((.(((((.....))).)))))))).))).))))).))))......))).))..... (-20.76 = -23.65 + 2.89)



| Location | 11,824,584 – 11,824,678 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 79.86 |
| Mean single sequence MFE | -37.18 |
| Consensus MFE | -23.90 |
| Energy contribution | -24.48 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.571920 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11824584 94 - 20766785 UUUUUCCUCGGCGG---AAUCGCGUGCUCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGACUCACAACCGUGGGCGCUGAC-UGC- .......((((((.---..((((((((((((.(((..(((.((((...)))))).)..)))))))))))))))(((((....))))))))))).-...- ( -45.40) >DroVir_CAF1 31718 91 - 1 -------UCGGCGGGAUAUUCGCGUGCGCCGUGCAAACGCUCCAGAUAUUGGACAGCCUGCUGCAGCACAUGGCUGACACCGCUUGGCUCCGAGCUGC- -------.(((((((......((((..((...))..))))(((((...)))))...)))))))((((.....)))).....(((((....)))))...- ( -30.80) >DroSec_CAF1 15136 94 - 1 CUUUUCCUCGACGG---AAUCGCGUGCUCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGACUCACAACCGUGGACGCUGGC-UGC- ....(((.((...(---(.((((((((((((.(((..(((.((((...)))))).)..))))))))))))))).)).....)).))).((....-.))- ( -38.90) >DroEre_CAF1 14970 94 - 1 CUCUUCCUCGGGGG---AAUCACGUGCGCCAUGCAAUCGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGACUGACAACCGUGGGCGCUGGC-UGC- .....((.(((..(---..((((((((.(((.(((..(((.((((...)))))).)..)))))).))))))))....)..))).))((....))-...- ( -36.80) >DroYak_CAF1 15280 94 - 1 CUUUUCCUCGGUGG---AAUCGCGUGCGCCAUGCAAUCGCUCCAGAUAUUGGGCGGCCUGCUGGAGCACGUGACUCACAACCGUGGGCGCUGAC-UGC- ....(((.((((.(---(.((((((((.(((.(((.((((.((((...))))))))..)))))).)))))))).))...)))).))).((....-.))- ( -41.90) >DroMoj_CAF1 38849 91 - 1 -------UCGACUGGAAAAUCGCGUGCGCCGUGCAAACGUUCCAGAUACUGCGAAGCCUGCUGGAGCACUUGGCUGACGCCGCUGGGCUCGGAU-UGCA -------..........((((.((.((.(((.((....(((((((...((....))....)))))))....(((....)))))))))).)))))-)... ( -29.30) >consensus CUUUUCCUCGGCGG___AAUCGCGUGCGCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGACUCACAACCGUGGGCGCUGAC_UGC_ .......((((.(......((((((((.(((.(((...(((((((...))))..))).)))))).))))))))(((((....)))))).))))...... (-23.90 = -24.48 + 0.59)


| Location | 11,824,607 – 11,824,697 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 83.37 |
| Mean single sequence MFE | -41.97 |
| Consensus MFE | -25.85 |
| Energy contribution | -26.55 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.585353 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11824607 90 + 20766785 GUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGAGCACGCG---AUUCCGCCGAGGAAAAAACGGA---GGAAGUGCAG---CCAG------ .....((((((((((((((.((((((...)))).)))))))).))))))))(((---.((((.(((.........))).---)))).)))..---....------ ( -42.50) >DroSec_CAF1 15159 96 + 1 GUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGAGCACGCG---AUUCCGUCGAGGAAAAGACGGA---GGAAGUGCAG---CCAGAUGGUG .....((((((((((((((.((((((...)))).)))))))).))))))))(((---.(((((((........))))))---)....))).(---((....))). ( -47.00) >DroSim_CAF1 14014 96 + 1 GUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGUUUGCAUGGAGCACGCG---AUUCCGUCGAGGAAAAGACGGA---GGAAGUGCAG---CCAGAUGGUG .....((((((((((((((.((((((...)))).)))))))).))))))))(((---.(((((((........))))))---)....))).(---((....))). ( -47.00) >DroEre_CAF1 14993 96 + 1 GUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGAUUGCAUGGCGCACGUG---AUUCCCCCGAGGAAGAGGCGGA---GGAAGUGCAG---CCAGAUGGCG .((((((((.(((((((..(((((((...)))).))).)))).))).)))))))---)((((.(((.........))).---)))).....(---((....))). ( -43.20) >DroYak_CAF1 15303 99 + 1 GUCACGUGCUCCAGCAGGCCGCCCAAUAUCUGGAGCGAUUGCAUGGCGCACGCG---AUUCCACCGAGGAAAAGGCGGAGGAGGAAGUGCAG---CCAGAUGGCC (.(((((((.(((((((..((((((.....))).))).)))).))).)))).(.---.((((.((........)).))))..)...)))).(---((....))). ( -38.80) >DroAna_CAF1 13840 93 + 1 GUCACGUGCUCCAGCAGGCGGCCCAGUAUCUGGAGCGAUUGCAUGGCUCACGCGACGAUCCCGUCGAGC---------C---GGAAGUGCCCGUAUCCGAGGCCG ((....((((((((...((......))..))))))))...)).((((((....((((....))))))))---------)---)...(.(((((....)).)))). ( -33.30) >consensus GUCACGUGCUCCAGCAGGCUGCCCAGUAUCUGGAGCGAUUGCAUGGAGCACGCG___AUUCCGCCGAGGAAAAGACGGA___GGAAGUGCAG___CCAGAUGGCG .....((((.((((((..(.((((((...)))).)))..))).))).))))(((....((((.(((.........)))....)))).)))............... (-25.85 = -26.55 + 0.70)


| Location | 11,824,607 – 11,824,697 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 83.37 |
| Mean single sequence MFE | -41.68 |
| Consensus MFE | -29.42 |
| Energy contribution | -29.95 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.882727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
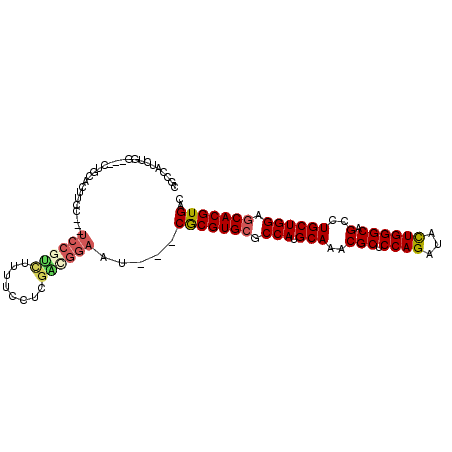
>2R_DroMel_CAF1 11824607 90 - 20766785 ------CUGG---CUGCACUUCC---UCCGUUUUUUCCUCGGCGGAAU---CGCGUGCUCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGAC ------....---......((((---.(((.........))).))))(---(((((((((((.(((..(((.((((...)))))).)..))))))))))))))). ( -39.60) >DroSec_CAF1 15159 96 - 1 CACCAUCUGG---CUGCACUUCC---UCCGUCUUUUCCUCGACGGAAU---CGCGUGCUCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGAC ..((....))---..........---((((((........)))))).(---(((((((((((.(((..(((.((((...)))))).)..))))))))))))))). ( -41.30) >DroSim_CAF1 14014 96 - 1 CACCAUCUGG---CUGCACUUCC---UCCGUCUUUUCCUCGACGGAAU---CGCGUGCUCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGAC ..((....))---..........---((((((........)))))).(---(((((((((((.(((..(((.((((...)))))).)..))))))))))))))). ( -41.30) >DroEre_CAF1 14993 96 - 1 CGCCAUCUGG---CUGCACUUCC---UCCGCCUCUUCCUCGGGGGAAU---CACGUGCGCCAUGCAAUCGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGAC .(((....))---).....((((---((((.........))))))))(---(((((((.(((.(((..(((.((((...)))))).)..)))))).)))))))). ( -42.50) >DroYak_CAF1 15303 99 - 1 GGCCAUCUGG---CUGCACUUCCUCCUCCGCCUUUUCCUCGGUGGAAU---CGCGUGCGCCAUGCAAUCGCUCCAGAUAUUGGGCGGCCUGCUGGAGCACGUGAC ((((....))---))...........((((((........)))))).(---(((((((.(((.(((.((((.((((...))))))))..)))))).)))))))). ( -43.70) >DroAna_CAF1 13840 93 - 1 CGGCCUCGGAUACGGGCACUUCC---G---------GCUCGACGGGAUCGUCGCGUGAGCCAUGCAAUCGCUCCAGAUACUGGGCCGCCUGCUGGAGCACGUGAC ..(((.((....)))))..((((---(---------......)))))..((((((((..(((.(((..((..((((...))))..))..))))))..)))))))) ( -41.70) >consensus CGCCAUCUGG___CUGCACUUCC___UCCGUCUUUUCCUCGACGGAAU___CGCGUGCGCCAUGCAAACGCUCCAGAUACUGGGCAGCCUGCUGGAGCACGUGAC ..........................((((((........)))))).....(((((((.(((.(((..(((.((((...)))))).)..)))))).))))))).. (-29.42 = -29.95 + 0.53)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:57 2006