
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
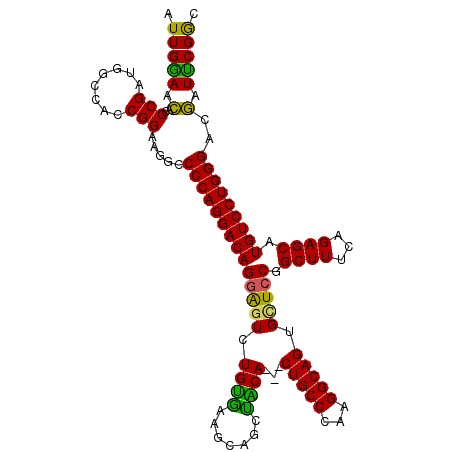
| Location | 11,791,500 – 11,791,610 |
| Length | 110 |
| Max. P | 0.794289 |

| Location | 11,791,500 – 11,791,610 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 87.69 |
| Mean single sequence MFE | -54.55 |
| Consensus MFE | -37.90 |
| Energy contribution | -36.35 |
| Covariance contribution | -1.55 |
| Combinations/Pair | 1.28 |
| Mean z-score | -3.95 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.794289 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
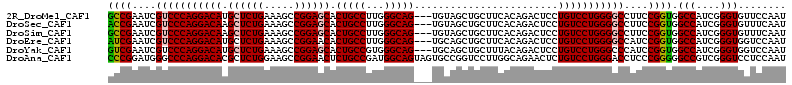
>2R_DroMel_CAF1 11791500 110 + 20766785 AUUGGAACACCCGAUGGCCACCGGAAGGCCCCAGGACAGGAGUCUGUGAAGCAGCUACA---CUGCCCAAGGCAGUGCUCCGGCUUUCAGAGCAUGUCCUGGGACGAUUCGGC (((((.....))))).(((.((....)).(((((((((.(..((((.(((((.....((---(((((...))))))).....))))))))).).))))))))).......))) ( -50.70) >DroSec_CAF1 11855 110 + 1 AUUGAAACACCCGAUGGCCACCGGAAGGCCCCAGGACAGGAGUCUGUGAAGCAGCUACA---CUGCCCAAGGCAGUGCUCCGGCUUUCAGAGCUUGUCCUGGGACGAUUCGGU ..........((((......((....)).(((((((((((..((((.(((((.....((---(((((...))))))).....))))))))).))))))))))).....)))). ( -51.90) >DroSim_CAF1 12886 110 + 1 AUUGAAACACCCGAUGGCCACCGGAAGGCCCCAGGACAGGAGUCUGUGAAGCAGCUACA---CUGCCCAAGGCAGUGCUCCGGCUUUCAGAGCUUGUCCUGGGACGAUUCGGC ..........((((......((....)).(((((((((((..((((.(((((.....((---(((((...))))))).....))))))))).))))))))))).....)))). ( -51.70) >DroEre_CAF1 11932 110 + 1 AUUGGACCACCCGAUGGCCACCGGAUGGCCCCAGGACAGGAGUCUGUGAAGCAGCUGCA---CUGCCCAAGGCAGUGUUCCGGCUUUCAGAGCAUGUCCUGGGACGAUUCGAU ..(((.(((.....)))))).(((((.(.(((((((((.(..((((.(((((....(((---(((((...))))))))....))))))))).).))))))))).).))))).. ( -54.30) >DroYak_CAF1 11964 110 + 1 AUUGGACCACCCGAUGGCCACCGGAUGGGCCCAGGACAGGAGUCUGUAAAGCAGCUGCA---CUGCCCACGGCAGUGCUCCGGCUUUCAGAGCAUGUCCUGGGACGAUUCGAC ..(((.(((.....)))))).(((((.(.(((((((((.(..((((.(((((....(((---(((((...))))))))....))))))))).).))))))))).).))))).. ( -56.40) >DroAna_CAF1 14163 113 + 1 AUUGGAGGACCCGACGGCCCCCGGGAGGUCCCAGGACAGAGUUCUGCCAAGGACCGGCACUACUGCCAUCGGCAGAGUUCCGGCUUCCAGAGCGUGUCCUGGGCCCAUCCGGG ......((.((....))))(((((..((.(((((((((..((((((...(((.((((.(((.(((((...)))))))).))))))).)))))).))))))))).))..))))) ( -62.30) >consensus AUUGGAACACCCGAUGGCCACCGGAAGGCCCCAGGACAGGAGUCUGUGAAGCAGCUACA___CUGCCCAAGGCAGUGCUCCGGCUUUCAGAGCAUGUCCUGGGACGAUUCGGC .(((((.(..(((........))).....((((((((((((((.((((.......))))...(((((...))))).))))).((((...)))).)))))))))..).))))). (-37.90 = -36.35 + -1.55)

| Location | 11,791,500 – 11,791,610 |
|---|---|
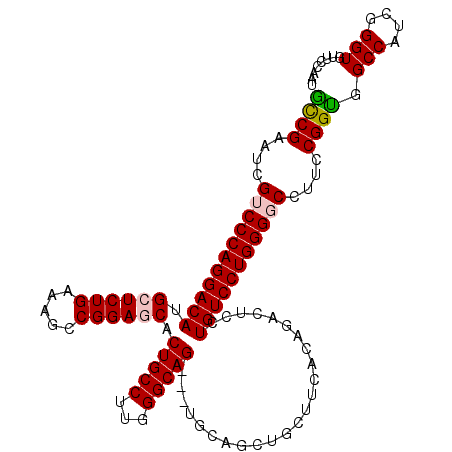
| Length | 110 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 87.69 |
| Mean single sequence MFE | -52.65 |
| Consensus MFE | -39.37 |
| Energy contribution | -39.37 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.752859 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11791500 110 - 20766785 GCCGAAUCGUCCCAGGACAUGCUCUGAAAGCCGGAGCACUGCCUUGGGCAG---UGUAGCUGCUUCACAGACUCCUGUCCUGGGGCCUUCCGGUGGCCAUCGGGUGUUCCAAU ((((.(..(((((((((((.(.((((.((((.(..((((((((...)))))---)))..).))))..))))..).)))))))))))..).)))).(((....)))........ ( -49.00) >DroSec_CAF1 11855 110 - 1 ACCGAAUCGUCCCAGGACAAGCUCUGAAAGCCGGAGCACUGCCUUGGGCAG---UGUAGCUGCUUCACAGACUCCUGUCCUGGGGCCUUCCGGUGGCCAUCGGGUGUUUCAAU ...(((.((((((((((((((.((((.((((.(..((((((((...)))))---)))..).))))..))))))..)))))))))))..((((((....)))))).).)))... ( -49.90) >DroSim_CAF1 12886 110 - 1 GCCGAAUCGUCCCAGGACAAGCUCUGAAAGCCGGAGCACUGCCUUGGGCAG---UGUAGCUGCUUCACAGACUCCUGUCCUGGGGCCUUCCGGUGGCCAUCGGGUGUUUCAAU ...(((.((((((((((((((.((((.((((.(..((((((((...)))))---)))..).))))..))))))..)))))))))))..((((((....)))))).).)))... ( -49.90) >DroEre_CAF1 11932 110 - 1 AUCGAAUCGUCCCAGGACAUGCUCUGAAAGCCGGAACACUGCCUUGGGCAG---UGCAGCUGCUUCACAGACUCCUGUCCUGGGGCCAUCCGGUGGCCAUCGGGUGGUCCAAU ............(((((((.(.((((.((((.((..(((((((...)))))---))...))))))..))))..).)))))))((((((((((((....))))))))))))... ( -51.30) >DroYak_CAF1 11964 110 - 1 GUCGAAUCGUCCCAGGACAUGCUCUGAAAGCCGGAGCACUGCCGUGGGCAG---UGCAGCUGCUUUACAGACUCCUGUCCUGGGCCCAUCCGGUGGCCAUCGGGUGGUCCAAU ........(.(((((((((.(.(((((((((.(..((((((((...)))))---)))..).))))).))))..).))))))))))(((((((((....)))))))))...... ( -54.80) >DroAna_CAF1 14163 113 - 1 CCCGGAUGGGCCCAGGACACGCUCUGGAAGCCGGAACUCUGCCGAUGGCAGUAGUGCCGGUCCUUGGCAGAACUCUGUCCUGGGACCUCCCGGGGGCCGUCGGGUCCUCCAAU .((((..((.((((((((..(((..((..(((((.((((((((...))))).))).)))))))..)))((....)))))))))).))..))))(((((....)))))...... ( -61.00) >consensus GCCGAAUCGUCCCAGGACAUGCUCUGAAAGCCGGAGCACUGCCUUGGGCAG___UGCAGCUGCUUCACAGACUCCUGUCCUGGGGCCUUCCGGUGGCCAUCGGGUGUUCCAAU ((((....(((((((((((.((((((.....)))))).(((((...)))))........................)))))))))))....)))).(((....)))........ (-39.37 = -39.37 + 0.00)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:30 2006