| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
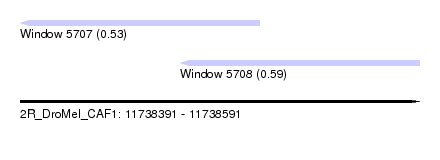
| Location | 11,738,391 – 11,738,591 |
| Length | 200 |
| Max. P | 0.590753 |

| Location | 11,738,391 – 11,738,511 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.00 |
| Mean single sequence MFE | -45.77 |
| Consensus MFE | -33.11 |
| Energy contribution | -31.92 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.72 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.530387 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11738391 120 - 20766785 UUUCCAGGGCAUUACCUAUGCUCUGUGCUAUACGUGCAUUGUGGCCUACGCCUCGGCGGUGGCUCCUCCGGGAACAUCGGCCACCGUUCAAGGAUUGAUGGCCGGCAUGGACGAUGGACU ....((((((((.....))))))))..((((.(((.(((..(((((....(((.((((((((((((....)))......)))))))))..)))......)))))..))).)))))))... ( -48.80) >DroVir_CAF1 6702 120 - 1 CUUCCAGGGCGUUACCUAUGCCCUCUGCUACACUUGCAUUGUUGCCUAUGCUUCGGCAGUGGCACCACCCGGCACCUCAGCCACUGUACAGGGUCUCAUGGCGGGCAUGGACGAUGGGUU ..((((...(((...((((((((((((.((((.......((.((((..(((....)))..)))).))...(((......)))..))))))))(((....)))))))))))))).)))).. ( -45.50) >DroGri_CAF1 12705 120 - 1 CUUUCAGGGCAUCACAUAUGCCCUUUGCUACACCUGCAUUGUGGCGUACGCAUCGGCAGUUGCUCCACCCGGCACCUCUGCCACCGUUCAGGGACUCAUGGCUGGCAUGGAUGAUGGAUU .....(((((((.....)))))))..((((((.......)))))).....(((((((((.((((......))))...))))).((((((((..........)))).))))..)))).... ( -38.90) >DroWil_CAF1 25393 120 - 1 UUUCCAGGGCAUAACCUAUGCUCUCUGUUAUACCUGCAUCGUGGCCUAUGCCUCGGCCGUGGCUCCACCCGGAACUUCGGCCACUGUGCAGGGUCUAAUGGCUGGAAUGGAUGAUGGCUU .((((((((((((...))))))..((((((..(((((((.((((((...(((.(....).)))(((....))).....)))))).)))))))...))))))))))))............. ( -46.90) >DroMoj_CAF1 14732 120 - 1 CUUCCAGGGCGUAACCUACGCCCUCUGCUACACUUGCAUUGUGGCCUAUGCCUCGGCCGUGGCCCCGCCCGGCACCUCAGCCACGGUCCAAGGCCUCAUGGCUGGCAUGGACGAUGGGCU ..(((((((((((...)))))))...((((((.......))))))(((((((..(((((((((.((....)).......)))))))))...((((....)))))))))))....)))).. ( -52.60) >DroAna_CAF1 5355 120 - 1 CUUCCAGGGCAUCACCUAUGCCCUUUGCUACACUUGCAUUGUGGCCUACGCCUCGGCAGUGGCUCCUCCUGGCACUUCGGCUACUGUUCAAGGACUGAUGGCUGGGAUGGACGAUGGUCU ...(((((((((.....))))))...(((((...(((...(.(((....))).).))))))))((((((..((...((((...((.....))..))))..))..))).)))...)))... ( -41.90) >consensus CUUCCAGGGCAUAACCUAUGCCCUCUGCUACACUUGCAUUGUGGCCUACGCCUCGGCAGUGGCUCCACCCGGCACCUCGGCCACUGUUCAAGGACUCAUGGCUGGCAUGGACGAUGGACU ..((((((((((.....))))))...((((..((((.((.((((((...(((.(....).))).((....))......)))))).)).))))......)))).....))))......... (-33.11 = -31.92 + -1.19)
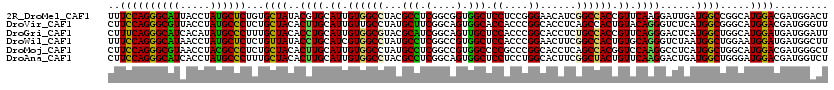
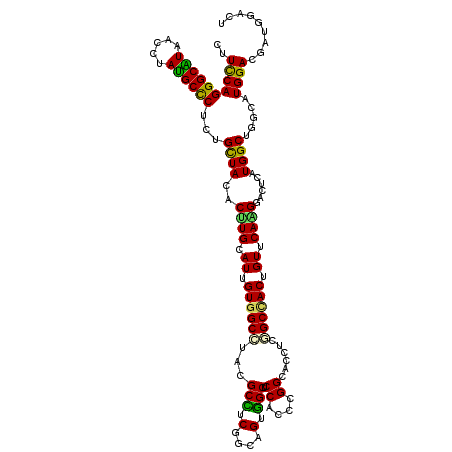
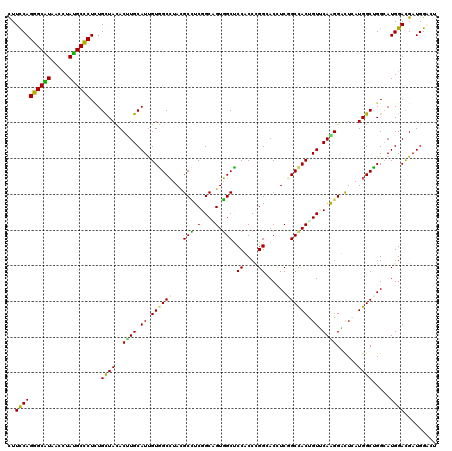
| Location | 11,738,471 – 11,738,591 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.33 |
| Mean single sequence MFE | -37.00 |
| Consensus MFE | -23.05 |
| Energy contribution | -22.00 |
| Covariance contribution | -1.05 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.590753 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11738471 120 - 20766785 UGCUUUUUCUUCUACGCUGUGCGACUGUCGCUGAUCGCCUGGAUUCCCAAUCCCUGGUAUCUGGUGGGCGUGGAGCUCUUUUUCCAGGGCAUUACCUAUGCUCUGUGCUAUACGUGCAUU (((......(((((((((..(((.....)))....((((.((((..(((.....))).))))))))))))))))).........((((((((.....))))))))..........))).. ( -38.10) >DroVir_CAF1 6782 120 - 1 UGCUUCUUCUUUUAUGCGGUACGCUUAUCGCUCAUAUCCUGGAUACCAAAUCCCUGGUAUCUAGUGGGCGUGGAGCUGUUCUUCCAGGGCGUUACCUAUGCCCUCUGCUACACUUGCAUU .............((((((.((((((..((((((....((((((((((......))))))))))))))))..)))).))....))(((((((.....)))))))...........)))). ( -42.00) >DroGri_CAF1 12785 120 - 1 UGCUUCUUCUUUUAUGCUGUGCGCCUGUCACUCAUUUCAUGGAUCCCGAAUCCCUGGUAUCUGGUUGGCGUGGAGCUCUUCUUUCAGGGCAUCACAUAUGCCCUUUGCUACACCUGCAUU (((............(((.((((((..(((......(((.((((.....)))).)))....)))..)))))).))).........(((((((.....)))))))...........))).. ( -32.90) >DroWil_CAF1 25473 120 - 1 UGUUUCUUUUUCUAUGCCGUCCGUUUGUCACUCAUCGCUUGGAUACCAAAUCCUUGGUAUUUGGUGGGUGUUGAGCUCUUUUUCCAGGGCAUAACCUAUGCUCUCUGUUAUACCUGCAUC .............((((.....(((((.(((((((((....((((((((....))))))))))))))))).))))).........((((((((...))))))))...........)))). ( -31.20) >DroMoj_CAF1 14812 120 - 1 UGCUUCUUCUUCUAUGCAGUGCGGCUGUCGCUCAUCUCCUGGAUACCGAAUCCCUGGUAUCUAGUAGGCGUGGAGCUGUUCUUCCAGGGCGUAACCUACGCCCUCUGCUACACUUGCAUU .............((((((((..((....((((.(((.((((((((((......)))))))))).)))....)))).........((((((((...))))))))..))..))).))))). ( -45.40) >DroAna_CAF1 5435 120 - 1 UGCUUCUUUUUCUAUGCGGUGAGACUGUCGUUGAUAGCUUGGAUUCCCAAUCCCUGGUACCUGGUGGGCGUGGAGCUCUUCUUCCAGGGCAUCACCUAUGCCCUUUGCUACACUUGCAUU .............((((((((((.(((((...))))))))((((.....)))).....))).(((((((((((((......)))))((((((.....))))))..)))).)))).)))). ( -32.40) >consensus UGCUUCUUCUUCUAUGCGGUGCGACUGUCGCUCAUCGCCUGGAUACCAAAUCCCUGGUAUCUGGUGGGCGUGGAGCUCUUCUUCCAGGGCAUAACCUAUGCCCUCUGCUACACUUGCAUU (((......((((((((.(((.......))).......((((((.(((......))).))))))...))))))))..........(((((((.....)))))))...........))).. (-23.05 = -22.00 + -1.05)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:06 2006