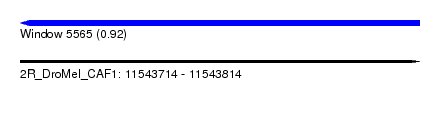
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,543,714 – 11,543,814 |
| Length | 100 |
| Max. P | 0.920766 |

| Location | 11,543,714 – 11,543,814 |
|---|---|
| Length | 100 |
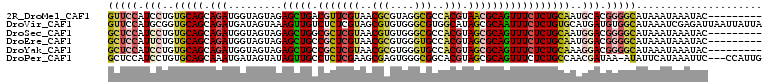
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 79.09 |
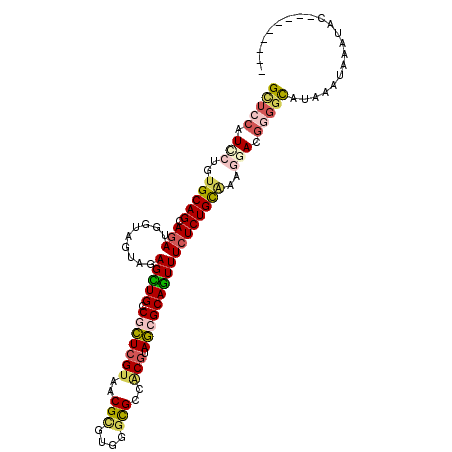
| Mean single sequence MFE | -36.28 |
| Consensus MFE | -23.16 |
| Energy contribution | -24.05 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.920766 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11543714 100 - 20766785 GUUCCAUCCUGUGCAGCAGAUGGUAGUAGAGCUGACGUUCGUAACGCGUAGGCGCCACGUAACGCAGUUUCUCUGCAAUGCACGGGGCAUAAAUAAAUAC--------- ......(((((((((.......((((..((((((.(((((((..(((....)))..))).))))))))))..))))..))))))))).............--------- ( -36.70) >DroVir_CAF1 4504 109 - 1 GUUCCAUGCGGUGCAGCAGAUGAUAGUAAAGUUGUCUCUCGUAGCGUGUGGGCGUGGCAUAGCGCAAUUUCUCUGUGCAUGAUGUGGCAUAAAUCGAGAUUAAUUAUUA ((.(((((((.(((((.(((..((......))..))))).))).)))))))))((.((((.(((((.......)))))...)))).))..................... ( -32.60) >DroSec_CAF1 3842 100 - 1 GCUCCAUCCUGUGCAGCAGAUGGUAGUAGAGCUGGCGCUCGUAACGUGUGGGCGCCACGUAGCGCAGUUUCUCUGCAAUGGACGGGGCAUAAAUAAAUAC--------- (((((.(((..(((((.(((..((.((...(.(((((((((.......))))))))))...))))....))))))))..))).)))))............--------- ( -39.20) >DroEre_CAF1 3857 100 - 1 GCUCCAUUCUGUGCAGCAGAUGGUAGUAGAGCUGCCGCUCGUAACGCGUGGGUGCCACGUAGCGCAGUUUCUCUGCAAUGGACGGGGCAUAAAUAAAUAC--------- (((((.(((..(((((.(((.(((((.....)))))((((((...((((((...)))))).))).))).))))))))..))).)))))............--------- ( -38.30) >DroYak_CAF1 3851 100 - 1 GCUCCAUCCUGUGCAGCAGAUGGUAGUAGAGCUGCCGCUCGUAACGCGUGGGUGCCACGUAGCGCAGUUUCUCUGCAAAGGACGGGGCAUAAAUAAAUAC--------- (((((.((((.(((((.(((.(((((.....)))))((((((...((((((...)))))).))).))).)))))))).)))).)))))............--------- ( -43.40) >DroPer_CAF1 3894 105 - 1 GCUCCAUCCUGUGCAGCAAAUGAUAGUAUAGUUGCCUCUCGAAGCGAGUGGGCGGCACGUAGCGCAGUUUCUCUGCCAACGAUAA-AUAUUCAUAAAUUC---CCAUUG (((.(((...))).)))..((((..((((.(((((((((((...)))).))))))).(((.(.((((.....))))).)))....-))))))))......---...... ( -27.50) >consensus GCUCCAUCCUGUGCAGCAGAUGGUAGUAGAGCUGCCGCUCGUAACGCGUGGGCGCCACGUAGCGCAGUUUCUCUGCAAAGGACGGGGCAUAAAUAAAUAC_________ (((((.(((..(((((.(((.........(((((.(((((((..(((....)))..))).)))))))))))))))))..))).)))))..................... (-23.16 = -24.05 + 0.89)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:54:47 2006