| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,439,768 – 11,439,883 |
| Length | 115 |
| Max. P | 0.918654 |

| Location | 11,439,768 – 11,439,883 |
|---|---|
| Length | 115 |
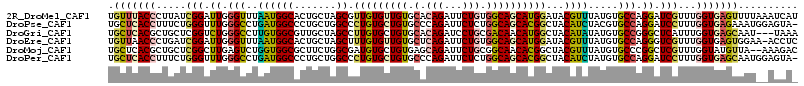
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 75.38 |
| Mean single sequence MFE | -42.52 |
| Consensus MFE | -21.87 |
| Energy contribution | -21.27 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918654 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11439768 115 - 20766785 UGUUUACCCUUAUCGGAUUGGGUUUAAUGGCACUGCUAGCGUUGUGUUGUGCACAGAUUCUGUGGCAGCAUGGAUACGUUUAUGUGCCAGGAUCGUUUGGUGAGUUUUAAAUCAU .(((((..((((((((((..((((...((((((....(((((.(((((((.(((((...))))))))))))....)))))...)))))).))))))))))))))...)))))... ( -37.60) >DroPse_CAF1 56850 114 - 1 UGCUCACCUUUCUGGGUUUGGGCCUGAUGGCCCUGCUGGCCCUGUGCUGUGCCCAGAUUCUCUGGCAGCACGGCUACAUCUACGUGCCAGGAUCCUUUGGUGAGAAAUGGAGUA- ..((((((.....(((((.(((((....)))))..(((((.(((((((((((((((.....))))..))))))).))).....).))))))))))...))))))..........- ( -51.10) >DroGri_CAF1 67234 112 - 1 UGCUCACGCUGCUCGGUCUGGGCCUUGUGGCGUUGCUAGCCUUGUGCUGUGCACAGAUCCUGCGACAACAUGGCUACAUAUAUGUGCCGGGCUCAUUUGGUGAGCAAU---UAAA ((((((((((....))).(((((((.(((..(((((..(..((((((...))))))..)..)))))..)))(((.(((....)))))))))))))....)))))))..---.... ( -42.20) >DroEre_CAF1 55450 114 - 1 UGUUAACCCUGAUCGGAUUGGGUUUAAUGGCACUGCUAGCUUUGUGUUGUGCUCAGAUUCUGUGGCAGCAUGGAUACGUUUAUGUGCCAGGGUCGUUUGGUGAGUGGAA-ACCUC ......((((.((((((((((((...(..((((..........))))..))))))((((((..((((.((((((....))))))))))))))))))))))).)).))..-..... ( -33.70) >DroMoj_CAF1 66376 113 - 1 UGCUCACGCUGCUCGGCUUGAGUCUGGUGGCGCUUCUGGCGAUGUGCUGUGAGCAGAUUCUGCGGCAACACGGCUACGUUUAUGUGCCCGGCUCGUUUGGUAUGUUA--AAAGAC .(((((.(((....))).)))))((..(((((..((..((((...((((...((((...))))((((..(((....))).....))))))))))))..))..)))))--..)).. ( -34.90) >DroPer_CAF1 59655 114 - 1 UGCUCACCUUUCUGGGUUUGGGCCUGAUGGCCCUGCUGGCCCUGUGCUGUGCCCAGAUUCUCUGGCAGCACGGCUACAUCUAUGUGCCAGGAUCCUUUGGUGAGCAAUGGAGUA- ((((((((.....(((((.(((((....)))))..(((((.(((((((((((((((.....))))..))))))).))).....).))))))))))...))))))))........- ( -55.60) >consensus UGCUCACCCUGCUCGGUUUGGGCCUGAUGGCACUGCUAGCCUUGUGCUGUGCACAGAUUCUGUGGCAGCACGGCUACAUUUAUGUGCCAGGAUCGUUUGGUGAGUAAU__AGCA_ .(((((((.....(((.((.(((..((((((.......)).(((((((((.(.(((...))).))))))))))...)))).....))).)).)))...))))))).......... (-21.87 = -21.27 + -0.61)
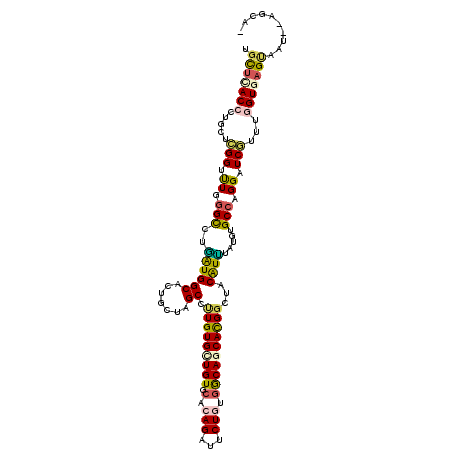

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:54:04 2006