
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,379,756 – 11,379,996 |
| Length | 240 |
| Max. P | 0.999896 |

| Location | 11,379,756 – 11,379,876 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.42 |
| Mean single sequence MFE | -46.74 |
| Consensus MFE | -41.06 |
| Energy contribution | -40.86 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.27 |
| Structure conservation index | 0.88 |
| SVM decision value | 4.43 |
| SVM RNA-class probability | 0.999896 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11379756 120 + 20766785 GAAAUCCUUGGAGUGGUUCUCCGACAGAGUCUCUAGCACGUUGGUCUUAAGUUUUUCGUUUGCCGUCAUUUGGGCGGACAGAAACUUAGCCAACAUGCCGGCGCUCUUCAGCGGACUCUU .......((((((.....)))))).((((((.(..(((.((((((..(((((((((.(((((((........)))))))))))))))))))))).)))..)((((....)))))))))). ( -48.30) >DroSec_CAF1 7014 120 + 1 AAAGUCCUUGGAGUGGUUCUCCGACAGAGUUUCCAGCACGUUGGUCUUAAGCUUCUCGUUUGCCGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGAACUCUU ...(((...((((.....)))))))((((((.((.(((..(((((..((((.((((.(((((((........))))))))))).)))))))))..))).))((((....)))))))))). ( -44.50) >DroSim_CAF1 8447 120 + 1 AAAGUCCUUGGAGUGGUUCUCCGACAGAGUUUCCAGCACGUUGGUCUUUAGUUUCUCGUUUGACGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGAACUCUU ...(((...((((.....)))))))((((((.((.(((..((((.((..(((((((.(((...((((.....)))))))))))))).))))))..))).))((((....)))))))))). ( -43.40) >DroEre_CAF1 7171 120 + 1 GAAGUCCUUGGGGUGGUUCUCCGACAGAGUUUCCAACACGUUGGUCUUGAGUUUCUCGUUUGCUCCCAUUUGGGCGGACAGAAACUUAGCCAAUGUGCCGACGCUCUUCAGCGGACUCUU ..(((((..((((.....))))((.(((((.((...(((((((((..(((((((((.((((((((......))))))))))))))))))))))))))..)).)))))))...)))))... ( -51.40) >DroYak_CAF1 7314 120 + 1 GAAGUCCUUGGAGUGGUUCUCCGACAGAGUCUCCAGCACGUUUGUCUUGAGUUUAUCGUUUGCUCCCAUUUGGGCGGACAGAAACUUGGACAAUGUGCCGGCGCUCUUCAGCGGAUUCUU ..(((((..((((.....))))((.((((.(.((.(((((((.((((.((((((...((((((((......))))))))..))))))))))))))))).)).)))))))...)))))... ( -46.10) >consensus GAAGUCCUUGGAGUGGUUCUCCGACAGAGUUUCCAGCACGUUGGUCUUAAGUUUCUCGUUUGCCGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGGACUCUU .......((((((.....)))))).((((((.((.(((.((((((..(((((((((.(((((((........)))))))))))))))))))))).))).))((((....)))))))))). (-41.06 = -40.86 + -0.20)

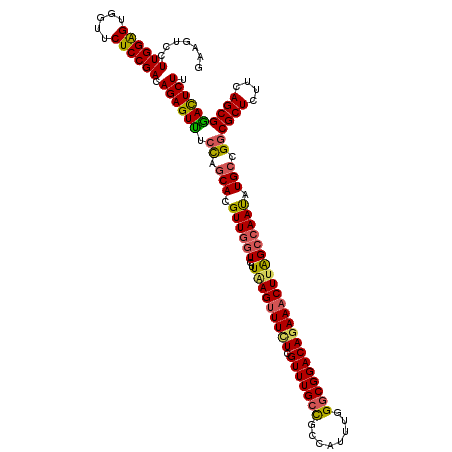
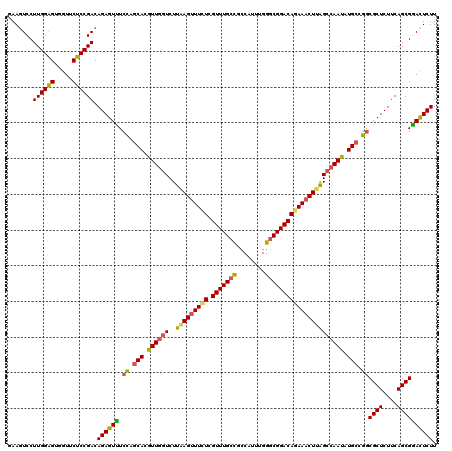
| Location | 11,379,756 – 11,379,876 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.42 |
| Mean single sequence MFE | -43.90 |
| Consensus MFE | -34.72 |
| Energy contribution | -36.24 |
| Covariance contribution | 1.52 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.34 |
| Structure conservation index | 0.79 |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.996937 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11379756 120 - 20766785 AAGAGUCCGCUGAAGAGCGCCGGCAUGUUGGCUAAGUUUCUGUCCGCCCAAAUGACGGCAAACGAAAAACUUAAGACCAACGUGCUAGAGACUCUGUCGGAGAACCACUCCAAGGAUUUC ..((((((...(((((((.(..(((((((((((((((((.(((..(((........)))..)))..)))))).)).)))))))))..).).)))).))((((.....))))..)))))). ( -43.40) >DroSec_CAF1 7014 120 - 1 AAGAGUUCGCUGAAGAGCGCCGGCAUAUUGGCUAAGUUUCUGUCCGCCCAAAUGGCGGCAAACGAGAAGCUUAAGACCAACGUGCUGGAAACUCUGUCGGAGAACCACUCCAAGGACUUU .(((((((...((((((..((((((..(((((((((((((((((((((.....)))))...)).)))))))).)).))))..))))))...)))).))((((.....))))..))))))) ( -51.60) >DroSim_CAF1 8447 120 - 1 AAGAGUUCGCUGAAGAGCGCCGGCAUAUUGGCUAAGUUUCUGUCCGCCCAAAUGGCGUCAAACGAGAAACUAAAGACCAACGUGCUGGAAACUCUGUCGGAGAACCACUCCAAGGACUUU .(((((((...((((((..((((((..((((((.(((((((((.((((.....))))....)).)))))))..)).))))..))))))...)))).))((((.....))))..))))))) ( -45.70) >DroEre_CAF1 7171 120 - 1 AAGAGUCCGCUGAAGAGCGUCGGCACAUUGGCUAAGUUUCUGUCCGCCCAAAUGGGAGCAAACGAGAAACUCAAGACCAACGUGUUGGAAACUCUGUCGGAGAACCACCCCAAGGACUUC ..((((((...((((((..(((((((.((((((.(((((((((..((((.....)).))..)).)))))))..)).)))).)))))))...)))).))((.(......)))..)))))). ( -40.00) >DroYak_CAF1 7314 120 - 1 AAGAAUCCGCUGAAGAGCGCCGGCACAUUGUCCAAGUUUCUGUCCGCCCAAAUGGGAGCAAACGAUAAACUCAAGACAAACGUGCUGGAGACUCUGUCGGAGAACCACUCCAAGGACUUC ..((((((...(((((((.(((((((.(((((..(((((.(((..((((.....)).))..)))..)))))...)))))..))))))).).)))).))((((.....))))..))).))) ( -38.80) >consensus AAGAGUCCGCUGAAGAGCGCCGGCAUAUUGGCUAAGUUUCUGUCCGCCCAAAUGGCGGCAAACGAGAAACUCAAGACCAACGUGCUGGAAACUCUGUCGGAGAACCACUCCAAGGACUUC ..((((((...((((((..(((((((.(((((..((((((((((((((.....)))))...)).)))))))...).)))).)))))))...)))).))((((.....))))..)))))). (-34.72 = -36.24 + 1.52)

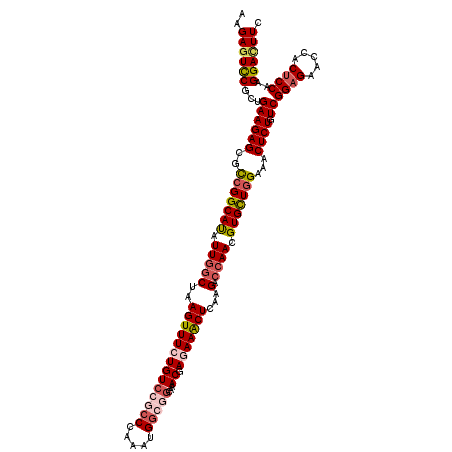
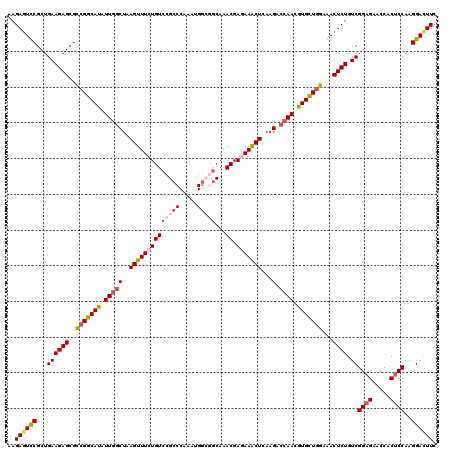
| Location | 11,379,796 – 11,379,916 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.42 |
| Mean single sequence MFE | -40.70 |
| Consensus MFE | -36.28 |
| Energy contribution | -36.96 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.38 |
| SVM RNA-class probability | 0.993187 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11379796 120 + 20766785 UUGGUCUUAAGUUUUUCGUUUGCCGUCAUUUGGGCGGACAGAAACUUAGCCAACAUGCCGGCGCUCUUCAGCGGACUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGUAAUUGC (((((..(((((((((.(((((((........)))))))))))))))))))))..(((.(((((((((..((((....))))..))).)))))))))..((((.(....)))))...... ( -41.80) >DroSec_CAF1 7054 120 + 1 UUGGUCUUAAGCUUCUCGUUUGCCGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGAACUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGUAAUUGC (((((..((((.((((.(((((((........))))))))))).)))))))))..(((.(((((((((..((((....))))..))).)))))))))..((((.(....)))))...... ( -41.10) >DroSim_CAF1 8487 120 + 1 UUGGUCUUUAGUUUCUCGUUUGACGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGAACUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGUAAUUGC ((((.((..(((((((.(((...((((.....)))))))))))))).))))))..(((.(((((((((..((((....))))..))).)))))))))..((((.(....)))))...... ( -40.00) >DroEre_CAF1 7211 120 + 1 UUGGUCUUGAGUUUCUCGUUUGCUCCCAUUUGGGCGGACAGAAACUUAGCCAAUGUGCCGACGCUCUUCAGCGGACUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGUAAUUGC (((((..(((((((((.((((((((......)))))))))))))))))))))).((((...((((....))))(((....)))......)))).((((.((((.(....)))))..)))) ( -42.00) >DroYak_CAF1 7354 120 + 1 UUUGUCUUGAGUUUAUCGUUUGCUCCCAUUUGGGCGGACAGAAACUUGGACAAUGUGCCGGCGCUCUUCAGCGGAUUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGCAAUUGC .((((((.((((((...((((((((......))))))))..))))))))))))..(((((((((((((..((((....))))..))).))))))((.............))))))..... ( -38.62) >consensus UUGGUCUUAAGUUUCUCGUUUGCCGCCAUUUGGGCGGACAGAAACUUAGCCAAUAUGCCGGCGCUCUUCAGCGGACUCUUGUCAAAGUAGCGCUGCAAAACCGACUCCAGCGGUAAUUGC (((((..(((((((((.(((((((........)))))))))))))))))))))..(((.(((((((((..((((....))))..))).)))))))))..((((.(....)))))...... (-36.28 = -36.96 + 0.68)

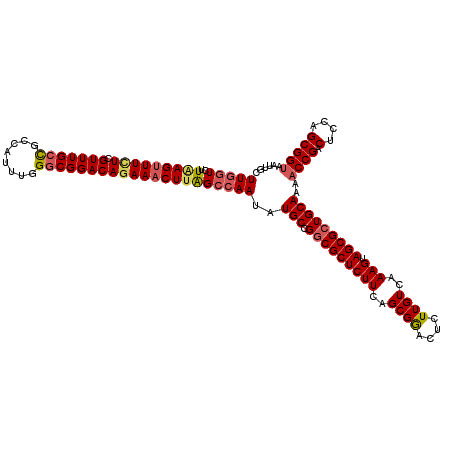
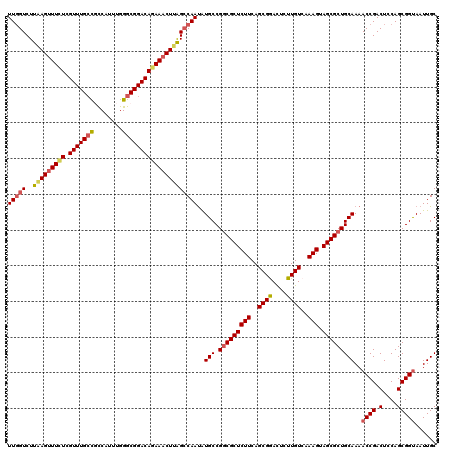
| Location | 11,379,876 – 11,379,996 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.50 |
| Mean single sequence MFE | -46.06 |
| Consensus MFE | -39.28 |
| Energy contribution | -39.48 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.873733 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11379876 120 - 20766785 GAAGUGGAGUCUGAACUCGAGACAGCCGGCCAGGAUCUAGCCAUCAUGGAUCCAGAAGCCGCUCUUGAUCUGGCGGUGGCGCAAUUACCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.(.(((....(((((((((.(((..(((((((.......)))))))....))))))..(((((..(((((((.....)))))))..)).))))))))))))).)))))))... ( -44.70) >DroSec_CAF1 7134 120 - 1 GAAGUGGAGUCUGAACUCGAGCCAGCAAGCCAAGAUCUAGCCAUCAUGGAUCCACAAGCCGCUCUUGAUCUGGCGGUGGCGCAAUUACCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.((..((((.(((.(((((..((((.((((.(((...(.((......)).)..)))...))))))))(((((.....))))))))))..)))))))....)).)))))))... ( -41.00) >DroSim_CAF1 8567 120 - 1 GAAGUGGAGUCUGAACACGAGCCAGCAGGCCAGGAUCUAGCCAUCAUGGAUCCAGAAGCCGCUCUUGAUCUGGCGGUGGCGCAAUUACCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.(.(((.....((((((((.(((..(((((((.......)))))))....))))))...((((..(((((((.....)))))))..)).)))))))..)))).)))))))... ( -46.00) >DroEre_CAF1 7291 120 - 1 GAAGUGGAGUCUGAUCCCGACCCAGCUGGUCAGGAUCUAGCCAUCAUGGAUCCAGAGGCCGCACUCGAUCUGGUGGUGGAGCAAUUACCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.(.(((...((((((((((.((((.(((((((.......))))))).(((......))))))).((((((......))))))))))).))))).....)))).)))))))... ( -51.90) >DroYak_CAF1 7434 120 - 1 GAAGUGGAGUCUGAACCAGAGCCAGCUGGUCAGGAUCUAGCCAUCAUUGAUCCAGAGGCCGCACUUGACCUGGUGGUGGAGCAAUUGCCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.(.(((...(((((((.((.((((.(((((...........)))))...))))))....((((..((((..(.....)..))))..).)))))))))))))).)))))))... ( -46.70) >consensus GAAGUGGAGUCUGAACCCGAGCCAGCAGGCCAGGAUCUAGCCAUCAUGGAUCCAGAAGCCGCUCUUGAUCUGGCGGUGGCGCAAUUACCGCUGGAGUCGGUUUUGCAGCGCUACUUUGAC (((((((.(.(((.....(((((.((.(((..(((((((.......)))))))....)))))....((((..(((((((.....)))))))..).))))))))..)))).)))))))... (-39.28 = -39.48 + 0.20)

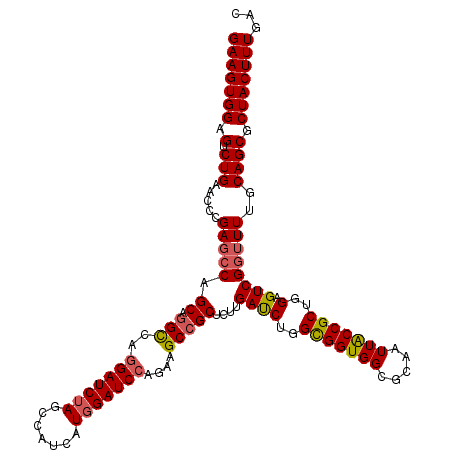
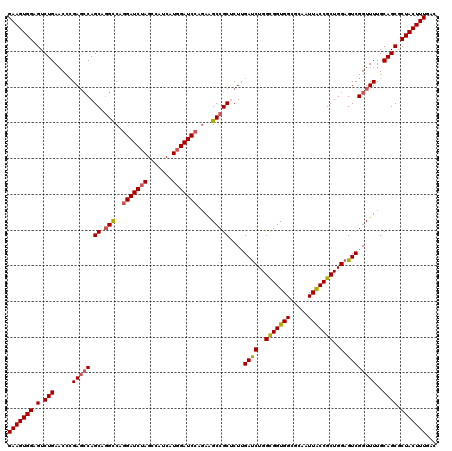
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:53:29 2006