
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,148,281 – 11,148,375 |
| Length | 94 |
| Max. P | 0.976530 |

| Location | 11,148,281 – 11,148,375 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.50 |
| Mean single sequence MFE | -25.63 |
| Consensus MFE | -13.97 |
| Energy contribution | -15.03 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976530 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
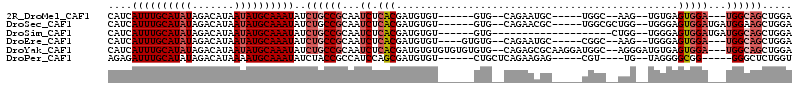
>2R_DroMel_CAF1 11148281 94 + 20766785 UCCAGCUGCCA---UCCACUCACA--CUU--GCCA-----GCAUUCUG--CAC------ACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG ....((((.((---..........--..)--).))-----))..((((--(.(------.((.(((....))))).))))))(((((((((((.......)))))))))))... ( -28.60) >DroSec_CAF1 63061 99 + 1 UCCAGCUUCCAUCAUCCACUCCCA--CCAGCGCCA-----GCGUUCUG--CAC------ACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG .........((((...........--...((....-----))..((((--(.(------.((.(((....))))).)))))).((((((((((.......)))))))))))))) ( -26.30) >DroSim_CAF1 61535 86 + 1 UCCAGCUGCCAUCAUCCACUCCCA--CCAG--------------------CAC------ACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG ....(((((.(((.....((....--..))--------------------..(------((.....))).))).)))))...(((((((((((.......)))))))))))... ( -21.90) >DroEre_CAF1 65738 96 + 1 UCCAGCUGCCA---UCCACUCCCA--CUU--GCCG-----GCAUUCUG--CACAC----ACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG ....((((.((---..........--..)--).))-----))..((((--(.(.(----(...(((....))))).))))))(((((((((((.......)))))))))))... ( -26.60) >DroYak_CAF1 62589 107 + 1 UCCAGCUGCCA---UCCACUCACAUCCCU--GCCAUCCUUGCGCUCUG--CACACACACACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG .....(((((.---....(((((......--((.......))((...)--)...............))))).....))))).(((((((((((.......)))))))))))... ( -26.10) >DroPer_CAF1 60603 92 + 1 ACCAGAGCCC-----CCGCCCCUA--CA----ACG-----CUCUUCUGAGCAG------ACACAUCGCUGGAUGGCGGUAGAUAUUUGCAUUUUAUGUCUAUAUGCAAAUCUCU ..........-----(((((((..--(.----..(-----(((....)))).(------(....)))..))..))))).(((.((((((((...........)))))))).))) ( -24.30) >consensus UCCAGCUGCCA___UCCACUCCCA__CCU__GCCA_____GCAUUCUG__CAC______ACACAUCGUGAGAUUGCGGCAGAUAUUUGCAUAUUAUGUCUAUAUGCAAAUGAUG .....(((((....(((((...............................................))).))....))))).(((((((((((.......)))))))))))... (-13.97 = -15.03 + 1.06)


| Location | 11,148,281 – 11,148,375 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.50 |
| Mean single sequence MFE | -30.82 |
| Consensus MFE | -14.14 |
| Energy contribution | -15.20 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.771918 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
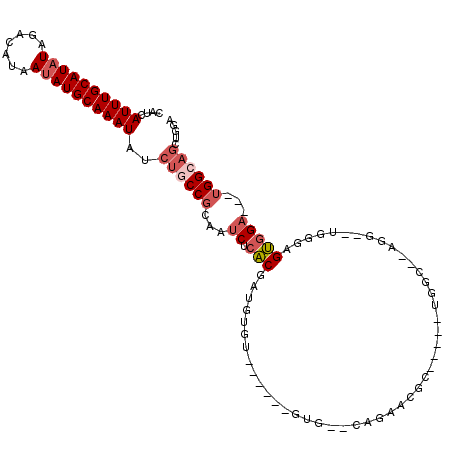
>2R_DroMel_CAF1 11148281 94 - 20766785 CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGU------GUG--CAGAAUGC-----UGGC--AAG--UGUGAGUGGA---UGGCAGCUGGA ....((((((((((.......))))))))))..(((((((...((((((...(((------..(--(.....))-----..))--)..--))))))..).---))))))..... ( -31.50) >DroSec_CAF1 63061 99 - 1 CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGU------GUG--CAGAACGC-----UGGCGCUGG--UGGGAGUGGAUGAUGGAAGCUGGA ((((((((((((((.......)))))))))).....((((..((((((.(.((((------..(--(.....))-----..))))).)--)))))))))..))))......... ( -32.10) >DroSim_CAF1 61535 86 - 1 CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGU------GUG--------------------CUGG--UGGGAGUGGAUGAUGGCAGCUGGA ....((((((((((.......))))))))))..((((((((.((((((.(.((..------..)--------------------)).)--))))).....)).))))))..... ( -25.60) >DroEre_CAF1 65738 96 - 1 CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGU----GUGUG--CAGAAUGC-----CGGC--AAG--UGGGAGUGGA---UGGCAGCUGGA ....((((((((((.......))))))))))..(((((((..((((((..(((..----((((.--....))))-----..))--).)--)))))...).---))))))..... ( -30.30) >DroYak_CAF1 62589 107 - 1 CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGUGUGUGUGUG--CAGAGCGCAAGGAUGGC--AGGGAUGUGAGUGGA---UGGCAGCUGGA ....((((((((((.......))))))))))..(((((((...(((((.((.(.(((...((((--(...)))))......))--).).)))))))..).---))))))..... ( -33.20) >DroPer_CAF1 60603 92 - 1 AGAGAUUUGCAUAUAGACAUAAAAUGCAAAUAUCUACCGCCAUCCAGCGAUGUGU------CUGCUCAGAAGAG-----CGU----UG--UAGGGGCGG-----GGGCUCUGGU ((((((((((((...........))))))))..(..(((((..((.(((((((.(------((....)))...)-----)))----))--).)))))))-----..)))))... ( -32.20) >consensus CAUCAUUUGCAUAUAGACAUAAUAUGCAAAUAUCUGCCGCAAUCUCACGAUGUGU______GUG__CAGAACGC_____UGGC__AGG__UGGGAGUGGA___UGGCAGCUGGA ....((((((((((.......))))))))))..((((((...((.(((...............................................)))))...))))))..... (-14.14 = -15.20 + 1.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:51:16 2006