
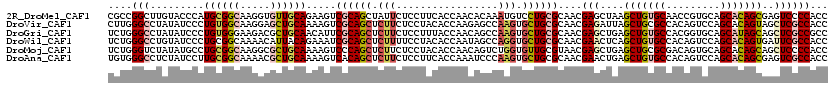
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 11,029,568 – 11,029,688 |
| Length | 120 |
| Max. P | 0.790154 |

| Location | 11,029,568 – 11,029,688 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.67 |
| Mean single sequence MFE | -41.39 |
| Consensus MFE | -23.27 |
| Energy contribution | -24.55 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.790154 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11029568 120 + 20766785 CGCCGGCUUGUACCCAUGCGGCAAGGUGUUGCAGAAGUCGCAGCUAUUCUCCUUCACCAACACAAAUGUCCUGCGCAACGAGCUAAGCUGUGCAACCGUGCAGCACAGCGAGUCCCCACC (((.(((((((.....(((((((..((((((..((((..(........)..))))..))))))...)))....)))))))))))..((((..(....)..))))...))).......... ( -36.00) >DroVir_CAF1 173066 120 + 1 CUUGGGCCUAUAUCCCUGUGGCAAGGAGCUGCAAAAGUCGCAGCUCUUCUCCUACACCAAGAGCCAAGUGCUGCGCAACGAGAUUAGCUGCGCCACAGUCCAGCACAGUAGCUCGCCACC ...(((.......))).(((((.(((((((((.......)))))))))............((((...((((((((((.(.......).)))))........)))))....))))))))). ( -43.50) >DroGri_CAF1 168593 120 + 1 UCUGGGCCUAUAUCCCUGUGGGAAGACGCUGCAACAUUCGCAGCUCUUCUCCUUUACCAACAGCCAAGUGCUGCGCAACGAGCUGAGCUGUGCCACGGUGCAGCAUAGCAGCUCGCCGCC ....(((.........((((((((((.(((((.......)))))))))))..........((((.....))))))))..((((((.((((..(....)..))))....)))))))))... ( -48.40) >DroWil_CAF1 189664 120 + 1 UCUGGGCCUGUAUCCCUGCGGCAAAACAUUACAGAAAUCGCAGCUCUUUUCCUACACCAAUAGCCAGGUGCUGCGCAACGAACUCAGCUGUGCCACAGUCCAGCACAGUGAUUCGCCACC ...(((.......)))...(((................((((((.(((...(((......)))..))).))))))....(((.(((.((((((.........)))))))))))))))... ( -32.50) >DroMoj_CAF1 200219 120 + 1 UCUGGGUCUAUAUGCCUGCGGCAAGGCGCUGCAAAAGUCCCAGCUCUUCUCCUACACCAACAGUCUGGUGUUGCGUAACGAGCUGAGCUGCGCGACAGUGCAGCACAGCAGCUCCCCACC ..((((.......((((......))))(((((........((((((....((.((((((......)))))).).)....)))))).(((((((....)))))))...)))))..)))).. ( -52.10) >DroAna_CAF1 145808 120 + 1 UGUGGGCCUCUAUCCUUGCGGCAAAACGCUGCAAAAGUCACAGCUCUUCUCCUUCACCAAAUCCCAAGUGCUGCGCAACGAACUGAGCUGUGCCACAGUCCAGCACAGCGAGUCGCCACC .((((((........(((((((.....)))))))......((((.(((.................))).)))).))..(((.((..(((((((.........))))))).))))))))). ( -35.83) >consensus UCUGGGCCUAUAUCCCUGCGGCAAGACGCUGCAAAAGUCGCAGCUCUUCUCCUACACCAACAGCCAAGUGCUGCGCAACGAGCUGAGCUGUGCCACAGUCCAGCACAGCAACUCGCCACC ....(((.........((((((.....)))))).....((((((.(((.................))).))))))....(((....(((((((.........)))))))..))))))... (-23.27 = -24.55 + 1.28)
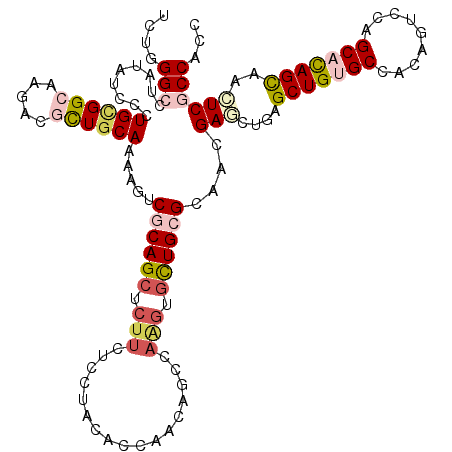
| Location | 11,029,568 – 11,029,688 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.67 |
| Mean single sequence MFE | -47.97 |
| Consensus MFE | -30.16 |
| Energy contribution | -31.88 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.619673 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 11029568 120 - 20766785 GGUGGGGACUCGCUGUGCUGCACGGUUGCACAGCUUAGCUCGUUGCGCAGGACAUUUGUGUUGGUGAAGGAGAAUAGCUGCGACUUCUGCAACACCUUGCCGCAUGGGUACAAGCCGGCG ((..(((....(((((((.((...)).))))))).......(((((.(((.((....)).)))..((((..(........)..)))).))))).)))..))((...(((....))).)). ( -43.00) >DroVir_CAF1 173066 120 - 1 GGUGGCGAGCUACUGUGCUGGACUGUGGCGCAGCUAAUCUCGUUGCGCAGCACUUGGCUCUUGGUGUAGGAGAAGAGCUGCGACUUUUGCAGCUCCUUGCCACAGGGAUAUAGGCCCAAG .((((((((((((.(.((..(..(((.(((((((.......))))))).))).)..)).).....)))).....(((((((((...))))))))))))))))).(((.......)))... ( -51.90) >DroGri_CAF1 168593 120 - 1 GGCGGCGAGCUGCUAUGCUGCACCGUGGCACAGCUCAGCUCGUUGCGCAGCACUUGGCUGUUGGUAAAGGAGAAGAGCUGCGAAUGUUGCAGCGUCUUCCCACAGGGAUAUAGGCCCAGA .(((((((((((((.(((..(...)..))).)))..))))))))))(((((.....))))).......((.((((((((((((...))))))).)))))))...(((.......)))... ( -54.60) >DroWil_CAF1 189664 120 - 1 GGUGGCGAAUCACUGUGCUGGACUGUGGCACAGCUGAGUUCGUUGCGCAGCACCUGGCUAUUGGUGUAGGAAAAGAGCUGCGAUUUCUGUAAUGUUUUGCCGCAGGGAUACAGGCCCAGA .(..(((((((((((((((.......))))))).))).)))))..)((.(((((........))))).((...(((((((((.....))))..))))).)))).(((.......)))... ( -42.00) >DroMoj_CAF1 200219 120 - 1 GGUGGGGAGCUGCUGUGCUGCACUGUCGCGCAGCUCAGCUCGUUACGCAACACCAGACUGUUGGUGUAGGAGAAGAGCUGGGACUUUUGCAGCGCCUUGCCGCAGGCAUAUAGACCCAGA ..((((...(((..((((((((..(((.(.((((((..(((.((((((((((......)))).)))))))))..))))))))))...))))))))..((((...))))..))).)))).. ( -50.80) >DroAna_CAF1 145808 120 - 1 GGUGGCGACUCGCUGUGCUGGACUGUGGCACAGCUCAGUUCGUUGCGCAGCACUUGGGAUUUGGUGAAGGAGAAGAGCUGUGACUUUUGCAGCGUUUUGCCGCAAGGAUAGAGGCCCACA .((((.(.(((((((((((.......)))))))).....((.((((....((((........))))..((..(((.((((..(...)..)))).)))..)))))).))..))).))))). ( -45.50) >consensus GGUGGCGAGCCGCUGUGCUGCACUGUGGCACAGCUCAGCUCGUUGCGCAGCACUUGGCUGUUGGUGAAGGAGAAGAGCUGCGACUUUUGCAGCGCCUUGCCGCAGGGAUAUAGGCCCAGA .(((((((((.(((((((((.....)))))))))...)))))))))((..((((........))))..((......((((((.....))))))......)))).(((.......)))... (-30.16 = -31.88 + 1.73)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:50:33 2006