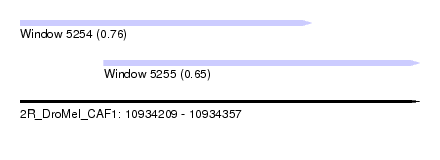
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,934,209 – 10,934,357 |
| Length | 148 |
| Max. P | 0.758446 |

| Location | 10,934,209 – 10,934,317 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.84 |
| Mean single sequence MFE | -40.46 |
| Consensus MFE | -27.48 |
| Energy contribution | -27.06 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.758446 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10934209 108 + 20766785 ---------GCAGGUUCUUCGGUAAAAUCUUCAAGUGGUCGAAGAAGUCGUAGGAGGAGGAUCUUUGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG---GCGGCAGCAUG ---------((...((((((((................))))))))(((((..((((.....))))((((.(((((((((((...))))))))))).)))).....---))))).))... ( -41.89) >DroVir_CAF1 64437 99 + 1 ---------GCAGAUUCUUUGGCAAAAUCUUCAAGUGGUCCAAAAAAUCGUAA------GUUCUUCGGGUGGAGGGGGCCACGCGGUGGUUCCCUUAGCCCCCAGG------GCAGCAUG ---------((.((((.(((((....(((.......)))))))).))))....------(((((..((((.(((((((((((...))))))))))).))))..)))------)).))... ( -39.20) >DroGri_CAF1 62298 99 + 1 ---------ACAGAUUCUUUGGCAAAAUCUUCAAGUGGUCCAAAAAAUCGUAG------UUUCUUCGGGUGGAGGGGGCCACGGGGUGGUUCCCUUAGCCCACAGG------GCAGCAUG ---------...((((.(((((....(((.......)))))))).))))((.(------((.((..((((.(((((((((((...))))))))))).))))..)))------)).))... ( -34.30) >DroWil_CAF1 84355 108 + 1 ACCAUUGAUUUAGAUUCUUCGGCAAAAUCUUCAAGUGGUCGAAAAAAUCGUAA------AUUCUUUGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG------GCAGCAUG .(((..((((((((((.((((((...((......)).))))))..)))).)))------).))..)))((((((((((((((...))))))))))).((((...))------))..))). ( -38.20) >DroMoj_CAF1 71168 99 + 1 ---------ACAGAUUCUUUGGGAAAAUCUUCAAGUGGUCGAAGAAAUCGUAG------UUUCUUCGGGUGGAGGGGACCACACGGUGGUUCCCUUAGCCCACAGG------GCAGCAUG ---------...(((..((((((......))))))..)))((((((((....)------)))))))((((.(((((((((((...))))))))))).)))).....------........ ( -39.60) >DroPer_CAF1 45188 114 + 1 AACGUGCUCGCAGAUUCUUCGGUAAAAUCUUCAAGUGGUCGAAGAAAUCGUAA------GUUCUUCGGGUGGAGGGGGCCACGAGGUGGUUCCCUUAGCCCCCGGGCCGGCGGCAGCAUG ..(((((((((((((((....)...))))).....(((((((((((..(....------)))))))((((.(((((((((((...))))))))))).))))...))))))))..)))))) ( -49.60) >consensus _________GCAGAUUCUUCGGCAAAAUCUUCAAGUGGUCGAAAAAAUCGUAA______GUUCUUCGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG______GCAGCAUG ............(((..((((((...((......)).))))))...)))((..........(((..((((.(((((((((((...))))))))))).))))..))).........))... (-27.48 = -27.06 + -0.42)

| Location | 10,934,240 – 10,934,357 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.22 |
| Mean single sequence MFE | -42.87 |
| Consensus MFE | -34.42 |
| Energy contribution | -33.95 |
| Covariance contribution | -0.47 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.650358 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10934240 117 + 20766785 GAAGAAGUCGUAGGAGGAGGAUCUUUGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG---GCGGCAGCAUGGCGUCCUACAACGGCGUGCUCAAGGAUUACAGUCACAGCA ......((.((.((.....(((((((((((.(((((((((((...))))))))))).(((...(((---(((.(.....).)))))).....)))..)))))))))))....)))).)). ( -47.80) >DroVir_CAF1 64468 108 + 1 CAAAAAAUCGUAA------GUUCUUCGGGUGGAGGGGGCCACGCGGUGGUUCCCUUAGCCCCCAGG------GCAGCAUGGCGUCCUAUCAGGGUGUGCUCAAGGAUUACAGUCACAGCA .............------(((((..((((.(((((((((((...))))))))))).))))..)))------)).((.(((((((((....(((....))).)))))....))))..)). ( -39.90) >DroPse_CAF1 44559 114 + 1 GAAGAAAUCGUAA------GUUCUUCGGGUGGAGGGGGCCACGAGGUGGUUCCCUUAGCCCCCGGGCCGGCGGCAGCAUGGCGUCUUACGGCGGCGUGCUCAAGGACUACAGCCACAGCA ((((((..(....------)))))))((((.(((((((((((...))))))))))).))))....((.(..(((((((((.((((....)))).))))))...(.....).))).).)). ( -52.00) >DroGri_CAF1 62329 108 + 1 CAAAAAAUCGUAG------UUUCUUCGGGUGGAGGGGGCCACGGGGUGGUUCCCUUAGCCCACAGG------GCAGCAUGGCGUCCUAUAAUGGUGUACUCAAGGAUUACAGUCACAGCA .........((((------((.(((.((((.(((((((((((...))))))))))).))))..(((------((.((...)))))))..............))))))))).......... ( -36.80) >DroWil_CAF1 84395 108 + 1 GAAAAAAUCGUAA------AUUCUUUGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG------GCAGCAUGGCAUCCUAUAACGGUGUGCUAAAGGAUUACAGUCACAACA .........((((------.((((((((((((((((((((((...))))))))))).((((...))------))..))).(((((.......))))).)))))))))))).......... ( -38.20) >DroMoj_CAF1 71199 108 + 1 GAAGAAAUCGUAG------UUUCUUCGGGUGGAGGGGACCACACGGUGGUUCCCUUAGCCCACAGG------GCAGCAUGGCGUCCUAUAAUGGCAUACUCAAGGACUACAGUCACAGCA ((((((((....)------)))))))((((.(((((((((((...))))))))))).))))..(((------((.((...)))))))......((...(....)(((....)))...)). ( -42.50) >consensus GAAAAAAUCGUAA______GUUCUUCGGGUGGAGGGGACCACGAGGUGGUUCCCUUAGCCCCCAGG______GCAGCAUGGCGUCCUAUAACGGCGUGCUCAAGGAUUACAGUCACAGCA .........((((.........((..((((.(((((((((((...))))))))))).))))..)).........((((((.(((......))).))))))......)))).......... (-34.42 = -33.95 + -0.47)
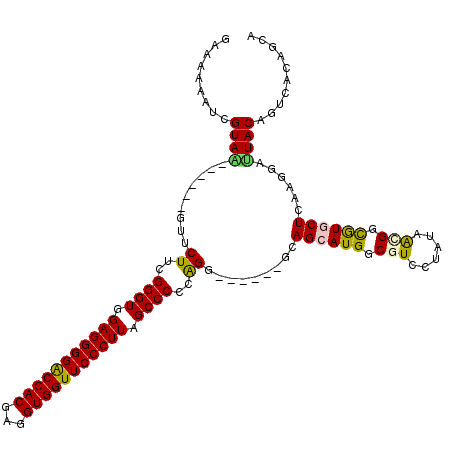
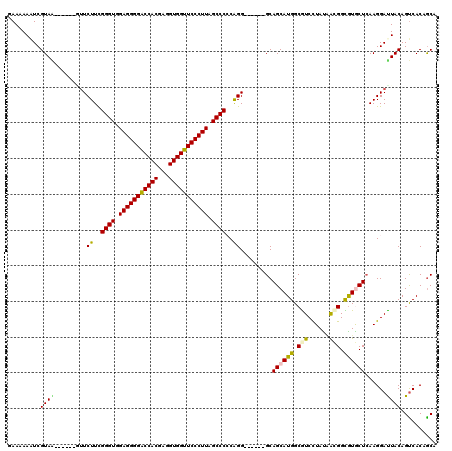
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:49:44 2006