
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,662,051 – 10,662,247 |
| Length | 196 |
| Max. P | 0.829539 |

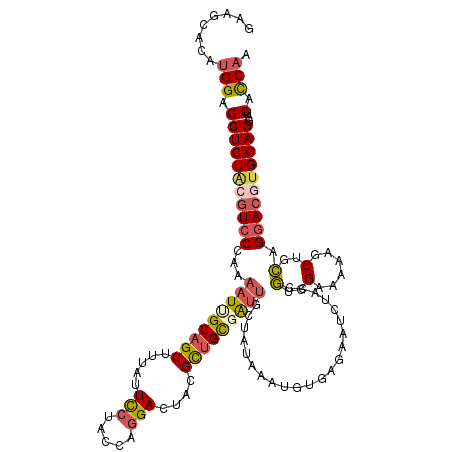
| Location | 10,662,051 – 10,662,171 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.89 |
| Mean single sequence MFE | -37.98 |
| Consensus MFE | -26.05 |
| Energy contribution | -26.75 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.561724 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
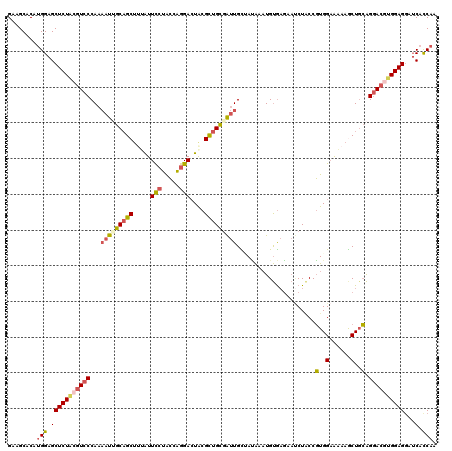
>2R_DroMel_CAF1 10662051 120 - 20766785 GAAGCACAUGGAGCUCUGCGUCCCAAAAUUGCAGCUUUAUUCCUACCAGGACUGCGCUGCGAUUGCUACAAAUGUGAGAAUCUACCGUGGAAAAAGCUGCAGGACGUGGAGGAUCAUCAA .......((((..(((..(((((......(((((((((..(((.....)))(..((.((.((((..(((....)))..)))))).))..)..))))))))))))))..)))..))))... ( -40.40) >DroSec_CAF1 95549 120 - 1 GAAGCACAUGGAGCUCUACGUCCCAAAAUUGCAGCUUUAUUCCUACCAGGACUACGCUGUUAUUGCUAUAAAUGUGAGAAUCUACCGUGGAAAAAGCUUCAGGACGUGGAGGAUCACCAA ..........((.((((((((((......((.((((((..(((.....)))(((((..(((.(..(.......)..).)))....)))))..)))))).))))))))))))..))..... ( -32.20) >DroSim_CAF1 96755 120 - 1 GAAGCACAAGGAGCUCUACGUCCCAAAAUUGCAGCUUUAUUCCUACCAGGACUACGCUGCGAUUGUUACAAAUGUGAGAAUCUACCGUGGAAAAAGCUGCAGGACUUGGAGGAUCACCAA ..........((.(((((.((((......(((((((((..(((.....)))(((((.((.((((.((((....)))).)))))).)))))..))))))))))))).)))))..))..... ( -39.90) >DroEre_CAF1 96676 120 - 1 GAAGCACAUGGAGCUCUACGUCCCAAAAUCGCAGCUACAUUCCUGCCAGGACUACGCUGCGAUUGUAAUAAAUGUGAGAAUCUACCGUGGAAAAAACUGCAGGACGUGGAGGAUCACCAA ..........((.((((((((((...(((((((((.....(((.....)))....)))))))))......................(..(......)..).))))))))))..))..... ( -37.60) >DroYak_CAF1 103881 120 - 1 GAAGCCCAUGGAGCUCUGCGUCCCAAAAUUGCUGCUUUAUUCCUACCAGGACUACGCUGCGAUUGCAAUAAAUGUGAGAAGCUGCCGUGGAAAAAGCUGCAGGACGUGGAGGAUCACCAA ..........((.(((..(((((......(((.(((((..(((.....)))((((((.((..(..((.....))..)...)).).)))))..))))).))))))))..)))..))..... ( -38.90) >DroAna_CAF1 129612 120 - 1 GAAGCUCAUGGAGCUCUUCGUCCCACCAUAGCAGCGCCGUUUGUGGCGGGAGUACGUUGUGGCUGUCAGGAUUGCACCGUCCUUCCUUGGCGGAAACUGAGGCAACUGGAGCAGCACCAA ........(((.(((((((...((.(((.......((((....))))(((((.(((.((..(((....)).)..)).))).))))).))).))......((....)))))).))).))). ( -38.90) >consensus GAAGCACAUGGAGCUCUACGUCCCAAAAUUGCAGCUUUAUUCCUACCAGGACUACGCUGCGAUUGCUAUAAAUGUGAGAAUCUACCGUGGAAAAAGCUGCAGGACGUGGAGGAUCACCAA ........(((.(((((((((((...(((((((((.....(((.....)))....)))))))))......................(..(......)..).))))))))))...).))). (-26.05 = -26.75 + 0.70)

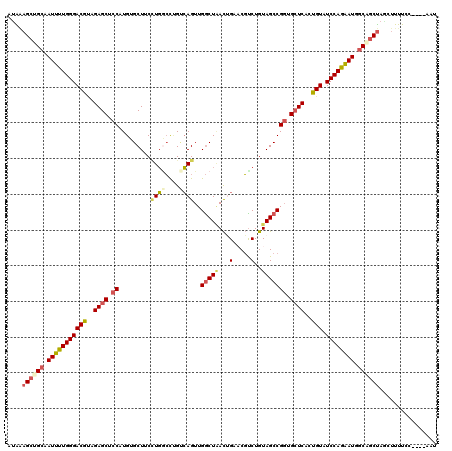
| Location | 10,662,131 – 10,662,247 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.84 |
| Mean single sequence MFE | -40.92 |
| Consensus MFE | -30.66 |
| Energy contribution | -31.25 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.829539 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
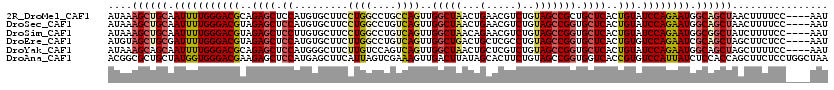
>2R_DroMel_CAF1 10662131 116 + 20766785 AUAAAGCUGCAAUUUUGGGACGCAGAGCUCCAUGUGCUUCCUGGCCUGCCAGUUGGCUAACUGAACGUCUGUAGCCGCUGCUCACUGUAUCCAGAAUGGCAGCUAACUUUUCC----AAU ....((((((.(((((((((((..((((..(.........((((....))))..(((((...(.....)..))))))..))))..))).)))))))).)))))).........----... ( -39.40) >DroSec_CAF1 95629 116 + 1 AUAAAGCUGCAAUUUUGGGACGUAGAGCUCCAUGUGCUUCCUGGCCUGUCAGUUGGCUAACUGAACGUCUGUAGCCGGUGCUCACUGUAUCCAGAAUGGCAGCUAACUUUUCC----AAU ....((((((.(((((((((((..((((.((.........((((....))))..(((((...(.....)..))))))).))))..))).)))))))).)))))).........----... ( -40.60) >DroSim_CAF1 96835 116 + 1 AUAAAGCUGCAAUUUUGGGACGUAGAGCUCCUUGUGCUUCCUGGCCUGUCAGUUGGCUAACAGAACGUCUGUAGCCGGUGCUCACUGUAUCCAGAAUGGCGGCUAUCUUUUCC----AAU ....((((((.(((((((((((..((((.((.........((((....))))..(((((.(((.....)))))))))).))))..))).)))))))).)))))).........----... ( -43.20) >DroEre_CAF1 96756 116 + 1 AUGUAGCUGCGAUUUUGGGACGUAGAGCUCCAUGUGCUUCUUGGCCUGUCAGUUGGCUGACUGCUCGCCUGUAGCCGGUGCUCACUGUGUCCAGAAUCGCAGCUAGCUUCUCC----AAU ...(((((((((((((((((((..((((.((..(.(((....(((..(((((....))))).....)))...)))))).))))..))).))))))))))))))))........----... ( -50.10) >DroYak_CAF1 103961 116 + 1 AUAAAGCAGCAAUUUUGGGACGCAGAGCUCCAUGGGCUUCUUGUCCAGUCAGUUGGCUAACUGCUCGUCUGUAGCCGGUGCUCACUGUAUCCAGAAUGGCAGCUAGCUUUUCC----AAU ..((((((((.(((((((((((..((((.((.(((((.....))))).......(((((.(.........)))))))).))))..))).))))))))....))).)))))...----... ( -38.70) >DroAna_CAF1 129692 120 + 1 ACGGCGCUGCUAUGGUGGGACGAAGAGCUCCAUGAGCUUCAUUAGUCGAAAGUUGACUUAUAGCACUUCUGUAGCCGGUGGUCACCGUGUCCAUUAUCUCCACCAGCUUCUCCUGGCUAA ..((.(..(((..((((((((...((((((...)))))).....)))((.(((.(((((((((.....)))))).((((....)))).))).))).)).))))))))..).))....... ( -33.50) >consensus AUAAAGCUGCAAUUUUGGGACGUAGAGCUCCAUGUGCUUCCUGGCCUGUCAGUUGGCUAACUGAACGUCUGUAGCCGGUGCUCACUGUAUCCAGAAUGGCAGCUAGCUUUUCC____AAU ....((((((.(((((((((((..((((.((.........((((....))))..(((((...(.....)..))))))).))))..))).)))))))).))))))................ (-30.66 = -31.25 + 0.59)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:48:01 2006