
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,657,947 – 10,658,046 |
| Length | 99 |
| Max. P | 0.969914 |

| Location | 10,657,947 – 10,658,046 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.44 |
| Mean single sequence MFE | -47.03 |
| Consensus MFE | -26.73 |
| Energy contribution | -28.07 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.645735 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
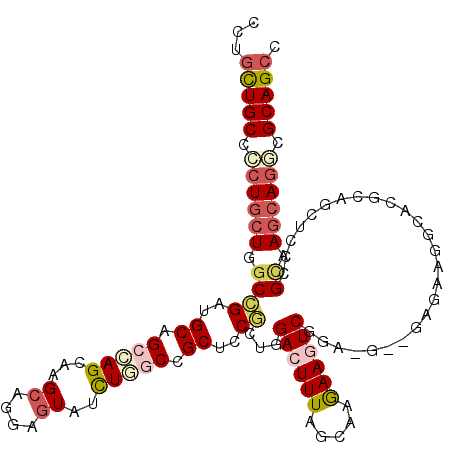
>2R_DroMel_CAF1 10657947 99 + 20766785 CCUAUUGCCACUGCUUGCGGAUGCGGCCAGUCAGCAAGAGUAUCUCGCUGCACCGCUGGACUUUAGCAAGAAGUC------------------CAGCUCGCGCAAGCAGGCGCAGCC ....((((..((((((((((.((((((.((...((....))..)).))))))))((((((((((.....))))))------------------))))....))))))))..)))).. ( -48.60) >DroVir_CAF1 138920 117 + 1 GCUGCUGCCACUGCUGGCCGAUGCAGCUAGCAAGCAAGAAUAUUUAACGGCACCGCUGGACUUUAGCAAAAAGUCAGAGGCAGAGACGGCGCGCAGCUCACGUAAACAGUCGCAGCC (((((((((..(((((((.......)))))))...............((((...))))((((((.....))))))...))))..(((.(((.(.....).))).....)))))))). ( -36.70) >DroPse_CAF1 116227 117 + 1 CUUGCUGCCCCUCCUGGCCGAUGCAGCCAGCAAGCAGGAGUAUCUGGCCGCUCCGCUGGACUUUAGCAAGAAGUCGGAUGGCGAGAACGGACGCAGCUCCCGCAAGCAGGCGCAGCC ...(((((.(((.(((((.......)))))...((.((((...(((((((.(((((((((((((.....))))))...))))).)).))).).))))))).))....))).))))). ( -45.70) >DroGri_CAF1 127916 117 + 1 GCUGCUGCCGCUGCUUGCCGAUGCGGCCAGCAAGCAGGAGUAUUUAACGGCGCCGCUCGACUUUAGCAAGAAAUCGGAAGCGGAAACGGCGCGCAGUUCGCGCAAGCAAGCGCAGCC ((((((((.((((((.((((...)))).))).)))..............(((((((((((.(((.....))).)))..)))(....)))))))))(((.((....)).)))))))). ( -49.40) >DroAna_CAF1 125775 111 + 1 CUUGCUGCCCCUGCUGGCGGAUGCGGCCAGCAAGCAGGAGUACCUGGCUGCUCCCCUGGACUUUAGCAAGAAAUCGGA------GGAGGCUCGCAGUUCCCGGAAGCAGGCGCAGCC ...(((((.((((((..(((.(((((((.(((..((((....))))..)))(((.((((..(((....)))..)))).------)))))).))))....)))..)))))).))))). ( -51.10) >DroPer_CAF1 117520 117 + 1 CUUGCUGCCCCUGCUGGCCGAUGCAGCCAGCAAGCAGGAGUAUCUGGCCGCUCCGCUGGACUUUAGCAAGAAGUCGGAUGGCGAGAACGGACGCAGCUCCCGCAAGCAGGCGCAGCC ...(((((.(((((((((.......))).....((.((((...(((((((.(((((((((((((.....))))))...))))).)).))).).))))))).)).)))))).))))). ( -50.70) >consensus CCUGCUGCCCCUGCUGGCCGAUGCAGCCAGCAAGCAGGAGUAUCUGGCCGCUCCGCUGGACUUUAGCAAGAAGUCGGA_G__GAGAAGGCACGCAGCUCCCGCAAGCAGGCGCAGCC ...(((((.((((((.((((..((.(((((...((....))..))))).))..))...((((((.....))))))..........................)).)))))).))))). (-26.73 = -28.07 + 1.34)

| Location | 10,657,947 – 10,658,046 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.44 |
| Mean single sequence MFE | -52.50 |
| Consensus MFE | -31.31 |
| Energy contribution | -31.45 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.969914 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
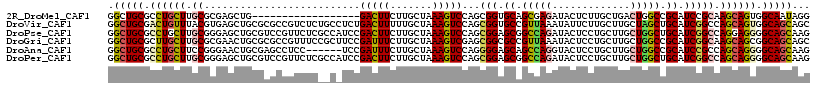
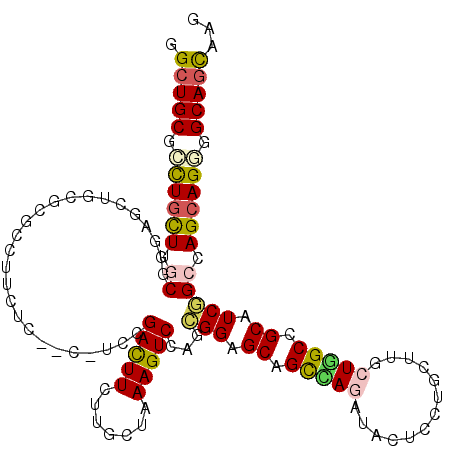
>2R_DroMel_CAF1 10657947 99 - 20766785 GGCUGCGCCUGCUUGCGCGAGCUG------------------GACUUCUUGCUAAAGUCCAGCGGUGCAGCGAGAUACUCUUGCUGACUGGCCGCAUCCGCAAGCAGUGGCAAUAGG ...(((..(((((((((.((((((------------------(((((.......)))))))))(((.(((((((.....)))))))....)))...)))))))))))..)))..... ( -50.30) >DroVir_CAF1 138920 117 - 1 GGCUGCGACUGUUUACGUGAGCUGCGCGCCGUCUCUGCCUCUGACUUUUUGCUAAAGUCCAGCGGUGCCGUUAAAUAUUCUUGCUUGCUAGCUGCAUCGGCCAGCAGUGGCAGCAGC .(((((.((((((..((((((((((((((((...(((.....((((((.....))))))))))))))).((...........))..)).)))).)).))...)))))).)))))... ( -42.20) >DroPse_CAF1 116227 117 - 1 GGCUGCGCCUGCUUGCGGGAGCUGCGUCCGUUCUCGCCAUCCGACUUCUUGCUAAAGUCCAGCGGAGCGGCCAGAUACUCCUGCUUGCUGGCUGCAUCGGCCAGGAGGGGCAGCAAG .(((((.(((((..((((((((((.(.((((((.(((.....(((((.......)))))..)))))))))))))...)))))))..))((((((...))))))..))).)))))... ( -57.30) >DroGri_CAF1 127916 117 - 1 GGCUGCGCUUGCUUGCGCGAACUGCGCGCCGUUUCCGCUUCCGAUUUCUUGCUAAAGUCGAGCGGCGCCGUUAAAUACUCCUGCUUGCUGGCCGCAUCGGCAAGCAGCGGCAGCAGC .(((((........((((.....))))((((...(((((..((((((.......)))))))))))...............(((((((((((.....))))))))))))))).))))) ( -48.70) >DroAna_CAF1 125775 111 - 1 GGCUGCGCCUGCUUCCGGGAACUGCGAGCCUCC------UCCGAUUUCUUGCUAAAGUCCAGGGGAGCAGCCAGGUACUCCUGCUUGCUGGCCGCAUCCGCCAGCAGGGGCAGCAAG .(((((.((((((..((((...((((.(((..(------(((.....(((....)))....))))(((((.((((....)))).))))))))))))))))..)))))).)))))... ( -56.30) >DroPer_CAF1 117520 117 - 1 GGCUGCGCCUGCUUGCGGGAGCUGCGUCCGUUCUCGCCAUCCGACUUCUUGCUAAAGUCCAGCGGAGCGGCCAGAUACUCCUGCUUGCUGGCUGCAUCGGCCAGCAGGGGCAGCAAG .(((((.(((....((((((((((.(.((((((.(((.....(((((.......)))))..)))))))))))))...)))))))..((((((((...))))))))))).)))))... ( -60.20) >consensus GGCUGCGCCUGCUUGCGGGAGCUGCGCGCCUUCUC__C_UCCGACUUCUUGCUAAAGUCCAGCGGAGCAGCCAGAUACUCCUGCUUGCUGGCCGCAUCGGCCAGCAGGGGCAGCAAG .(((((.((((((.((..........................(((((.......)))))...(((.((.(((((.............))))).)).))))).)))))).)))))... (-31.31 = -31.45 + 0.14)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:47:59 2006