| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,419,182 – 10,419,290 |
| Length | 108 |
| Max. P | 0.844687 |

| Location | 10,419,182 – 10,419,290 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.00 |
| Mean single sequence MFE | -46.87 |
| Consensus MFE | -30.61 |
| Energy contribution | -31.17 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.710201 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
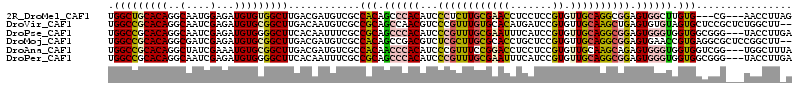
>2R_DroMel_CAF1 10419182 108 + 20766785 UGGCUGCACAGGCAAUGGAGAUGUGUGGCUUGACGAUGUCGCCACAGCCCACAUCCCUCUUGCGAACCUCCUCCGUGUUGCAGGCGGAGUGGCUUGUG---CG---AACCUUAG .((.(((((((((...((.((((((.((((((.((....)).)).)))))))))))).........(((((.((........)).)))).))))))))---))---..)).... ( -44.70) >DroVir_CAF1 31412 112 + 1 UGGCCGCACAGGCAAUCGAGAUGUGCGGCUUGACAAUGUCGCCGCAGCCAACGUCCCGUUUGUGCACAUGAUCCGUGUUGCAAGCUGAGUGUGUAGUGCUCCGCUCUGGCUU-- .(((((((((..(....)...))))))))).(((...)))((((.(((.........(((((((.(((((...)))))))))))).((((.......)))).))).))))..-- ( -37.70) >DroPse_CAF1 21241 111 + 1 UGGCCGCACAGGCAAUCGAGAUGUGGGGCUUCACAAUUUCGCCGCAGCCCACAUCCCGUUUGCGAAUUUCAUCCGUGUUGCAGGCGGAGUGGGUGGUGGCGGG---UACCUUGA .((((.((((..(....)...)))).))))...(((.((((((((.((((((...((((((((((....(....)..)))))))))).)))))).))))))))---....))). ( -48.50) >DroMoj_CAF1 30487 112 + 1 UGGCCGCACAGGCGAUCGAGAUGUGCGGCUUGACGAUGUCGCCACAGCCGACGUCUCGCUUGCGCACCUGCUCCGUGUUGCAGGCGGAGUGAACCGUGAGGCGCUCCGGCUU-- .((((((.(((((....(((((((.(((((((.((....)).)).))))))))))))))))).)).(((((........))))).((((((..((....)))))))))))).-- ( -57.40) >DroAna_CAF1 27284 111 + 1 UGGCCGCACAGGCUAUCGAAAUGUGCGGCUUGACGAUGUCGCCACAACCCACAUCCCGUUUCCGGACCUCCUCCGUGUUGCAAGCAGAGUGGGUGGUGGUCGG---UGGCUUUA .(((((((((..(....)...))))))))).(((...)))(((((...((((...(((....)))(((..(((.((.......)).)))..))).))))...)---)))).... ( -44.40) >DroPer_CAF1 27327 111 + 1 UGGCCGCACAGGCAAUCGAGAUGUGGGGCUUCACAAUUUCGCCGCAGCCCACAUCCCGUUUGCGAAUUUCAUCCGUGUUGCAGGCGGAGUGGGUGGUGGCGGG---UACCUUGA .((((.((((..(....)...)))).))))...(((.((((((((.((((((...((((((((((....(....)..)))))))))).)))))).))))))))---....))). ( -48.50) >consensus UGGCCGCACAGGCAAUCGAGAUGUGCGGCUUGACAAUGUCGCCACAGCCCACAUCCCGUUUGCGAACCUCAUCCGUGUUGCAGGCGGAGUGGGUGGUGGCGCG___UACCUU_A .(((((((((..(....)...)))))))))............(((.((((((...((((((((((((.......)).)))))))))).)))))).)))................ (-30.61 = -31.17 + 0.56)

| Location | 10,419,182 – 10,419,290 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.00 |
| Mean single sequence MFE | -43.87 |
| Consensus MFE | -27.38 |
| Energy contribution | -28.22 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.844687 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10419182 108 - 20766785 CUAAGGUU---CG---CACAAGCCACUCCGCCUGCAACACGGAGGAGGUUCGCAAGAGGGAUGUGGGCUGUGGCGACAUCGUCAAGCCACACAUCUCCAUUGCCUGUGCAGCCA ....((((---.(---((((((((.(((((.........)))))..)))).((((..((((((((((((.(((((....))))))))).))))))))..)))).))))))))). ( -53.30) >DroVir_CAF1 31412 112 - 1 --AAGCCAGAGCGGAGCACUACACACUCAGCUUGCAACACGGAUCAUGUGCACAAACGGGACGUUGGCUGCGGCGACAUUGUCAAGCCGCACAUCUCGAUUGCCUGUGCGGCCA --..(((.((((.(((.........))).))))(((.((((.....))))..(((.(((((.(((((((..((((....)))).))))).)).))))).)))....)))))).. ( -34.90) >DroPse_CAF1 21241 111 - 1 UCAAGGUA---CCCGCCACCACCCACUCCGCCUGCAACACGGAUGAAAUUCGCAAACGGGAUGUGGGCUGCGGCGAAAUUGUGAAGCCCCACAUCUCGAUUGCCUGUGCGGCCA ....((..---.((((((.....((.((((.........))))))......((((.(((((((((((((...(((....)))..)).))))))))))).)))).)).)))))). ( -38.40) >DroMoj_CAF1 30487 112 - 1 --AAGCCGGAGCGCCUCACGGUUCACUCCGCCUGCAACACGGAGCAGGUGCGCAAGCGAGACGUCGGCUGUGGCGACAUCGUCAAGCCGCACAUCUCGAUCGCCUGUGCGGCCA --..(((((((.(((....)))...))))((((((........))))))(((((.((((((.(((((((.(((((....)))))))))).)).))))....)).)))))))).. ( -52.20) >DroAna_CAF1 27284 111 - 1 UAAAGCCA---CCGACCACCACCCACUCUGCUUGCAACACGGAGGAGGUCCGGAAACGGGAUGUGGGUUGUGGCGACAUCGUCAAGCCGCACAUUUCGAUAGCCUGUGCGGCCA ....((((---(...((((.(((..(((((.........)))))..)))(((....)))...))))...)))))(((...)))..((((((((...........)))))))).. ( -46.00) >DroPer_CAF1 27327 111 - 1 UCAAGGUA---CCCGCCACCACCCACUCCGCCUGCAACACGGAUGAAAUUCGCAAACGGGAUGUGGGCUGCGGCGAAAUUGUGAAGCCCCACAUCUCGAUUGCCUGUGCGGCCA ....((..---.((((((.....((.((((.........))))))......((((.(((((((((((((...(((....)))..)).))))))))))).)))).)).)))))). ( -38.40) >consensus U_AAGCCA___CCCACCACCACCCACUCCGCCUGCAACACGGAGGAGGUUCGCAAACGGGAUGUGGGCUGCGGCGACAUCGUCAAGCCGCACAUCUCGAUUGCCUGUGCGGCCA .................................((..(((((.........((((.(((((((((((((...(((....)))..)))).))))))))).)))))))))..)).. (-27.38 = -28.22 + 0.84)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:45:24 2006