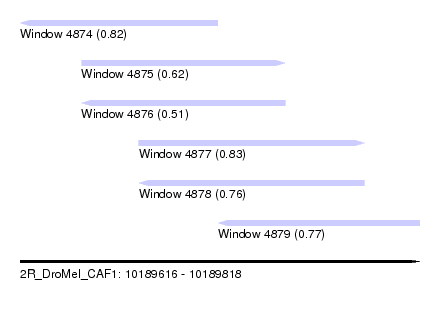
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,189,616 – 10,189,818 |
| Length | 202 |
| Max. P | 0.826075 |

| Location | 10,189,616 – 10,189,716 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 85.06 |
| Mean single sequence MFE | -46.38 |
| Consensus MFE | -38.41 |
| Energy contribution | -38.88 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.821983 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
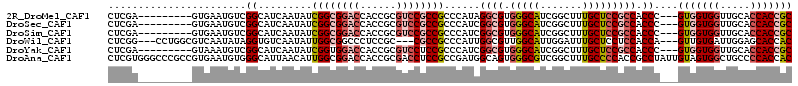
>2R_DroMel_CAF1 10189616 100 - 20766785 CACCAACUGUGAUCGUCGGAGCACCACCACCACCGCCCAUGGUCGUGAUGCCGCCGCCACGGCCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAG ......(((.(.((..((((((.((((((((((.((....(((((((..((....)))))))))....)).))))).)))))....))))))..)))))) ( -46.20) >DroSec_CAF1 17642 100 - 1 CACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAUGGUCGUGAUGCCACCGCCACGGCCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAC ........(((..(..((((((.((((((((((.((....(((((((..((....)))))))))....)).)))).))))))....))))))..)..))) ( -50.00) >DroSim_CAF1 17877 100 - 1 CACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAUGGUCGUGAUGCCACCGCCACGGCCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAC ........(((..(..((((((.((((((((((.((....(((((((..((....)))))))))....)).)))).))))))....))))))..)..))) ( -50.00) >DroEre_CAF1 22497 94 - 1 ------CUGUGAUCGUCGGAGCACCGCCUCCACCACCCAUGGUCGUGAUGCCGCCGCCACGUCCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAC ------..(((..(..((((((...(((((((((((..(((((((((..((....))))))....))))).)))))))).)))...))))))..)..))) ( -46.50) >DroWil_CAF1 21768 94 - 1 CGCCA---GUGAUCAUUGGAGCUCCACC---GCCGCCUGUUAUAGUUGCACCACCGCCCCGGCCAGCUGUUGUGGUGGGUGGUCAGCCAGCAGUUGCCAC .....---(..((..((((....(((((---((.((........)).))..((((((..(((....)))..)))))))))))....))))..))..)... ( -31.60) >DroYak_CAF1 18303 100 - 1 CACCUACUGUGAUCGUCGGAGCACCACCUCCACCGCCCAUGGUCGUGGUGCCACCGCCACGACCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAC ........(((..(..((((((.((((((((((.((....((((((((((....))))))))))....)).)))).))))))....))))))..)..))) ( -54.00) >consensus CACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAUGGUCGUGAUGCCACCGCCACGGCCCACCGUUGUGGUGGGUGGCCAGGCUCCGGUGGCCAC ........(((..(((((((((.(((((..(((.((.....)).)))....((((((.((((....)))).)))))))))))....)))))))))..))) (-38.41 = -38.88 + 0.48)

| Location | 10,189,647 – 10,189,750 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -47.55 |
| Consensus MFE | -23.37 |
| Energy contribution | -25.93 |
| Covariance contribution | 2.56 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.621523 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10189647 103 + 20766785 CGGUGGGCCGUGGCGGC-----GGC-AUCACGACCAUGGGCGGUGGUGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAA (.((.(.((((((.((.-----.((-(((((..((((.(((.(((((.((((....)).)).))))).))).))))..)))))))..)).)))))).).)).)...... ( -53.60) >DroVir_CAF1 22995 106 + 1 CUGCGGGUCGUGGCAGUCCGUAGGUUUCGACAACAACUGGCGGUGGUGGUGGUGCAGCACCGAUUACAAUUGGU---GGCGCACCAAUAACGACUGGCGGCGGUGCGAA (((((((.(......))))))))..((((...((..(((.((((.((..((((((..(((((((....))))))---)..))))))...)).)))).)))..)).)))) ( -47.50) >DroSec_CAF1 17673 103 + 1 CGGUGGGCCGUGGCGGU-----GGC-AUCACGACCAUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAA (.((.(.((((((.(((-----.((-(((((..((((.(((.((((..((((....)).))..)))).))).))))..))))))).))).)))))).).)).)...... ( -51.10) >DroSim_CAF1 17908 103 + 1 CGGUGGGCCGUGGCGGU-----GGC-AUCACGACCAUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAA (.((.(.((((((.(((-----.((-(((((..((((.(((.((((..((((....)).))..)))).))).))))..))))))).))).)))))).).)).)...... ( -51.10) >DroWil_CAF1 21799 97 + 1 CAGCUGGCCGGGGCGGU-----GGU-GCAACUAUAACAGGCGGC---GGUGGAGCUCCAAUGAUCAC---UGGCGGUGGUGCUCCAAUCACAACUGGUGGAGGAGCAAA ..(((..(((((((..(-----(.(-((..((......))..))---).))..))))).....((((---..(..(((((......)))))..)..)))).)))))... ( -34.00) >DroYak_CAF1 18334 103 + 1 CGGUGGGUCGUGGCGGU-----GGC-ACCACGACCAUGGGCGGUGGAGGUGGUGCUCCGACGAUCACAGUAGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAA (.((.(.((((((.(((-----.((-(((((..((((..((.((((..((((....)).))..)))).))..))))..))))))).))).)))))).).)).)...... ( -48.00) >consensus CGGUGGGCCGUGGCGGU_____GGC_ACCACGACCAUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAA (.((.(.((((((.(((......((.(((((..((((.(((.((((...(((....)))....)))).))).))))..))))))).))).)))))).).)).)...... (-23.37 = -25.93 + 2.56)


| Location | 10,189,647 – 10,189,750 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -34.95 |
| Consensus MFE | -21.34 |
| Energy contribution | -23.24 |
| Covariance contribution | 1.90 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.61 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509139 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10189647 103 - 20766785 UUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCACCACCGCCCAUGGUCGUGAU-GCC-----GCCGCCACGGCCCACCG ..(((((((.(....(((((((.....)))))))(((.....)))...).))))))).(((.((((.......)))).)))..-(((-----(......))))...... ( -38.00) >DroVir_CAF1 22995 106 - 1 UUCGCACCGCCGCCAGUCGUUAUUGGUGCGCC---ACCAAUUGUAAUCGGUGCUGCACCACCACCACCGCCAGUUGUUGUCGAAACCUACGGACUGCCACGACCCGCAG ...........((..(((((...(((((((.(---(((..........)))).)))))))......(((...(((........)))...)))......)))))..)).. ( -28.90) >DroSec_CAF1 17673 103 - 1 UUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAUGGUCGUGAU-GCC-----ACCGCCACGGCCCACCG ..(((((((.(....(((((((.....)))))))(((.....)))...).)))))))...........(((..((((.(((..-..)-----)).)))).)))...... ( -37.80) >DroSim_CAF1 17908 103 - 1 UUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAUGGUCGUGAU-GCC-----ACCGCCACGGCCCACCG ..(((((((.(....(((((((.....)))))))(((.....)))...).)))))))...........(((..((((.(((..-..)-----)).)))).)))...... ( -37.80) >DroWil_CAF1 21799 97 - 1 UUUGCUCCUCCACCAGUUGUGAUUGGAGCACCACCGCCA---GUGAUCAUUGGAGCUCCACC---GCCGCCUGUUAUAGUUGC-ACC-----ACCGCCCCGGCCAGCUG ..((((((..(((.....)))...))))))......(((---((....)))))((((...((---(..((.(((.......))-)..-----...))..)))..)))). ( -23.50) >DroYak_CAF1 18334 103 - 1 UUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCUACUGUGAUCGUCGGAGCACCACCUCCACCGCCCAUGGUCGUGGU-GCC-----ACCGCCACGACCCACCG ..(((((((.(....(((((((.....)))))))(((.....)))...).))))))).................(((((((((-(..-----..))))))))))..... ( -43.70) >consensus UUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCACCACCGCCCAUGGUCGUGAU_GCC_____ACCGCCACGGCCCACCG ...((((((.(....(((((((.....)))))))(((.....)))...).))))))..................(((((((................)))))))..... (-21.34 = -23.24 + 1.90)

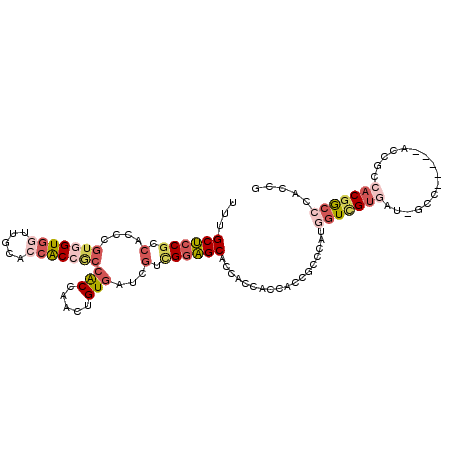
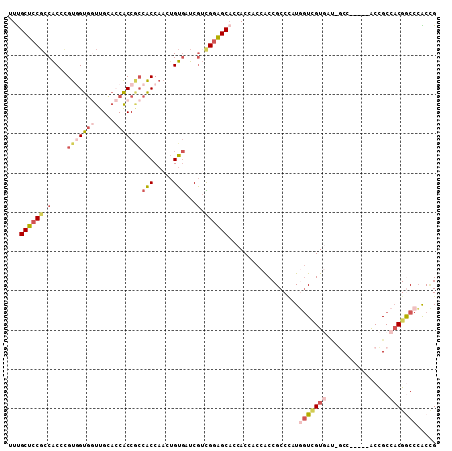
| Location | 10,189,676 – 10,189,790 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 81.46 |
| Mean single sequence MFE | -55.10 |
| Consensus MFE | -36.70 |
| Energy contribution | -39.73 |
| Covariance contribution | 3.03 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.826075 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10189676 114 + 20766785 AUGGGCGGUGGUGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAAGCCGAUGCCCACGCCUAUGGGCGGCGGACGCGGUGGUCCG .((((((....((((..(((((((.(.(((...((.(((((..(.....)..))))))).))).))))))))..)))).))))))((((....)))).(((((......))))) ( -59.20) >DroVir_CAF1 23030 105 + 1 ACUGGCGGUGGUGGUGGUGCAGCACCGAUUACAAUUGGU---GGCGCACCAAUAACGACUGGCGGCGGUGCGAAGCCAAUGCCCACGCCAAUGGGCGGCGGUCGU------GCC .(((.((((.((..((((((..(((((((....))))))---)..))))))...)).)))).))).((..(((.(((...(((((......))))))))..))).------.)) ( -54.10) >DroSec_CAF1 17702 114 + 1 AUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAAGCCGAUGCCCACGCCGAUGGGCGGCGGACGCGGUGGUCCG .((((((.....(((..(((((((.(.(((...((.(((((..(.....)..))))))).))).))))))))..)))..))))))((((....)))).(((((......))))) ( -56.80) >DroSim_CAF1 17937 114 + 1 AUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAAGCCGAUGCCCACGCCGAUGGGCGGCGGACGCGGUGGUCCG .((((((.....(((..(((((((.(.(((...((.(((((..(.....)..))))))).))).))))))))..)))..))))))((((....)))).(((((......))))) ( -56.80) >DroWil_CAF1 21828 105 + 1 ACAGGCGGC---GGUGGAGCUCCAAUGAUCAC---UGGCGGUGGUGCUCCAAUCACAACUGGUGGAGGAGCAAAUCCAAUGCCAACGCCAAUGGGCGGCG---GCGGAGGGCCG ....(((((---(.(((((((((.....((((---..(..(((((......)))))..)..)))).)))))...)))).))))..((((....))))))(---((.....))). ( -46.50) >DroYak_CAF1 18363 114 + 1 AUGGGCGGUGGAGGUGGUGCUCCGACGAUCACAGUAGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAAGCCGAUGCCCACGCCGAUGGGCGGAGGACGCGGUGGUCCA .((((((.....(((..(((((((.(.(((...((.(((((..(.....)..))))))).))).))))))))..)))..))))))((((....))))..((((......)))). ( -57.20) >consensus AUGGGCGGUGGGGGUGGUGCUCCGACGAUCACAGUUGGUGGCGGUGGUGCAACCACCACGGGUGGCGGAGCAAAGCCGAUGCCCACGCCGAUGGGCGGCGGACGCGGUGGUCCG .((((((.....(((..(((((((....((((...((((((..(.....)..))))))...)))))))))))..)))..))))))((((....))))..((((......)))). (-36.70 = -39.73 + 3.03)

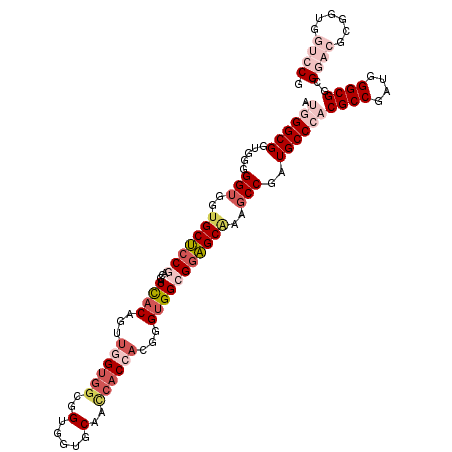
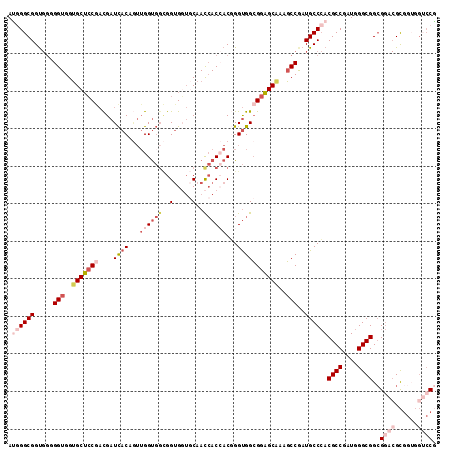
| Location | 10,189,676 – 10,189,790 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 81.46 |
| Mean single sequence MFE | -44.83 |
| Consensus MFE | -30.06 |
| Energy contribution | -32.57 |
| Covariance contribution | 2.51 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.764369 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10189676 114 - 20766785 CGGACCACCGCGUCCGCCGCCCAUAGGCGUGGGCAUCGGCUUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCACCACCGCCCAU (((((......))))).((((....))))(((((...(((........)))....((((((((.((.((..((.(((.....)))..))..)).))))))))))....))))). ( -48.20) >DroVir_CAF1 23030 105 - 1 GGC------ACGACCGCCGCCCAUUGGCGUGGGCAUUGGCUUCGCACCGCCGCCAGUCGUUAUUGGUGCGCC---ACCAAUUGUAAUCGGUGCUGCACCACCACCACCGCCAGU (((------(((((.((.((((((....))))))...(((........)))))..)))))...(((((((.(---(((..........)))).)))))))........)))... ( -43.00) >DroSec_CAF1 17702 114 - 1 CGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAU .((((......))))((((.....))))((((((...((...(((((((.(....(((((((.....)))))))(((.....)))...).)))))))......))...)))))) ( -47.20) >DroSim_CAF1 17937 114 - 1 CGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCCCCACCGCCCAU .((((......))))((((.....))))((((((...((...(((((((.(....(((((((.....)))))))(((.....)))...).)))))))......))...)))))) ( -47.20) >DroWil_CAF1 21828 105 - 1 CGGCCCUCCGC---CGCCGCCCAUUGGCGUUGGCAUUGGAUUUGCUCCUCCACCAGUUGUGAUUGGAGCACCACCGCCA---GUGAUCAUUGGAGCUCCACC---GCCGCCUGU ((((.....((---((.((((....)))).))))..((((..((((((..(((.....)))...))))))......(((---((....)))))...))))..---))))..... ( -38.60) >DroYak_CAF1 18363 114 - 1 UGGACCACCGCGUCCUCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCUACUGUGAUCGUCGGAGCACCACCUCCACCGCCCAU .((((......))))..((((....))))(((((...((...(((((((.(....(((((((.....)))))))(((.....)))...).)))))))......))...))))). ( -44.80) >consensus CGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCCGUGGUGGUUGCACCACCGCCACCAACUGUGAUCGUCGGAGCACCACCACCACCGCCCAU .((((......))))..((((....))))(((((...((...(((((((.(....(((((((.....)))))))(((.....)))...).)))))))...))......))))). (-30.06 = -32.57 + 2.51)

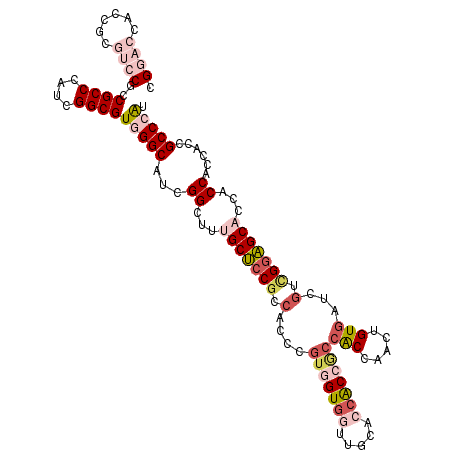
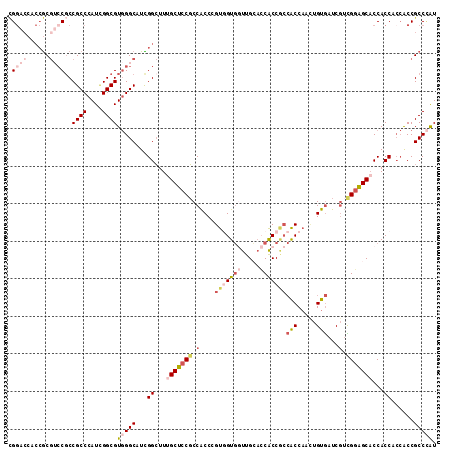
| Location | 10,189,716 – 10,189,818 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 81.47 |
| Mean single sequence MFE | -46.63 |
| Consensus MFE | -30.44 |
| Energy contribution | -31.88 |
| Covariance contribution | 1.45 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.772640 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10189716 102 - 20766785 CUCGA---------GUGAAUGUCGGCAUCAAUAUCGGCGGACCACCGCGUCCGCCGCCCAUAGGCGUGGGCAUCGGCUUUGCUCCGCCACCC---GUGGUGGUUGCACCACCGC ...((---------(..((.(((((..........(((((((......)))))))((((((....)))))).)))))))..)))........---(((((((.....))))))) ( -48.40) >DroSec_CAF1 17742 102 - 1 CUCGA---------GUGAAUGUCGGCAUCAAUAUCGGCGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCC---GUGGUGGUUGCACCACCGC ...((---------(..((.(((((..........(((((((......)))))))((((((....)))))).)))))))..)))........---(((((((.....))))))) ( -48.20) >DroSim_CAF1 17977 102 - 1 CUCGA---------GUGAAUGUCGGCAUCAAUAUCGGCGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCC---GUGGUGGUUGCACCACCGC ...((---------(..((.(((((..........(((((((......)))))))((((((....)))))).)))))))..)))........---(((((((.....))))))) ( -48.20) >DroWil_CAF1 21862 105 - 1 CUCGG---CCUGGCGUCAAUAUAGGUGUCAAUAUUGGCGGCCCUCCGC---CGCCGCCCAUUGGCGUUGGCAUUGGAUUUGCUCCUCCACCA---GUUGUGAUUGGAGCACCAC ....(---((.(((((((((...(((.........((((((.....))---))))))).))))))))))))..(((...((((((..(((..---...)))...))))))))). ( -40.80) >DroYak_CAF1 18403 102 - 1 CUCGA---------GUAAAUGUCGGCAUCAAUAUCGGUGGACCACCGCGUCCUCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCC---GUGGUGGUUGCACCACCGC ...((---------(((((.(((((..........((.((((......)))).))((((((....)))))).))))))))))))........---(((((((.....))))))) ( -43.20) >DroAna_CAF1 17707 114 - 1 CUCGUGGGCCCGCCGUGAAUGUGGGCAUUAACAUUGGCGGACCACCGCGACCUCCGCCGAUGGCAGUGGGCGUCGGCUUUGCCCCACCGCCUAUUGUAGUGGCUGCCCCACCAC ...((((((((((.......)))))).........(((((.((((.(((.....))).....((((((((((..(((...)))....)))))))))).)))))))))))))... ( -51.00) >consensus CUCGA_________GUGAAUGUCGGCAUCAAUAUCGGCGGACCACCGCGUCCGCCGCCCAUCGGCGUGGGCAUCGGCUUUGCUCCGCCACCC___GUGGUGGUUGCACCACCGC .......................((.........((((((((......))))))))......((((.(((((.......))))))))).))....(((((((.....))))))) (-30.44 = -31.88 + 1.45)
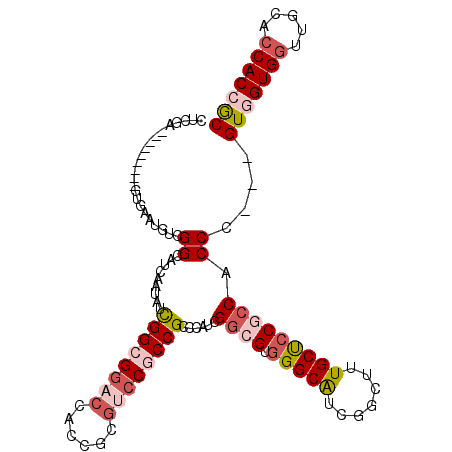
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:43:47 2006