
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 10,008,404 – 10,008,539 |
| Length | 135 |
| Max. P | 0.711486 |

| Location | 10,008,404 – 10,008,499 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.43 |
| Mean single sequence MFE | -22.26 |
| Consensus MFE | -13.53 |
| Energy contribution | -14.53 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.671381 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10008404 95 + 20766785 UCUUGCGGUUACGAAAACAGCCAUAAAACU--GCAAUUUUAAACCGAGCAAAUAGGCUU---GAGCCCAUAAAUAUGUUUAUGCGUUUGCCGGCAAUAA--------------U ..((((((((................))))--)))).......(((.((((((.(((..---..)))((((((....)))))).)))))))))......--------------. ( -23.19) >DroSec_CAF1 11500 95 + 1 UCUUGUGGUUACGAAAACAGCCAUAAAACU--GCAAUUUGAAACCGAGCAAAUAGGCUU---GAGCCCAUAAAUAUGUUUAUGCGUUUGCCAGCAAUAA--------------U ..((((((((........))))))))...(--((.(((((........))))).(((..---((((.((((((....)))))).))))))).)))....--------------. ( -21.80) >DroSim_CAF1 11946 95 + 1 UCUUGCGGUUACGAAAACAGCCAUAAAACU--GCAAUUUGAAACCGAGCAAAUAGGCUU---GAGCCCAUAAAUAUGUUUAUGCGUUUGCCAGCAAUAA--------------U ..((((((((.((((..(((........))--)...)))).))))(.((((((.(((..---..)))((((((....)))))).))))))).))))...--------------. ( -23.40) >DroEre_CAF1 11693 102 + 1 UGUUGCGGUUACGAAAACAGCCAUAAAACU--GCAAUUUUAAGCCGAGCAAAUAGGCUU---GAGCCCAUAAAUAUGUUUAUGCGUUUGCCAGCAAUAAUAUA-------UAAU ((((((((((........))))........--((((..(((((((.........)))))---))((.((((((....)))))).))))))..)))))).....-------.... ( -23.90) >DroYak_CAF1 11979 109 + 1 UCUUGCGGUUACGAAAACAGCCAUAAAACU--GCAAUUUUAAGCCGAGCAAAUAGGCUU---GAGCCCAUAAAUAUGUUUAUGCGUUUGCCAGCAAUAAUAUACAAUAUACAAU ..((((((((................))))--))))......((.(.((((((.(((..---..)))((((((....)))))).))))))).)).................... ( -21.49) >DroPer_CAF1 11528 90 + 1 --------CAACAAAAACAGUCGUAAAAUAGCGCGACUCGACGCUGAGCUAAUAAGCCCCAGGGGCCCAUAAAUAUAUUUAUGCGUUUGACAGCAGAA---------------- --------..........((((((........))))))....((((.........((((...)))).((((((....)))))).......))))....---------------- ( -19.80) >consensus UCUUGCGGUUACGAAAACAGCCAUAAAACU__GCAAUUUGAAACCGAGCAAAUAGGCUU___GAGCCCAUAAAUAUGUUUAUGCGUUUGCCAGCAAUAA______________U ..((((((((........)))).......................(.((((((.(((((...)))))((((((....)))))).))))))).)))).................. (-13.53 = -14.53 + 1.00)

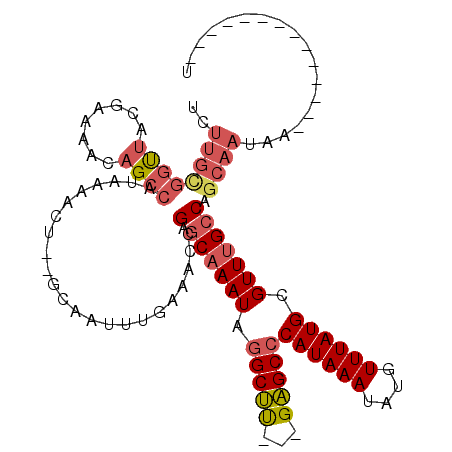

| Location | 10,008,404 – 10,008,499 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.43 |
| Mean single sequence MFE | -23.13 |
| Consensus MFE | -17.73 |
| Energy contribution | -16.73 |
| Covariance contribution | -0.99 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.711486 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10008404 95 - 20766785 A--------------UUAUUGCCGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUUAAAAUUGC--AGUUUUAUGGCUGUUUUCGUAACCGCAAGA .--------------...((((.((((((..((((((....))))))(((..---..)))..)))))).((((.......((--((((....))))))......)))))))).. ( -24.82) >DroSec_CAF1 11500 95 - 1 A--------------UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUUCAAAUUGC--AGUUUUAUGGCUGUUUUCGUAACCACAAGA .--------------........((((((..((((((....))))))(((..---..)))..)))))).((((.......((--((((....))))))......))))...... ( -21.22) >DroSim_CAF1 11946 95 - 1 A--------------UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUUCAAAUUGC--AGUUUUAUGGCUGUUUUCGUAACCGCAAGA .--------------...((((.((((((..((((((....))))))(((..---..)))..)))))).((((.......((--((((....))))))......)))))))).. ( -25.12) >DroEre_CAF1 11693 102 - 1 AUUA-------UAUAUUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGCUUAAAAUUGC--AGUUUUAUGGCUGUUUUCGUAACCGCAACA ....-------.......((((.((((((..((((((....))))))(((..---..)))..)))))).((.(((..(..((--((((....))))))..)..))))))))).. ( -23.70) >DroYak_CAF1 11979 109 - 1 AUUGUAUAUUGUAUAUUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGCUUAAAAUUGC--AGUUUUAUGGCUGUUUUCGUAACCGCAAGA ...(((.((((((.......(((((((((..((((((....))))))(((..---..)))..))))).)))).......)))--)))..))).((.(((.....))).)).... ( -24.54) >DroPer_CAF1 11528 90 - 1 ----------------UUCUGCUGUCAAACGCAUAAAUAUAUUUAUGGGCCCCUGGGGCUUAUUAGCUCAGCGUCGAGUCGCGCUAUUUUACGACUGUUUUUGUUG-------- ----------------....((((.......((((((....))))))(((((...)))))...))))..(((((......)))))......((((.......))))-------- ( -19.40) >consensus A______________UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC___AAGCCUAUUUGCUCGGCUUAAAAUUGC__AGUUUUAUGGCUGUUUUCGUAACCGCAAGA ....................(((((((((..((((((....))))))((((.....))))..))))).))))................((((((......))))))........ (-17.73 = -16.73 + -0.99)

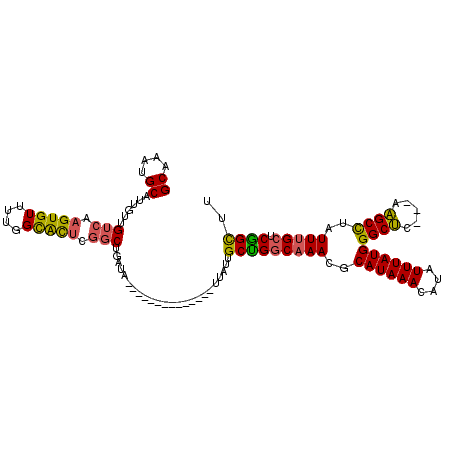
| Location | 10,008,442 – 10,008,539 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 84.50 |
| Mean single sequence MFE | -27.50 |
| Consensus MFE | -22.42 |
| Energy contribution | -21.90 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.666130 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 10008442 97 - 20766785 GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACUCGGCUGAUA--------------UUAUUGCCGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUU ((((((((....(((((.(((((....))))).(((.....--------------.....))))))))..))))..........((((((..---..))))))))))....... ( -27.20) >DroSec_CAF1 11538 97 - 1 GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACCCGGCUGGUA--------------UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUU ((((((((.....(((((((...)))))))....((..(((--------------....)))..))....))))..........((((((..---..))))))))))....... ( -26.80) >DroSim_CAF1 11984 97 - 1 GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACUCGGCUGAUA--------------UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGUUU ((....))..(((((((.(((((....))))).))).))))--------------.....(((((((((..((((((....))))))(((..---..)))..))))).)))).. ( -24.30) >DroEre_CAF1 11731 104 - 1 GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACUUGGCUGAUAUUA-------UAUAUUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGCUU ((....))..(((((((((((((....))))))))).))))...-------.........(((((((((..((((((....))))))(((..---..)))..))))).)))).. ( -30.70) >DroYak_CAF1 12017 111 - 1 GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACUCGGCUGAUAUUGUAUAUUGUAUAUUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC---AAGCCUAUUUGCUCGGCUU (((((((((.(((((((.(((((....))))).))).)))).))))).))))........(((((((((..((((((....))))))(((..---..)))..))))).)))).. ( -33.30) >DroPer_CAF1 11560 98 - 1 GCAAAUGCAUUAUUGUCAAGUGUUUGGGUGUUUUUCUGUU----------------UUCUGCUGUCAAACGCAUAAAUAUAUUUAUGGGCCCCUGGGGCUUAUUAGCUCAGCGU ((....((...........(((((((.((((((..(.((.----------------....)).)..)))))).)))))))....((((((((...))))))))..))...)).. ( -22.70) >consensus GCAAAUGCAUUGUUGUCAAGUGUUUUGGCACUCGGCUGAUA______________UUAUUGCUGGCAAACGCAUAAACAUAUUUAUGGGCUC___AAGCCUAUUUGCUCGGCUU ((....))......(((.(((((....))))).)))........................(((((((((..((((((....))))))((((.....))))..))))).)))).. (-22.42 = -21.90 + -0.52)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:42:15 2006