
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,980,258 – 9,980,386 |
| Length | 128 |
| Max. P | 0.898652 |

| Location | 9,980,258 – 9,980,364 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 90.57 |
| Mean single sequence MFE | -22.20 |
| Consensus MFE | -18.62 |
| Energy contribution | -19.38 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.898652 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
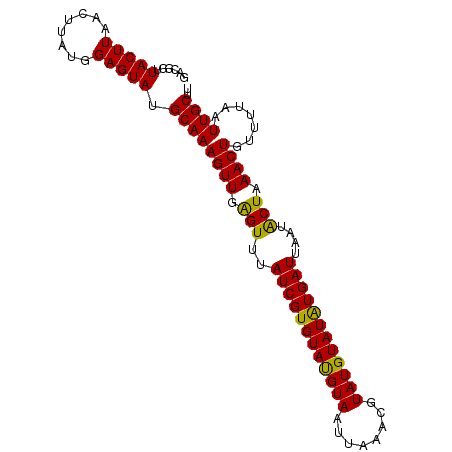
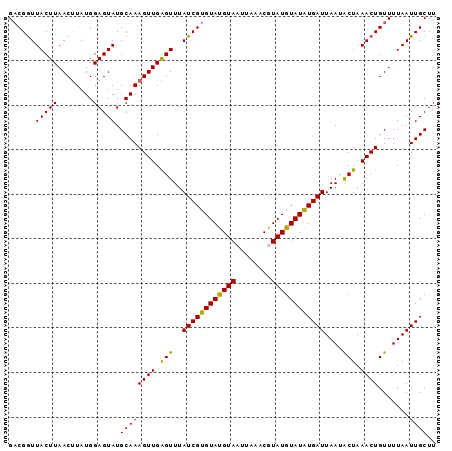
>2R_DroMel_CAF1 9980258 106 - 20766785 UAUGCAAAGUUGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUUUACGUUACAACGUUAUAAGUGGCAAGACCUAU ..(((..((((.(((..(((((((((((((......)))))))))))))....))).)))).........((((.((((....))))...)))).)))........ ( -23.10) >DroSec_CAF1 37352 96 - 1 ----------UGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUUCUAAACUGUUUUAAUUGCUUUACGUUACAACGUUAUAAGUGACAAGACCUAU ----------.(((((.(((((((((((((......))))))))))))).)))))......(((((..(..(((.((((....))))...)))..).))))).... ( -22.30) >DroSim_CAF1 38728 106 - 1 UAUGCAAAGUUGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUUUACGUUACAACGUUAUAAGUGGCAAGACCUAU ..(((..((((.(((..(((((((((((((......)))))))))))))....))).)))).........((((.((((....))))...)))).)))........ ( -23.10) >DroEre_CAF1 37619 105 - 1 UAUGCAAAGUUGGGUUUAUCGUGUAUGUAA-UUAUCGUAUGUAUAUGAUUAAUGCUAAACUGUUUUAAUUGCUUUACGUUACAACGCUAUAAGUGGCAAGACCUAU ..........(((((((((((((((((((.-......))))))))))))...(((((...((((.((((........)))).)))).......)))))))))))). ( -20.00) >DroYak_CAF1 39637 106 - 1 UAUGCAAAGUUGGGUUUAUCGUGUACGUAAAUAAUCGUAUGUAUGUGAUUAAUACUAAACUGUUUUAAUUGCUUUACGUUACAACGCUAUAAGUGGCAAGACCUAU ..........(((((((...(((.(((((((.............(..(((((.((......)).)))))..)))))))))))...((((....)))).))))))). ( -22.51) >consensus UAUGCAAAGUUGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUUUACGUUACAACGUUAUAAGUGGCAAGACCUAU .......((((.(((..(((((((((((((......)))))))))))))....))).))))(((((..(..(((.((((....))))...)))..).))))).... (-18.62 = -19.38 + 0.76)



| Location | 9,980,290 – 9,980,386 |
|---|---|
| Length | 96 |
| Sequences | 4 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 92.71 |
| Mean single sequence MFE | -20.70 |
| Consensus MFE | -18.17 |
| Energy contribution | -17.36 |
| Covariance contribution | -0.81 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.812873 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9980290 96 - 20766785 GACGGUUACUUAACUUAUGGAGUAUGCAAAGUUGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUU ((((((((((((((((.((.......)))))))))))..(((((((((((((......)))))))))))))........))))))).......... ( -22.50) >DroSim_CAF1 38760 96 - 1 GUCGGUUACUUAACUUAUGGAGUAUGCAAAGUUGAGUUUAUCGUGUAUGUAUUUAUACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUU ..((((((((((((((.((.......)))))))))))..(((((((((((((......)))))))))))))........)))))............ ( -20.10) >DroEre_CAF1 37651 95 - 1 GACGGUUACUUAACUUAUGGAGUAUGCAAAGUUGGGUUUAUCGUGUAUGUAA-UUAUCGUAUGUAUAUGAUUAAUGCUAAACUGUUUUAAUUGCUU ((((((((((((((((.((.......)))))))))))..((((((((((((.-......))))))))))))........))))))).......... ( -20.00) >DroYak_CAF1 39669 96 - 1 GACGAUUACUUAACCUAUGGAGUAUGCAAAGUUGGGUUUAUCGUGUACGUAAAUAAUCGUAUGUAUGUGAUUAAUACUAAACUGUUUUAAUUGCUU .(((((.(((((((...((.......))..)))))))..)))))(((((((........)))))))(..(((((.((......)).)))))..).. ( -20.20) >consensus GACGGUUACUUAACUUAUGGAGUAUGCAAAGUUGAGUUUAUCGUGUAUGUAAUUAAACGUAUGUAUAUGAUUAAUACUAAACUGUUUUAAUUGCUU ......(((((........))))).((((((((.(((..((((((((((((........))))))))))))....))).)))).......)))).. (-18.17 = -17.36 + -0.81)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:41:56 2006