
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,673,942 – 9,674,095 |
| Length | 153 |
| Max. P | 0.626333 |

| Location | 9,673,942 – 9,674,061 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.46 |
| Mean single sequence MFE | -37.41 |
| Consensus MFE | -20.63 |
| Energy contribution | -20.88 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.557148 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9673942 119 + 20766785 UCAUCAUGUGCGCCUUUACCAUGGGUUCGGCCAUCGUACAGUUCAUCAACAUCUAUAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGGCAUAUGGUGGUGGGUUU-GCAGAGGUUU (((((..((((((((......(((...(((((((((....((((.............))))....)))))))))))).))))))))((((......)))))))))....-.......... ( -38.22) >DroVir_CAF1 161 114 + 1 UUAUUAUGUGCGCCUUUACCAUGGGCGCAGCCAUUGUGCAAUUCAUAAACAUUUAUCGCACAAAUUGGUGGAUGCCACAGUUCCAAACGCGGCACAUGGUGGUAAGU----GUAUG-UU- .....((((((((..(((((((((.(((.((((((((((..................))))))..))))(((.........)))....))).).))))))))...))----)))))-).- ( -36.27) >DroPse_CAF1 170 120 + 1 UGAUAAUGUGUGCCUUUACUAUGGGAGCAGCCAUUGUGCAGUUCAUAAACAUAUAUCGCACAAACUGGUGGCUGUCCCAGGCUCACACAAGGCACAUGGUGGUGGGUUUCGCCAAAGUUU .......(((((((((.....(((((.((((((((((((.((............)).))))).....)))))))))))).........)))))))))(((((......)))))....... ( -41.94) >DroGri_CAF1 159 117 + 1 UGAUAAUGUGCGCCUUUACAAUGGGUGCGGCAAUUGUGCAAUUCAUCAAUAUAUAUCGCACCAACUGGUGGCUGCCGCAAUUCCAGACAAGGCACAUGGUGGUAAGCUU-GGCAAA-UU- ........((((((((.....(((((((((((((((((.....)).))))........((((....))))..)))))))..))))...))))).((.(((.....))))-))))..-..- ( -32.60) >DroAna_CAF1 167 119 + 1 UGAUCAUGUGCGCCUUUACCAUGGGUUCAGCUAUAGUACAGUUUAUAAAUAUCUACAGAACCAAUUGGUGGCUCCCACAGGCUCACACCAGGCACAUGGUGGUAGGCAG-GCUCAGAAUA ...((.((.(((((((((((((((((((.((....))...((..((....))..)).)))))..(((((((..((....))..).))))))...)))))))).))))..-)).))))... ( -33.50) >DroPer_CAF1 170 120 + 1 UGAUAAUGUGUGCCUUUACUAUGGGAGCAGCCAUUGUGCAGUUCAUAAACAUAUAUCGCACAAACUGGUGGCUGUCCCAGGCUCACACAAGGCACAUGGUGGUGGGUUUCGCCAAAGUUU .......(((((((((.....(((((.((((((((((((.((............)).))))).....)))))))))))).........)))))))))(((((......)))))....... ( -41.94) >consensus UGAUAAUGUGCGCCUUUACCAUGGGUGCAGCCAUUGUGCAGUUCAUAAACAUAUAUCGCACAAACUGGUGGCUGCCACAGGCUCACACAAGGCACAUGGUGGUAGGUUU_GCCAAAGUUU ...........((((((((((((...(((((((((((((.((............)).)))).....))))))))).....(((.......))).)))))))).))))............. (-20.63 = -20.88 + 0.26)
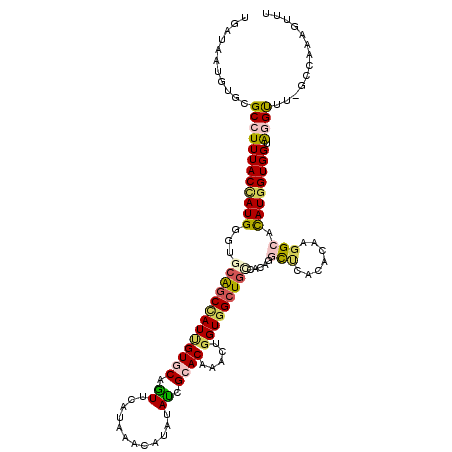
| Location | 9,673,982 – 9,674,095 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.52 |
| Mean single sequence MFE | -32.17 |
| Consensus MFE | -20.77 |
| Energy contribution | -21.22 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.626333 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9673982 113 + 20766785 GUUCAUCAACAUCUAUAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGGCAUAUGGUGGUGGGUUU-GCAGAGGUUUUCCAUAUGUAUCAACAAGGACAUUAAUCCAUAAU--- ((((......((((((...((((.((((((((.(((...))))).)))))).....)))).)))))).(-(((..((....))...)))).......)))).............--- ( -30.90) >DroSec_CAF1 210 113 + 1 GUUCAUCAACAUCUACAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGGCACAUGGUGGUGGGUUU-GCAGAGGUUUUCCAUCUGUAUCAACAAGGAAACUAAUCCGUAAU--- (((.......((((((...((((.((((((((.(((...))))).)))))).....)))).)))))).(-(((((((....)).))))))..)))..(((......))).....--- ( -35.70) >DroSim_CAF1 240 113 + 1 GUUCAUCAACAUCUACAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGGCACAUGGUGGUGGGUUU-GCAGAGGUUUCUCAUCUGUAUCAAAAAGGAAACUAAUCCAUAAU--- ...(((((...((....)).....((((((((.(((...))))).)))))).....)))))((((((((-(((((.(.....).))))))......((....)))))))))...--- ( -33.70) >DroEre_CAF1 213 113 + 1 GUUCAUCAACAUCUACAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGACACAUGGUGGUGGGUUU-GGAGAGGUUCCCCAACUUAAUCAUGAAGGACACUAAUCUAUUAA--- ((((.(((...(((...(((((..((((((((.(((...))))).)))))).(((......))))))))-..)))((....))..........))).)))).............--- ( -33.10) >DroYak_CAF1 210 112 + 1 GUUCAUCAACAUCUACAGAACCAACUGGUGGCUGCCUCAGGCACACACCAGGCACAUGGUGGUGGGUUU-GGCGAGGU-CCCCAACUUCUUUAUUAAGAACCCUAAUCUAUCAA--- ((((.((((.((((((...((((.((((((..((((...))))..)))))).....)))).))))))))-)).(((((-.....)))))........)))).............--- ( -34.00) >DroPer_CAF1 210 110 + 1 GUUCAUAAACAUAUAUCGCACAAACUGGUGGCUGUCCCAGGCUCACACAAGGCACAUGGUGGUGGGUUUCGCCAAAGUUUUACAUGCAUA-------GAAUAGAUGUCUAUAGAACC (((((((.((((.(((.(((.(((((..((((...((((...((((.((.......))))))))))....)))).)))))....))))))-------......)))).))).)))). ( -25.60) >consensus GUUCAUCAACAUCUACAGAACCAACUGGUGGCUGCCACAGGCGCACACCAGGCACAUGGUGGUGGGUUU_GCAGAGGUUUCCCAUCUGUAUCAACAAGGACACUAAUCCAUAAA___ ((((......((((((...((((.((((((..((((...))))..)))))).....)))).))))))........((....))..............))))................ (-20.77 = -21.22 + 0.45)

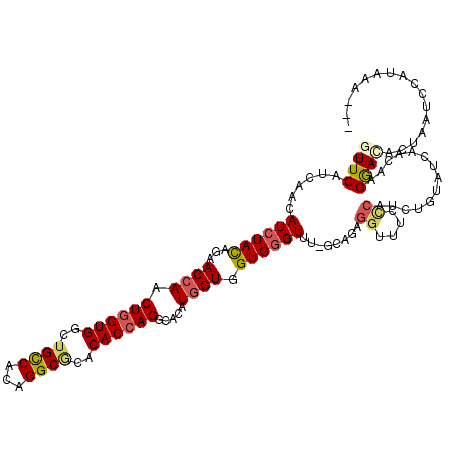
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:45 2006