
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,607,970 – 9,608,157 |
| Length | 187 |
| Max. P | 0.952877 |

| Location | 9,607,970 – 9,608,090 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.39 |
| Mean single sequence MFE | -53.67 |
| Consensus MFE | -48.12 |
| Energy contribution | -47.77 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.888512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9607970 120 - 20766785 AGAACCCGGCCAAUACGGACGCCAUGUCCGGGUCGGGUGCGUUUGACGAUGAGCCGCCGCCGGAGCCGCGGGACGGAUGGCUACUGGUGCGCAUCCAUGUGCCGGAGCUGAAUGUCUACA .(.((((((((....(((((.....))))))))))))).)((..((((.(.(((.((((((((((((((......).))))).)))))).)).(((.......)))))).).)))).)). ( -51.50) >DroVir_CAF1 26744 120 - 1 AGAACCCGGCCAAUAUGGACAGCACAUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCAGAACCCCGGGAUGGCUGGCUACUGGUGCGCAUCCAUGUGCCGGAGCUGAAUGUCUACA .(.((((((((...(((.......)))...)))))))).)((..((((.(.(((.(((((((...(....)..)))).)))..((((..((......))..)))).))).).)))).)). ( -50.40) >DroGri_CAF1 28640 120 - 1 AGAACCCGGCCAACAUGGACAACACGUCCGGGCCGGGUGCAUUUGACGAUGAGCCGCCGCCAGAGCCCCGAGAUGGUUGGCUUCUGGUGCGCAUUCAUGUGCCCGAGCUGAAUGUCUACA .(.((((((((....(((((.....))))))))))))).)....((((.(.(((((..(((((((((((.....))..))).))))))(((((....))))).)).))).).)))).... ( -51.30) >DroWil_CAF1 23742 120 - 1 AGAACCCGGCCAAUAUGGACGCCACGUCCGGGCCGGGGGCGUUUGACGAUGAGCCACCGCCAGAGCCGAGAGAUGGUUGGCUACUGGUGCGCAUCCAUGUGCCAGAGCUGAAUGUCUACA .(..(((((((....((((((...)))))))))))))..)((..((((.(.((((...(((((((((((.......)))))).)))))(((((....)))))..).))).).)))).)). ( -53.70) >DroMoj_CAF1 28368 120 - 1 AGAACCCGGCCAACAUGGACAGCACGUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCGGAGCCACGGGAUGGCUGGCUACUGGUGCGCAUCCAUGUGCCGGAGCUGAAUGUCUACA .(.((((((((....(((((.....))))))))))))).)((..((((...(((((.(.(((......)))).)))))((((.((((..((......))..))))))))...)))).)). ( -53.60) >DroAna_CAF1 22204 120 - 1 AGAACCCGGCCAAUACGGACGCCAUGUCCGGGCCGGGGGCGUUUGACGAUGAGCCGCCACCGGAGCCCCGGGACGGAUGGCUGCUGGUGCGGAUCCACGUGCCGGAGCUGAACGUCUACA ....(((((((....(((((.....))))))))))))(((((((......))).)))).((((....))))((((..(((((.((((..((......))..)))))))))..)))).... ( -61.50) >consensus AGAACCCGGCCAAUAUGGACACCACGUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCAGAGCCCCGGGAUGGAUGGCUACUGGUGCGCAUCCAUGUGCCGGAGCUGAAUGUCUACA .(.((((((((....(((((.....))))))))))))).)((..((((.(.(((.(((((((...(.....).))).))))..((((..((......))..)))).))).).)))).)). (-48.12 = -47.77 + -0.36)

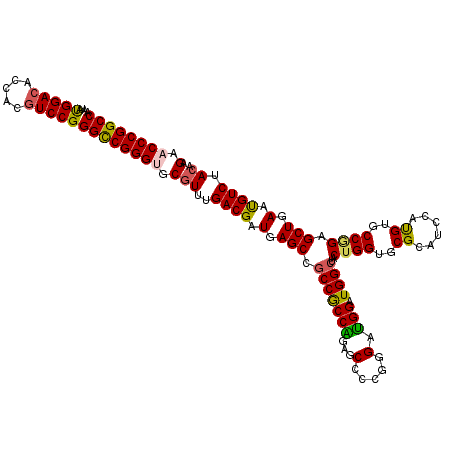

| Location | 9,608,010 – 9,608,130 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.50 |
| Mean single sequence MFE | -54.52 |
| Consensus MFE | -47.40 |
| Energy contribution | -46.90 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.952877 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9608010 120 + 20766785 CCAUCCGUCCCGCGGCUCCGGCGGCGGCUCAUCGUCAAACGCACCCGACCCGGACAUGGCGUCCGUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGGUAUGUCGUAAUUUU ((((((..(((((.((....)).)))(((((..((((((((.(((((.(((((((.....)))))....)).)))))..))))))))..)))))..))..)))))).............. ( -57.70) >DroVir_CAF1 26784 120 + 1 CCAGCCAUCCCGGGGUUCUGGCGGCGGCUCAUCGUCAAACGCACCCGGCCCGGAUGUGCUGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUU (((.(((((((((....)))).)...(((((..((((((((.((((((((..((((((.....))))))))))))))..))))))))..))))))))).))).................. ( -51.50) >DroGri_CAF1 28680 120 + 1 CCAACCAUCUCGGGGCUCUGGCGGCGGCUCAUCGUCAAAUGCACCCGGCCCGGACGUGUUGUCCAUGUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUU .((.((((((((..((....))..))(((((..((((((((.((((((((..((((((.....)))))))))))))).).)))))))..)))))..)))))).))............... ( -48.50) >DroWil_CAF1 23782 120 + 1 CCAACCAUCUCUCGGCUCUGGCGGUGGCUCAUCGUCAAACGCCCCCGGCCCGGACGUGGCGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUU .((.(((((((.(.((....)).).)(((((..((((((((..(((((((.(((((...))))).....)))))))..).)))))))..)))))..)))))).))............... ( -49.40) >DroMoj_CAF1 28408 120 + 1 CCAGCCAUCCCGUGGCUCCGGCGGCGGCUCAUCGUCAAACGCACCCGGCCCGGACGUGCUGUCCAUGUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUU .((.(((((((((.((....)).)))(((((..((((((((.((((((((..((((((.....))))))))))))))..))))))))..)))))..)))))).))............... ( -59.30) >DroAna_CAF1 22244 120 + 1 CCAUCCGUCCCGGGGCUCCGGUGGCGGCUCAUCGUCAAACGCCCCCGGCCCGGACAUGGCGUCCGUAUUGGCCGGGUUCUGUUUGACCUUGGGCGUGGGUGGGUGGUACGUCGUAAUUUU (((((((((((((....)))).))).(((((..((((((((..((((((((((((.....)))))....)))))))..).)))))))..)))))..)))))).................. ( -60.70) >consensus CCAACCAUCCCGGGGCUCCGGCGGCGGCUCAUCGUCAAACGCACCCGGCCCGGACGUGGCGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUU .((.(((((((...(((.....))).(((((..((((((((.((((((((.((((.....)))).....)))))))).).)))))))..)))))).)))))).))............... (-47.40 = -46.90 + -0.50)


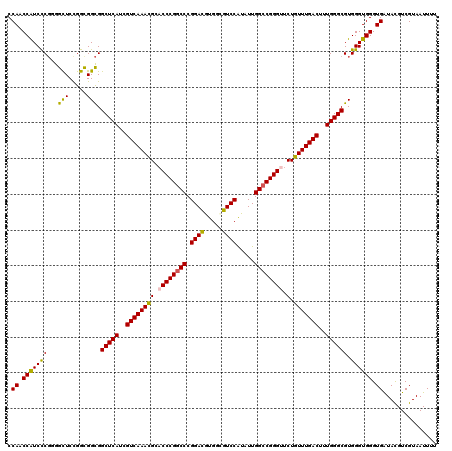
| Location | 9,608,010 – 9,608,130 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.50 |
| Mean single sequence MFE | -45.50 |
| Consensus MFE | -39.25 |
| Energy contribution | -38.95 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.888243 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9608010 120 - 20766785 AAAAUUACGACAUACCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUACGGACGCCAUGUCCGGGUCGGGUGCGUUUGACGAUGAGCCGCCGCCGGAGCCGCGGGACGGAUGG ..............(((.((.(((..((.((..(((((((...((((((((....(((((.....)))))))))))))..)))))))..)).)).((.((....)).)))))..)).))) ( -48.50) >DroVir_CAF1 26784 120 - 1 AAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACAGCACAUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCAGAACCCCGGGAUGGCUGG ..................(((.(((..(((...(((((((...((((((((...(((.......)))...))))))))..)))))))..((.((....)))).......))).))).))) ( -42.50) >DroGri_CAF1 28680 120 - 1 AAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAACAUGGACAACACGUCCGGGCCGGGUGCAUUUGACGAUGAGCCGCCGCCAGAGCCCCGAGAUGGUUGG .......((((.((((..........((.((..((((((....((((((((....(((((.....)))))))))))))...))))))..)).))((..((....))..)).)))))))). ( -41.20) >DroWil_CAF1 23782 120 - 1 AAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACGCCACGUCCGGGCCGGGGGCGUUUGACGAUGAGCCACCGCCAGAGCCGAGAGAUGGUUGG .......((((.((((..........((.((..(((((((.(..(((((((....((((((...)))))))))))))..))))))))..)).))..(.((....)).)...)))))))). ( -44.20) >DroMoj_CAF1 28408 120 - 1 AAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAACAUGGACAGCACGUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCGGAGCCACGGGAUGGCUGG ..................(((.(((..(((...(((((((...((((((((....(((((.....)))))))))))))..)))))))...(..(((....)))..)...))).))).))) ( -47.50) >DroAna_CAF1 22244 120 - 1 AAAAUUACGACGUACCACCCACCCACGCCCAAGGUCAAACAGAACCCGGCCAAUACGGACGCCAUGUCCGGGCCGGGGGCGUUUGACGAUGAGCCGCCACCGGAGCCCCGGGACGGAUGG ..............(((.((......((.((..(((((((.(..(((((((....(((((.....))))))))))))..))))))))..)).)).....((((....))))...)).))) ( -49.10) >consensus AAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACACCACGUCCGGGCCGGGUGCGUUUGACGAUGAGCCGCCGCCAGAGCCCCGGGAUGGAUGG ..........................((.((..(((((((...((((((((....(((((.....)))))))))))))..)))))))..)).))..((((((...(.....).))).))) (-39.25 = -38.95 + -0.30)

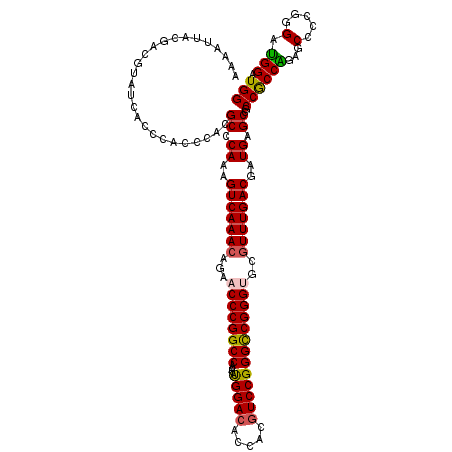
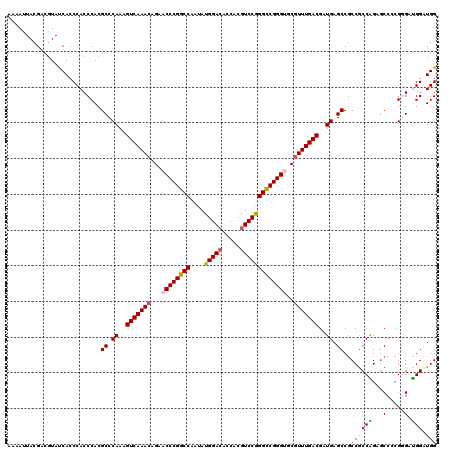
| Location | 9,608,050 – 9,608,157 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 81.40 |
| Mean single sequence MFE | -41.73 |
| Consensus MFE | -31.97 |
| Energy contribution | -31.92 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.547820 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9608050 107 + 20766785 GCACCCGACCCGGACAUGGCGUCCGUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGGUAUGUCGUAAUUUUGACGGCUUUCUAU-C--GC---------ACGGUCCACGA (.(((((.(((((((.....)))))....)).))))).)..........((((((((((((((.(((.(((((......)))))))).)))))-)--.)---------)).)))))... ( -39.00) >DroVir_CAF1 26824 114 + 1 GCACCCGGCCCGGAUGUGCUGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUUGAAGGCUUUUCG--C--GCA-CAGCGUCACAGUUUACAA (.((((((((..((((((.....)))))))))))))).)(((..((((...(((((..(((.((((...(((...........)))...)))--)--.))-).)))))..)))).))). ( -43.30) >DroGri_CAF1 28720 112 + 1 GCACCCGGCCCGGACGUGUUGUCCAUGUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUUGACGGCUUUUUC--C--GCGUCAGCGUCACAGUG---AA ..((((((((..((((((.....)))))))))))))).((((..(((....((((((((.((((....(((((......)))))))))..))--)--)))))...)))))))..---.. ( -45.90) >DroWil_CAF1 23822 109 + 1 GCCCCCGGCCCGGACGUGGCGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUUGACGGCUUUAAUU-UAAAC---------CGGGUACACAA ((((((.(((((((((...))))).....(.(((((.((.....)).))))).)..)))).)).....(((((......))))).........-.....---------.))))...... ( -36.30) >DroMoj_CAF1 28448 111 + 1 GCACCCGGCCCGGACGUGCUGUCCAUGUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUUGACGGCUUUUCG--C--GUA-CAGCGUCACAGUG---AA (.((((((((..((((((.....)))))))))))))).)...((.(((...(((((..(((.((((..(((((......))))).....)))--)--.))-).)))))..))).---)) ( -44.60) >DroAna_CAF1 22284 106 + 1 GCCCCCGGCCCGGACAUGGCGUCCGUAUUGGCCGGGUUCUGUUUGACCUUGGGCGUGGGUGGGUGGUACGUCGUAAUUUUGACGGCUUUUUAUAC--GC---------AC--UUUAUGG ((((.((((((((((.....)))))....)))))((((......))))..))))(((.(((...(((.(((((......)))))))).....)))--.)---------))--....... ( -41.30) >consensus GCACCCGGCCCGGACGUGGCGUCCAUAUUGGCCGGGUUCUGUUUGACUUUGGGCGUGGGUGGGUGAUACGUCGUAAUUUUGACGGCUUUUUA__C__GC_________ACAGUG_ACAA .(((((.((((((((.....)))).....(.(((((.((.....)).))))).)..)))))))))...(((((......)))))................................... (-31.97 = -31.92 + -0.05)



| Location | 9,608,050 – 9,608,157 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.40 |
| Mean single sequence MFE | -33.88 |
| Consensus MFE | -23.08 |
| Energy contribution | -23.22 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.786224 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9608050 107 - 20766785 UCGUGGACCGU---------GC--G-AUAGAAAGCCGUCAAAAUUACGACAUACCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUACGGACGCCAUGUCCGGGUCGGGUGC .(((((...(.---------((--.-.......)))(((........)))...........)))))................((((((((....(((((.....))))))))))))).. ( -32.50) >DroVir_CAF1 26824 114 - 1 UUGUAAACUGUGACGCUG-UGC--G--CGAAAAGCCUUCAAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACAGCACAUCCGGGCCGGGUGC .......(((((((..((-.((--(--.(((.....)))........((....)).........))).))..)))..)))).((((((((...(((.......)))...)))))))).. ( -31.00) >DroGri_CAF1 28720 112 - 1 UU---CACUGUGACGCUGACGC--G--GAAAAAGCCGUCAAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAACAUGGACAACACGUCCGGGCCGGGUGC ..---..(((((((..((.((.--(--(.......((((........))))..........)).))..))..)))..)))).((((((((....(((((.....))))))))))))).. ( -36.03) >DroWil_CAF1 23822 109 - 1 UUGUGUACCCG---------GUUUA-AAUUAAAGCCGUCAAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACGCCACGUCCGGGCCGGGGGC ..((((((..(---------((((.-.....)))))(((........)))))).)))........((((....((.....))..((((((....((((((...)))))))))))))))) ( -35.80) >DroMoj_CAF1 28448 111 - 1 UU---CACUGUGACGCUG-UAC--G--CGAAAAGCCGUCAAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAACAUGGACAGCACGUCCGGGCCGGGUGC ..---..(((((((..((-..(--(--(.....))((((........))))..............)..))..)))..)))).((((((((....(((((.....))))))))))))).. ( -35.60) >DroAna_CAF1 22284 106 - 1 CCAUAAA--GU---------GC--GUAUAAAAAGCCGUCAAAAUUACGACGUACCACCCACCCACGCCCAAGGUCAAACAGAACCCGGCCAAUACGGACGCCAUGUCCGGGCCGGGGGC .......--((---------(.--((.........((((........))))........)).)))((((..(......).....((((((....(((((.....))))))))))))))) ( -32.33) >consensus UUGU_AACUGU_________GC__G__CAAAAAGCCGUCAAAAUUACGACGUAUCACCCACCCACGCCCAAAGUCAAACAGAACCCGGCCAAUAUGGACACCACGUCCGGGCCGGGUGC ...................................((((........))))................................(((((((....(((((.....))))))))))))... (-23.08 = -23.22 + 0.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:33 2006