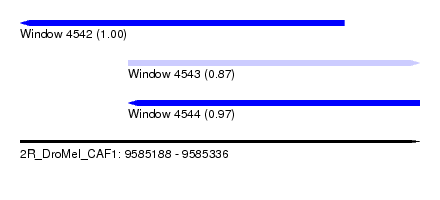
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,585,188 – 9,585,336 |
| Length | 148 |
| Max. P | 0.995257 |

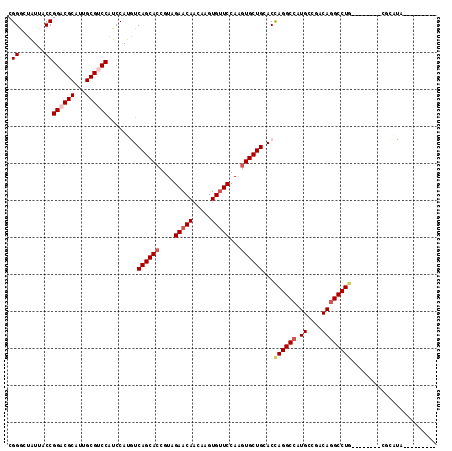
| Location | 9,585,188 – 9,585,308 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.33 |
| Mean single sequence MFE | -47.53 |
| Consensus MFE | -42.69 |
| Energy contribution | -42.38 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.56 |
| SVM RNA-class probability | 0.995257 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9585188 120 - 20766785 AUGGCCUGGUCCAGCACCUGGAACACCUGCUGUUCUACGGUGCUGAUAUGGACGGCCGCAAUGCGUCCGGCAAUAGUCCGCUGCAUGUGUGCGCUGUUAACAACCAAGAGGCUUGUGCUA ..((((((((.((((((((((((((.....))))))).))))))).....(((((((((((((((..((((....).))).))))).)))).))))))....)))...)))))....... ( -48.10) >DroVir_CAF1 336 120 - 1 AUGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCGUUGCAUGUUUGCGCUGUUAACAACCAAGAGGCUUGUGCUA ..(((((.(((((((((((((((((.....))))))).))))))).)))((..((((((...))))))(.(((((((.....(((....)))))))))).)..))...)))))....... ( -48.10) >DroPse_CAF1 315 120 - 1 ACGGCCUGGUUCAGCACUUGGAACACCUCCUGUUCUACGGUGCUGACAUGGAUGGCCGCAAUGCGUCCGGCAAUAGUCCGCUGCAUGUGUGCGCCGUCAACAACCAAGAGGCUUGUGCCC (((((((((((((((((((((((((.....))))))).))))))))....(((((((((((((((..((((....).))).))))).)))).))))))....)))...)))).))).... ( -49.40) >DroGri_CAF1 358 120 - 1 ACGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCGUUGCAUGUUUGUGCUGUUAACAACCAAGAGGCUUGUGCUA (((((((.(((((((((((((((((.....))))))).))))))).)))((..((((((...))))))(.(((((((.((.........)).))))))).)..))...)))).))).... ( -46.90) >DroWil_CAF1 303 120 - 1 AUGGCCUGGUUCAGCAUCUGGAACACUUGUUGUUCUACGGUGCUGAUAUGGAUGGACGCAAUGCGUCCGGUAAUACCCCGUUGCAUGUUUGCGCUGUUAACAACCAAGAGGCUUGUGCUA ..(((((.((((........)))).(((((((((..(((((((.((((((...((((((...))))))((.....))......)))))).))))))).))))).)))))))))....... ( -44.20) >DroMoj_CAF1 352 120 - 1 ACGGCCUAGUGCAGCACUUGGAGCACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCGUUGCAUGUUUGCGCUGUUAACAACCAAGAGGCUUGUGCUA ...((.....))(((((...((((.(((((((((..(((((((.((((((...((((((...))))))(((....))).....)))))).))))))).))))).))))..))))))))). ( -48.50) >consensus ACGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCGUUGCAUGUUUGCGCUGUUAACAACCAAGAGGCUUGUGCUA ..(((((.((.((((((((((((((.....))))))).))))))))).(((.(((((((...))))))).(((((((.....(((....))))))))))....)))..)))))....... (-42.69 = -42.38 + -0.30)

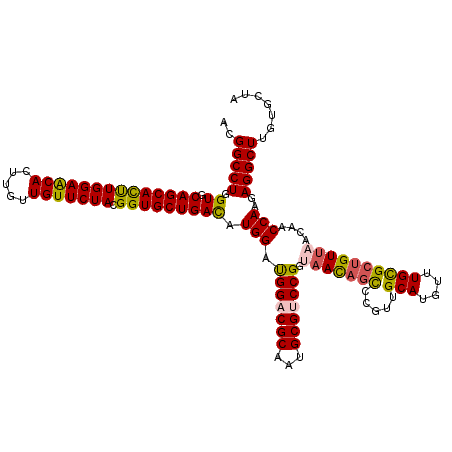
| Location | 9,585,228 – 9,585,336 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 80.20 |
| Mean single sequence MFE | -37.87 |
| Consensus MFE | -29.31 |
| Energy contribution | -30.08 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.872780 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
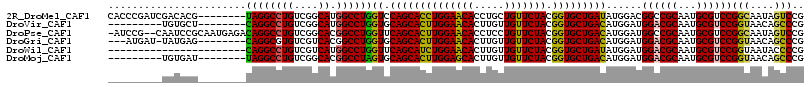
>2R_DroMel_CAF1 9585228 108 + 20766785 CGGACUAUUGCCGGACGCAUUGCGGCCGUCCAUAUCAGCACCGUAGAACAGCAGGUGUUCCAGGUGCUGGACCAGGCCAUGCCGACAGGCCUA--------CGUGUCGAUCGGGUG .........(((.(((((((...(((((((....((((((((...(((((.....)))))..))))))))....(((...)))))).))))..--------.))).)).)).))). ( -42.10) >DroVir_CAF1 376 99 + 1 CGGGCUGUUACCGGACGCAUUGCGUCCAUCCAUGUCAGCACCGUAGAACAACAAGUGUUCCAAGUGCUGCACCAGGCCAUGCCGACAGGCCUG--------AGCACA--------- (((((((.....((((((...))))))........)))).)))..(((((.....)))))...(((((....((((((.((....))))))))--------))))).--------- ( -38.82) >DroPse_CAF1 355 113 + 1 CGGACUAUUGCCGGACGCAUUGCGGCCAUCCAUGUCAGCACCGUAGAACAGGAGGUGUUCCAAGUGCUGAACCAGGCCGUGCCGACAGGCCUGUCUCAUUGCGGAUUG--CGGAU- (((.(....))))..((((..((((((.......(((((((....(((((.....)))))...)))))))....))))))(((....)))((((......))))..))--))...- ( -39.50) >DroGri_CAF1 398 104 + 1 CGGGCUGUUACCGGACGCAUUGCGUCCAUCCAUGUCAGCACCGUAGAACAACAAGUGUUCCAAGUGCUGCACCAGGCCGUGACGACACGCCUG--------CUCAUA-AUCAU--- .((((.......((((((...)))))).....((.((((((....(((((.....)))))...))))))))..((((.(((....))))))))--------)))...-.....--- ( -36.40) >DroWil_CAF1 343 93 + 1 CGGGGUAUUACCGGACGCAUUGCGUCCAUCCAUAUCAGCACCGUAGAACAACAAGUGUUCCAGAUGCUGAACCAGGCCAUGACGACAGGCCUG----------------------- .((((.....))((((((...))))))..))...((((((.(...(((((.....)))))..).))))))..((((((.((....))))))))----------------------- ( -36.50) >DroMoj_CAF1 392 99 + 1 CGGGCUGUUACCGGACGCAUUGCGUCCAUCCAUGUCAGCACCGUAGAACAACAAGUGCUCCAAGUGCUGCACUAGGCCGUGCCGACAGGCCUA--------AUCACA--------- .(((((......((((((...)))))).....((((.((((.((......)).(((((..(....)..))))).....)))).))))))))).--------......--------- ( -33.90) >consensus CGGGCUAUUACCGGACGCAUUGCGUCCAUCCAUGUCAGCACCGUAGAACAACAAGUGUUCCAAGUGCUGCACCAGGCCAUGCCGACAGGCCUG________CGCAUA_________ .((.......))((((((...))))))........((((((....(((((.....)))))...))))))...((((((.((....))))))))....................... (-29.31 = -30.08 + 0.78)

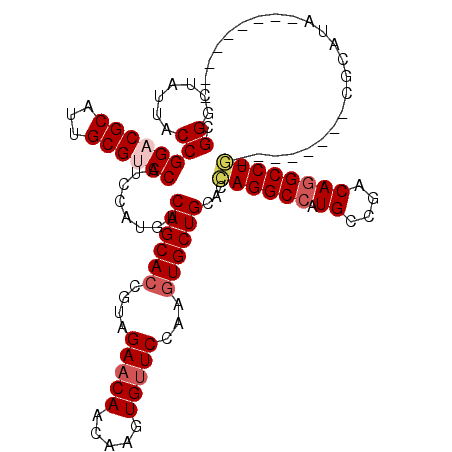
| Location | 9,585,228 – 9,585,336 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 80.20 |
| Mean single sequence MFE | -43.42 |
| Consensus MFE | -39.32 |
| Energy contribution | -38.52 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.970132 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9585228 108 - 20766785 CACCCGAUCGACACG--------UAGGCCUGUCGGCAUGGCCUGGUCCAGCACCUGGAACACCUGCUGUUCUACGGUGCUGAUAUGGACGGCCGCAAUGCGUCCGGCAAUAGUCCG .........(((...--------((((((((....)).)))))))))((((((((((((((.....))))))).)))))))....((((.((((.........))))....)))). ( -44.20) >DroVir_CAF1 376 99 - 1 ---------UGUGCU--------CAGGCCUGUCGGCAUGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCG ---------.(.(((--------((((((((....)).))))))(((((((((((((((((.....))))))).))))))).)))...(((((((...))))))).....))).). ( -44.00) >DroPse_CAF1 355 113 - 1 -AUCCG--CAAUCCGCAAUGAGACAGGCCUGUCGGCACGGCCUGGUUCAGCACUUGGAACACCUCCUGUUCUACGGUGCUGACAUGGAUGGCCGCAAUGCGUCCGGCAAUAGUCCG -....(--(.....)).(((...((((((((....)).))))))..(((((((((((((((.....))))))).)))))))))))((((.((((.........))))....)))). ( -44.10) >DroGri_CAF1 398 104 - 1 ---AUGAU-UAUGAG--------CAGGCGUGUCGUCACGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCG ---.....-......--------((((((((....))).)))))(((((((((((((((((.....))))))).))))))).)))....((((((...))))))(((....))).. ( -42.50) >DroWil_CAF1 343 93 - 1 -----------------------CAGGCCUGUCGUCAUGGCCUGGUUCAGCAUCUGGAACACUUGUUGUUCUACGGUGCUGAUAUGGAUGGACGCAAUGCGUCCGGUAAUACCCCG -----------------------((((((((....)).))))))..(((((((((((((((.....))))))).))))))))...((..((((((...))))))((.....)))). ( -42.90) >DroMoj_CAF1 392 99 - 1 ---------UGUGAU--------UAGGCCUGUCGGCACGGCCUAGUGCAGCACUUGGAGCACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCG ---------.(((((--------((((((((....)).)))))))).((((((((((((((.....))))))).))))))).)))....((((((...))))))(((....))).. ( -42.80) >consensus _________UAUGCG________CAGGCCUGUCGGCACGGCCUGGUGCAGCACUUGGAACACUUGUUGUUCUACGGUGCUGACAUGGAUGGACGCAAUGCGUCCGGUAACAGCCCG .......................((((((((....)).))))))((.((((((((((((((.....))))))).)))))))))......((((((...))))))(((....))).. (-39.32 = -38.52 + -0.80)
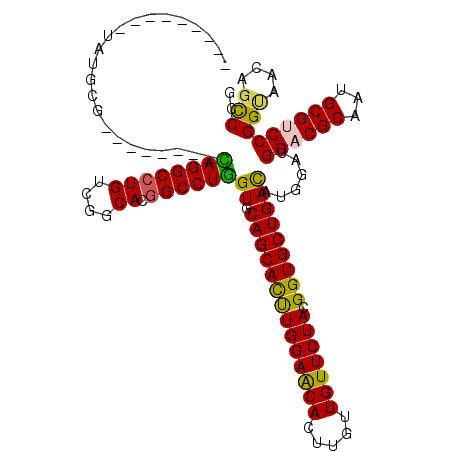
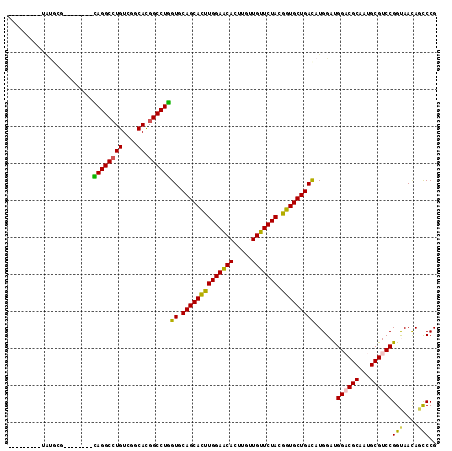
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:19 2006