
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,582,264 – 9,582,424 |
| Length | 160 |
| Max. P | 0.740502 |

| Location | 9,582,264 – 9,582,384 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -49.73 |
| Consensus MFE | -49.89 |
| Energy contribution | -49.23 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.38 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581556 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
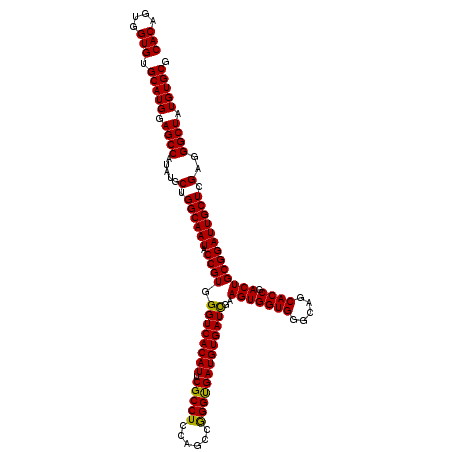
>2R_DroMel_CAF1 9582264 120 - 20766785 CACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCAGGUGAUGUGAUCGAAGUGGUGGGCAGCACCGACUGCGGAUUGCUCGAGGGCUAUGUGCG (((....))).(((((.((((.....(.((((((.((((.((((((((.(((((......)))))))))))))..(((((((.....)))).))))))))))))).)..)))).))))). ( -49.30) >DroSim_CAF1 5847 120 - 1 CACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUUCAGCCGGGUGAUGUGAUCGAAGUGGUGGGCAGCACCGACUGCGGAUUGCUCGAGGGCUAUGUGCG (((....))).(((((.((((.....(.((((((.((((.((((((((.(((((......)))))))))))))..(((((((.....)))).))))))))))))).)..)))).))))). ( -49.30) >DroYak_CAF1 6795 120 - 1 CACAGUGGUGUGCAUGGAGCCAUACGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCGGGCGAUGUGAUUGAAGUGGUGGGCAGCACCGACUGCGGAUUGCUCGAGGGCUAUGUGCG (((....))).(((((.((((...((..((((((.(((..((((((((.(((((......)))))))))))).....(((((.....))))).)..)))))))))))..)))).))))). ( -50.60) >consensus CACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCGGGUGAUGUGAUCGAAGUGGUGGGCAGCACCGACUGCGGAUUGCUCGAGGGCUAUGUGCG (((....))).(((((.((((.....(.((((((.((((.((((((((.(((((......)))))))))))))..(((((((.....)))).))))))))))))).)..)))).))))). (-49.89 = -49.23 + -0.66)

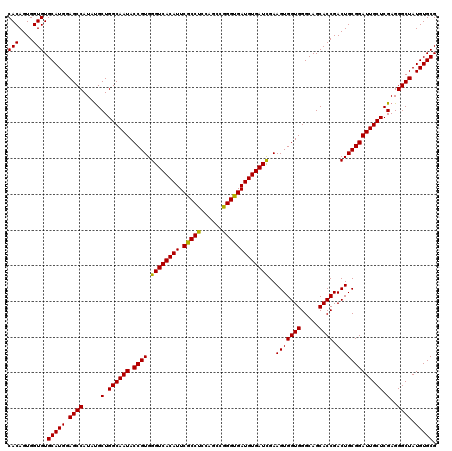
| Location | 9,582,304 – 9,582,424 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -48.20 |
| Consensus MFE | -46.88 |
| Energy contribution | -46.67 |
| Covariance contribution | -0.21 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.740502 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9582304 120 - 20766785 ACUCGGAUUCGGCAGCAUGUUCCAUCGGGCAUCCCAGCACCACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCAGGUGAUGUGAUCGAAGUGG .(.((((((.(.(((((((((((((((((...))).(((((.....)))))).)))))))...)))))).)))).))).)((((((((.(((((......)))))))))))))....... ( -50.40) >DroSim_CAF1 5887 120 - 1 ACUCGGAUUCGGCAGCAUGUUCCAUCGGCCAUCCCAGCACCACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUUCAGCCGGGUGAUGUGAUCGAAGUGG .(.((((((.(.((((((((((((((((.....)).(((((.....)))))).)))))))...)))))).)))).))).)((((((((.(((((......)))))))))))))....... ( -47.90) >DroYak_CAF1 6835 120 - 1 ACUCGGACUCGGCAGCCUGUUCCAUCGGGCAUCCUAGCACCACAGUGGUGUGCAUGGAGCCAUACGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCGGGCGAUGUGAUUGAAGUGG ..........(((.(((((......))))).(((..(((((.....)))))....))))))...((((((.....)).(.((((((((.(((((......))))))))))))).))))). ( -46.30) >consensus ACUCGGAUUCGGCAGCAUGUUCCAUCGGGCAUCCCAGCACCACAGUGGUGUGCAUGGAGCCAUAUGCUGGCAAUACCGUGGGUCACAUUCGCCUCCAGCCGGGUGAUGUGAUCGAAGUGG .(.((((((.(.((((((((((((((((.....)).(((((.....)))))).)))))))...)))))).)))).))).)((((((((.(((((......)))))))))))))....... (-46.88 = -46.67 + -0.21)


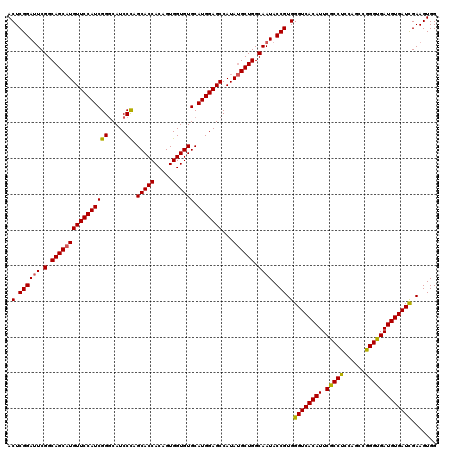
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:13 2006