
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,513,637 – 9,513,750 |
| Length | 113 |
| Max. P | 0.938273 |

| Location | 9,513,637 – 9,513,750 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.26 |
| Mean single sequence MFE | -23.96 |
| Consensus MFE | -21.86 |
| Energy contribution | -22.26 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.46 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.938040 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
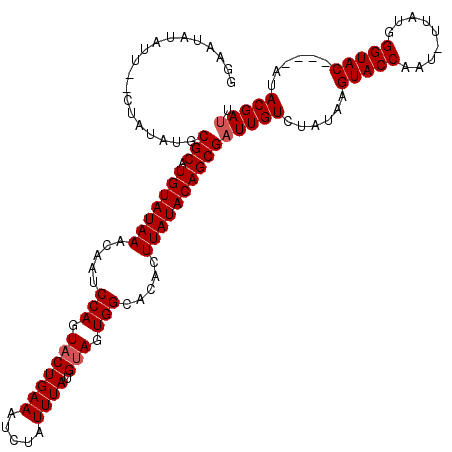
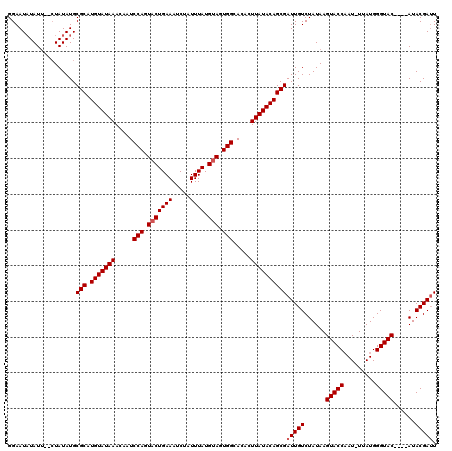
>2R_DroMel_CAF1 9513637 113 + 20766785 GGAAUAUAUU--CUAUAUGCGCUUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGUAGUGGCACACUUAUACAGCGAUUGUCUAUAAGUACCAAU-UUAUGGGUAC----AUACGAUU ((((....))--)).((((((((.((((((.....(((.(((((((.....)))).))).))).....))))))))))..(..((((((((....))-))))))..))----)))..... ( -23.70) >DroSec_CAF1 10689 116 + 1 GGAAUAUAUAUUCUAUAUGCGCAUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGCAGUGGAACACUUAUACAGCGAUUGUCUAUAAGUACCAAUUUUAUGGGUAC----AUACGAAU (((((....))))).....(((.(((((((....((((.(.(((((.....)))).).).))))....))))))))))...........(((((.........)))))----........ ( -21.40) >DroSim_CAF1 10723 116 + 1 GGAAUAUAUUCUAUAUAUGCGCAUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGUAGUGGCACACUUAUACAGCGAUUGUCUAUAAGUACCUAUUUUAUGGGUAC----AUACGAUU (((((((((...)))))).(((.(((((((.....(((.(((((((.....)))).))).))).....))))))))))....)))....((((((((...))))))))----........ ( -26.30) >DroEre_CAF1 10892 113 + 1 GGAAUAUAUU--CUAUAUGCGCAUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGUAGUGGCACACUUAUACAGCGAUUGUCUAUAAGUACCAAU-UUAUGGGUAC----AUACGAUU ((((....))--)).(((((((.(((((((.....(((.(((((((.....)))).))).))).....))))))))))..(..((((((((....))-))))))..))----)))..... ( -23.70) >DroYak_CAF1 10795 117 + 1 GGAAUCUAUU--CUAUAUGCGCAUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGUAGUGGCACACUUAUACAGCGAUUGUCUAUAAGUACCAAU-UUAUGGGUACAUACAUACGAUU ((((....))--)).(((((((.(((((((.....(((.(((((((.....)))).))).))).....))))))))))...........(((((...-.....)))))...))))..... ( -24.70) >consensus GGAAUAUAUU__CUAUAUGCGCAUGUAUAAACAAUCCAGUACUGAAAUCUAUUUAUGUAGUGGCACACUUAUACAGCGAUUGUCUAUAAGUACCAAU_UUAUGGGUAC____AUACGAUU ...................(((.(((((((.....(((.(((((((.....)))).))).))).....))))))))))(((((......(((((.........)))))......))))). (-21.86 = -22.26 + 0.40)

| Location | 9,513,637 – 9,513,750 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.26 |
| Mean single sequence MFE | -23.61 |
| Consensus MFE | -21.24 |
| Energy contribution | -21.48 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938273 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
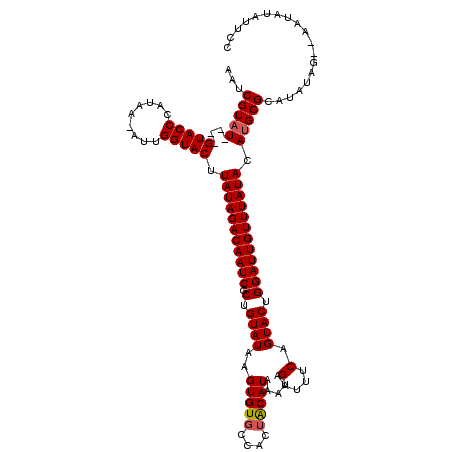
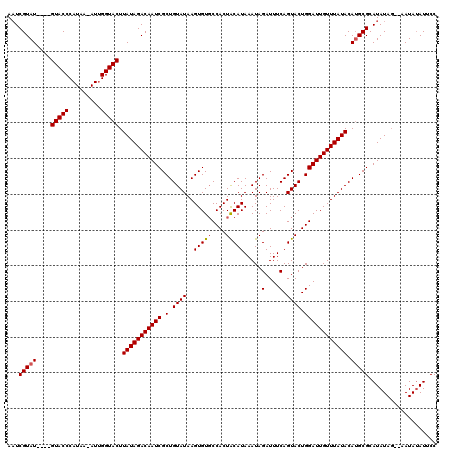
>2R_DroMel_CAF1 9513637 113 - 20766785 AAUCGUAU----GUACCCAUAA-AUUGGUACUUAUAGACAAUCGCUGUAUAAGUGUGCCACUACAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAAGCGCAUAUAG--AAUAUAUUCC ...(((..----(((((.....-...))))).(((((((((((.(.((((..(((((....))))).....(....).)))).))))))))))))...))).......--.......... ( -21.30) >DroSec_CAF1 10689 116 - 1 AUUCGUAU----GUACCCAUAAAAUUGGUACUUAUAGACAAUCGCUGUAUAAGUGUUCCACUGCAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAUGCGCAUAUAGAAUAUAUAUUCC ((((((((----((..(((......)))...............(((((((((..(.((((.(((..............))).)))).)..))))))).)))))))).))))......... ( -23.84) >DroSim_CAF1 10723 116 - 1 AAUCGUAU----GUACCCAUAAAAUAGGUACUUAUAGACAAUCGCUGUAUAAGUGUGCCACUACAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAUGCGCAUAUAUAGAAUAUAUUCC ...(((((----(((((.........))))).(((((((((((.(.((((..(((((....))))).....(....).)))).)))))))))))).)))))................... ( -22.80) >DroEre_CAF1 10892 113 - 1 AAUCGUAU----GUACCCAUAA-AUUGGUACUUAUAGACAAUCGCUGUAUAAGUGUGCCACUACAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAUGCGCAUAUAG--AAUAUAUUCC ...(((((----(((((.....-...))))).(((((((((((.(.((((..(((((....))))).....(....).)))).)))))))))))).))))).......--.......... ( -23.40) >DroYak_CAF1 10795 117 - 1 AAUCGUAUGUAUGUACCCAUAA-AUUGGUACUUAUAGACAAUCGCUGUAUAAGUGUGCCACUACAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAUGCGCAUAUAG--AAUAGAUUCC ...((((((((.(((((.....-...)))))...(((((((((.(.((((..(((((....))))).....(....).)))).)))))))))))))))))).......--.......... ( -26.70) >consensus AAUCGUAU____GUACCCAUAA_AUUGGUACUUAUAGACAAUCGCUGUAUAAGUGUGCCACUACAUAAAUAGAUUUCAGUACUGGAUUGUUUAUACAUGCGCAUAUAG__AAUAUAUUCC ...(((((....(((((.........))))).(((((((((((.(.((((..(((((....))))).....(....).)))).)))))))))))).)))))................... (-21.24 = -21.48 + 0.24)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:37:10 2006