
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,511,359 – 9,511,596 |
| Length | 237 |
| Max. P | 0.999842 |

| Location | 9,511,359 – 9,511,476 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.22 |
| Mean single sequence MFE | -28.53 |
| Consensus MFE | -27.04 |
| Energy contribution | -27.16 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511359 117 + 20766785 ---CGGGUGUGUUUGCUUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGUACGAUACAGAUGUGCCCAUAUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCUCUAGCUCUA ---.((((((....((((((((........))))))........................(((((((........))))))).))....))))))......((((((.......)))))) ( -29.10) >DroSec_CAF1 8403 116 + 1 ---CGGGUGUGUUUGCUUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGAACGAUACAGAUGUGACCAUGUACACAUCUAGCUAUAGCACUCUCUAUGUAGAGCCCCU-UAGCUCUA ---.(((.(((((.((((((((........))))))..((((.......)))).......(((((((........))))))).))...))))).)))....((((((....-..)))))) ( -30.10) >DroSim_CAF1 8423 119 + 1 GACCGGGUGUGUUUGCUUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCU-UAGCUCUA ....((((((....((((((((........))))))........................(((((((........))))))).))....))))))......((((((....-..)))))) ( -29.60) >DroEre_CAF1 8660 116 + 1 ---AGAGUGUGUUUGCGUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGUACGAUACAGAUGUGCCCAUAUACACAUCUAGCCAUAGCGCUCUUUAUGUAGAGCCCCU-UAGCUCUA ---(((((((....((.(((((........))))).........................(((((((........))))))).))....))))))).....((((((....-..)))))) ( -29.30) >DroYak_CAF1 8560 116 + 1 ---CGAGUGUGUUUGCUUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCCAUAGCGCUCUUUAUGUAGAUCCCCU-UAGCGCUA ---...(((..(((((((((((........))))))........)).)))..))).....(((((((........))))))).....(((((((.................-.))))))) ( -24.57) >consensus ___CGGGUGUGUUUGCUUUUGCCUUAAUCAGCAGAAAAUCAUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCU_UAGCUCUA ....((((((....((((((((........))))))........................(((((((........))))))).))....))))))......((((((.......)))))) (-27.04 = -27.16 + 0.12)

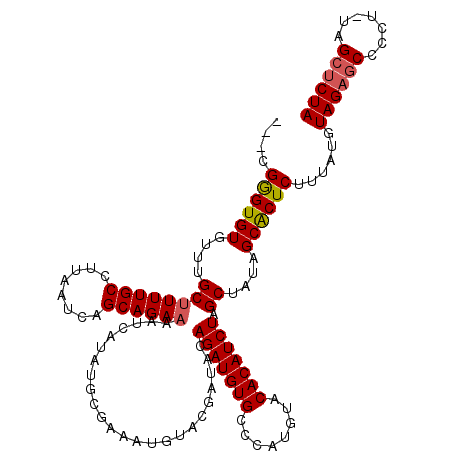
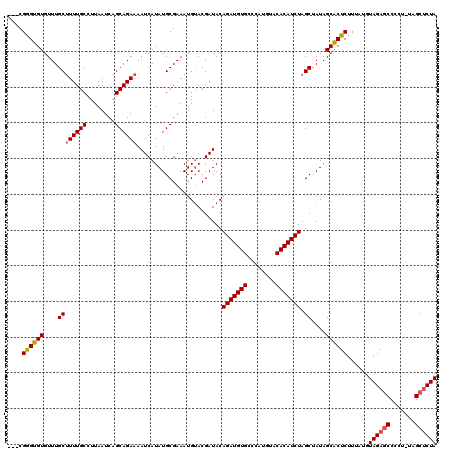
| Location | 9,511,396 – 9,511,516 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.15 |
| Mean single sequence MFE | -39.81 |
| Consensus MFE | -37.38 |
| Energy contribution | -37.98 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -4.97 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.90 |
| SVM RNA-class probability | 0.999696 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511396 120 + 20766785 AUAUGCGAAAUGUACGAUACAGAUGUGCCCAUAUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCUCUAGCUCUAGAGUCUAGACUCUAAAGUCUAGACCACAUAAACGCGUAUC (((((((.............(((((((........))))))).((....))....((((((((((((.......))))).(.(((((((((....)))))))))))))))))))))))). ( -39.40) >DroSec_CAF1 8440 119 + 1 AUAUGCGAAAUGAACGAUACAGAUGUGACCAUGUACACAUCUAGCUAUAGCACUCUCUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUC (((((((.............(((((((........))))))).((....))......((((((((((....-..))))).(.(((((((((....)))))))))))))))..))))))). ( -39.40) >DroSim_CAF1 8463 119 + 1 AUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUC (((((((.............(((((((........))))))).((....))....((((((((((((....-..))))).(.(((((((((....)))))))))))))))))))))))). ( -40.90) >DroEre_CAF1 8697 119 + 1 AUAUGCGAAAUGUACGAUACAGAUGUGCCCAUAUACACAUCUAGCCAUAGCGCUCUUUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUC ...............((((((((((((........))))))).......(((...((((((((((((....-..))))).(.(((((((((....))))))))))))))))))))))))) ( -40.90) >DroYak_CAF1 8597 119 + 1 AUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCCAUAGCGCUCUUUAUGUAGAUCCCCU-UAGCGCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUC (((((((..((((.......(((((((........))))))).....(((((((.................-.)))))))(.(((((((((....))))))))))))))...))))))). ( -38.47) >consensus AUAUGCGAAAUGUACGAUACAGAUGUGCCCAUGUACACAUCUAGCUAUAGCACUCUUUAUGUAGAGCCCCU_UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUC (((((((.............(((((((........))))))).((....))....((((((((((((.......))))).(.(((((((((....)))))))))))))))))))))))). (-37.38 = -37.98 + 0.60)



| Location | 9,511,396 – 9,511,516 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.15 |
| Mean single sequence MFE | -45.42 |
| Consensus MFE | -42.30 |
| Energy contribution | -42.50 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.05 |
| Mean z-score | -4.60 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511396 120 - 20766785 GAUACGCGUUUAUGUGGUCUAGACUUUAGAGUCUAGACUCUAGAGCUAGAGGGGCUCUACAUAAAGAGUGCUAUAGCUAGAUGUGUAUAUGGGCACAUCUGUAUCGUACAUUUCGCAUAU .....(((...((((((((((((((....)))))))))....((((((..((.(((((......))))).)).))))(((((((((......)))))))))..)).)))))..))).... ( -45.60) >DroSec_CAF1 8440 119 - 1 GAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAGAGAGUGCUAUAGCUAGAUGUGUACAUGGUCACAUCUGUAUCGUUCAUUUCGCAUAU (((((.......((.((((((((((....)))))))))).)).(((((-.((.(((((......))))).)).)))))(((((((........))))))))))))............... ( -43.30) >DroSim_CAF1 8463 119 - 1 GAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAAAGAGUGCUAUAGCUAGAUGUGUACAUGGGCACAUCUGUAUCGUACAUUUCGCAUAU .....(((...((((((((((((((....)))))))))....((((((-.((.(((((......))))).)).))))(((((((((......)))))))))..)).)))))..))).... ( -47.10) >DroEre_CAF1 8697 119 - 1 GAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAAAGAGCGCUAUGGCUAGAUGUGUAUAUGGGCACAUCUGUAUCGUACAUUUCGCAUAU .....(((...((((((((((((((....)))))))))....((((((-.((.(((((......))))).)).))))(((((((((......)))))))))..)).)))))..))).... ( -48.40) >DroYak_CAF1 8597 119 - 1 GAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGCGCUA-AGGGGAUCUACAUAAAGAGCGCUAUGGCUAGAUGUGUACAUGGGCACAUCUGUAUCGUACAUUUCGCAUAU .....(((...((((((((((((((....)))))))))..(((((((.-...(......)......)))))))..((.((((((((......))))))))))....)))))..))).... ( -42.70) >consensus GAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA_AGGGGCUCUACAUAAAGAGUGCUAUAGCUAGAUGUGUACAUGGGCACAUCUGUAUCGUACAUUUCGCAUAU (((((((.(((((((((((((((((....))))))))))..((((((.....)))))))))))))..))((....)).((((((((......)))))))))))))............... (-42.30 = -42.50 + 0.20)


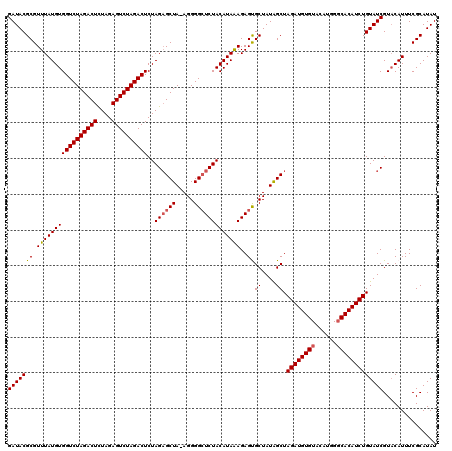
| Location | 9,511,436 – 9,511,556 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.32 |
| Mean single sequence MFE | -38.30 |
| Consensus MFE | -36.28 |
| Energy contribution | -36.40 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.95 |
| SVM decision value | 4.04 |
| SVM RNA-class probability | 0.999770 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511436 120 + 20766785 CUAGCUAUAGCACUCUUUAUGUAGAGCCCCUCUAGCUCUAGAGUCUAGACUCUAAAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...(((((...((.(((((((((((((.......))))).(.(((((((((....))))))))))))))))).).))....)))))..((..((((((.....))))))...))...... ( -36.50) >DroSec_CAF1 8480 119 + 1 CUAGCUAUAGCACUCUCUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...((....))......((((((((((....-..))))).(.(((((((((....)))))))))))))))..(.(((.(((.((....))..((((((.....))))))...)))))).) ( -35.40) >DroSim_CAF1 8503 119 + 1 CUAGCUAUAGCACUCUUUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...(((((...((.(((((((((((((....-..))))).(.(((((((((....))))))))))))))))).).))....)))))..((..((((((.....))))))...))...... ( -38.00) >DroEre_CAF1 8737 119 + 1 CUAGCCAUAGCGCUCUUUAUGUAGAGCCCCU-UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...(((((.((((..((((((((((((....-..))))).(.(((((((((....))))))))))))))))).))))....)))))..((..((((((.....))))))...))...... ( -44.80) >DroYak_CAF1 8637 119 + 1 CUAGCCAUAGCGCUCUUUAUGUAGAUCCCCU-UAGCGCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...(((((.((((..(((((((.......((-.......)).(((((((((....))))))))).))))))).))))....)))))..((..((((((.....))))))...))...... ( -36.80) >consensus CUAGCUAUAGCACUCUUUAUGUAGAGCCCCU_UAGCUCUAGAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGG ...(((((...((.(((((((((((((.......))))).(.(((((((((....))))))))))))))))).).))....)))))..((..((((((.....))))))...))...... (-36.28 = -36.40 + 0.12)

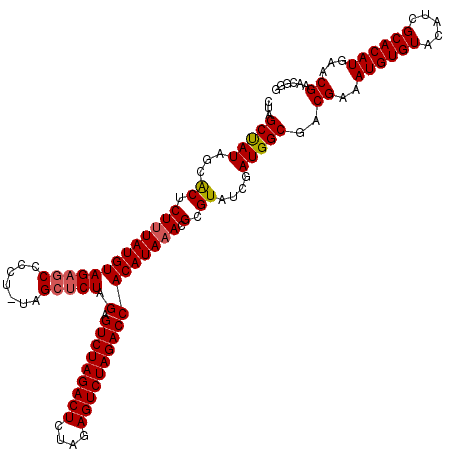
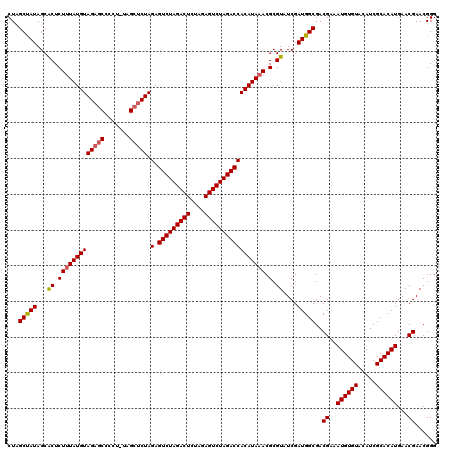
| Location | 9,511,436 – 9,511,556 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.32 |
| Mean single sequence MFE | -42.02 |
| Consensus MFE | -38.98 |
| Energy contribution | -38.98 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.00 |
| SVM RNA-class probability | 0.999748 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511436 120 - 20766785 CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUUUAGAGUCUAGACUCUAGAGCUAGAGGGGCUCUACAUAAAGAGUGCUAUAGCUAG ..(((..(((.(((.((((((.........))))))))).)))..)))....((.((((((((((....)))))))))).)).(((((..((.(((((......))))).)).))))).. ( -40.80) >DroSec_CAF1 8480 119 - 1 CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAGAGAGUGCUAUAGCUAG ..(((..(((.(((.((((((.........))))))))).)))..)))....((.((((((((((....)))))))))).)).(((((-.((.(((((......))))).)).))))).. ( -42.40) >DroSim_CAF1 8503 119 - 1 CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAAAGAGUGCUAUAGCUAG ..(((..(((.(((.((((((.........))))))))).)))..)))....((.((((((((((....)))))))))).)).(((((-.((.(((((......))))).)).))))).. ( -42.30) >DroEre_CAF1 8737 119 - 1 CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA-AGGGGCUCUACAUAAAGAGCGCUAUGGCUAG .((((.((((((((.((((((.........))))))))).........(((((((((((((((((....))))))))))..((((((.-...))))))))))))))))))..)))).... ( -45.20) >DroYak_CAF1 8637 119 - 1 CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGCGCUA-AGGGGAUCUACAUAAAGAGCGCUAUGGCUAG .((((.((((((((.((((((.........))))))))).........(((((((((((((((((....))))))))((((.......-.))))..))))))))))))))..)))).... ( -39.40) >consensus CCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUCUAGAGCUA_AGGGGCUCUACAUAAAGAGUGCUAUAGCUAG ..(((..(((.(((.((((((.........))))))))).)))..)))(((((((((((((((((....))))))))))..((((((.....))))))))))))).(((......))).. (-38.98 = -38.98 + 0.00)


| Location | 9,511,476 – 9,511,596 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.99 |
| Mean single sequence MFE | -33.58 |
| Consensus MFE | -30.58 |
| Energy contribution | -30.42 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.809007 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511476 120 + 20766785 GAGUCUAGACUCUAAAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAGUGAGUAUGUACAGUUUAAGAGAGCGAGGCAAUAUG ..(((((((((....)))))))))(((....((((((.(((.......))).))))))...((((...((......))..)))))))....(((...((((....))))...)))..... ( -32.20) >DroSec_CAF1 8519 120 + 1 GAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAGUGAGUAUGUACAGUUUAAGAGAGCGAGGCAAUACG ..(((((((((....)))))))))(((....((((((.(((.......))).))))))...((((...((......))..))))))).((((((...((((....))))...)).)))). ( -35.30) >DroSim_CAF1 8542 120 + 1 GAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAGUGAGUAUGUACAGUUUAAGAGAGCGAGGCAAUACG ..(((((((((....)))))))))(((....((((((.(((.......))).))))))...((((...((......))..))))))).((((((...((((....))))...)).)))). ( -35.30) >DroEre_CAF1 8776 116 + 1 GAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAG----UAUGUACAGUUUAAGAGAGCGAGGCAAUACG (.(((((((((....))))))))))......((((((.(((.......))).))))))...((((...((......))..))))(----(((((...((((....))))...)).)))). ( -32.50) >DroYak_CAF1 8676 116 + 1 GAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAG----UGUGUACAGUUUAAGAGAGCGAGGCAAUACG ..(((((((((....)))))))))(((....((((((.(((.......))).))))))...((((...((......))..)))))----))(((...((((....))))...)))..... ( -32.60) >consensus GAGUCUAGACUCUAGAGUCUAGACCACAUAAACGCGUAUCGAUGGCGACGAAAUGUGUACAUCGCACAUGAACGAACGGGGCGAGUGAGUAUGUACAGUUUAAGAGAGCGAGGCAAUACG (.(((((((((....))))))))))(((((.((((((.(((.......))).))))))...((((...((......))..)))).....)))))...((((....))))........... (-30.58 = -30.42 + -0.16)

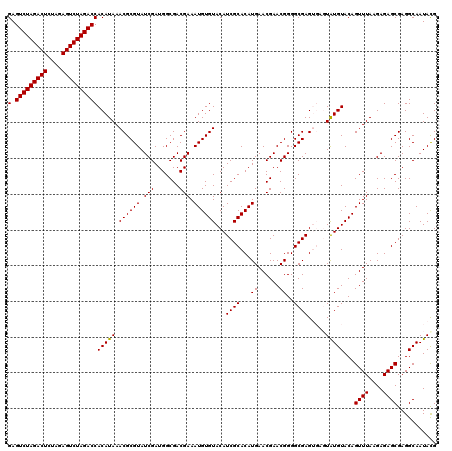
| Location | 9,511,476 – 9,511,596 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.99 |
| Mean single sequence MFE | -30.97 |
| Consensus MFE | -29.48 |
| Energy contribution | -29.32 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.95 |
| SVM decision value | 2.66 |
| SVM RNA-class probability | 0.996179 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9511476 120 - 20766785 CAUAUUGCCUCGCUCUCUUAAACUGUACAUACUCACUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUUUAGAGUCUAGACUC (((((.....(((..........(((((((.(.(((.................))).))))))))......((((....))))..)))...)))))(((((((((....))))))))).. ( -30.43) >DroSec_CAF1 8519 120 - 1 CGUAUUGCCUCGCUCUCUUAAACUGUACAUACUCACUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUC .........................((((((......(((.....(((...(((.((((((.........))))))))))))...)))..))))))(((((((((....))))))))).. ( -29.90) >DroSim_CAF1 8542 120 - 1 CGUAUUGCCUCGCUCUCUUAAACUGUACAUACUCACUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUC .........................((((((......(((.....(((...(((.((((((.........))))))))))))...)))..))))))(((((((((....))))))))).. ( -29.90) >DroEre_CAF1 8776 116 - 1 CGUAUUGCCUCGCUCUCUUAAACUGUACAUA----CUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUC .........................((((((----..(((.....(((...(((.((((((.........))))))))))))...)))..))))))(((((((((....))))))))).. ( -33.40) >DroYak_CAF1 8676 116 - 1 CGUAUUGCCUCGCUCUCUUAAACUGUACACA----CUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUC ...........................((((----..(((.....(((...(((.((((((.........))))))))))))...)))....))))(((((((((....))))))))).. ( -31.20) >consensus CGUAUUGCCUCGCUCUCUUAAACUGUACAUACUCACUCGCCCCGUUCGUUCAUGUGCGAUGUACACAUUUCGUCGCCAUCGAUACGCGUUUAUGUGGUCUAGACUCUAGAGUCUAGACUC ...........................((((......(((.....(((...(((.((((((.........))))))))))))...)))....))))(((((((((....))))))))).. (-29.48 = -29.32 + -0.16)

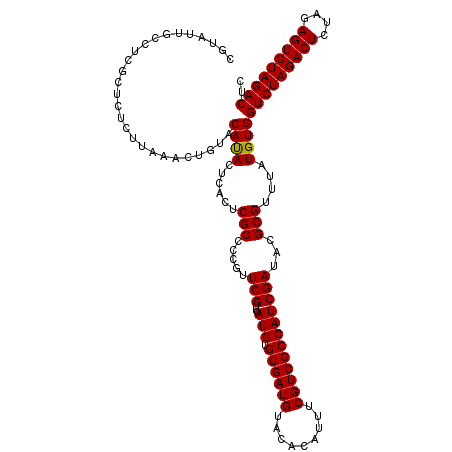
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:37:03 2006