
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,470,609 – 9,470,751 |
| Length | 142 |
| Max. P | 0.966758 |

| Location | 9,470,609 – 9,470,714 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.55 |
| Mean single sequence MFE | -52.63 |
| Consensus MFE | -35.07 |
| Energy contribution | -35.93 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.37 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966758 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
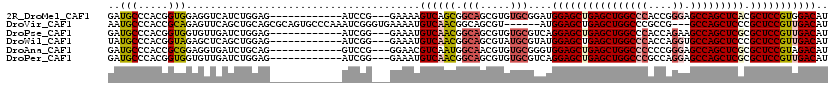
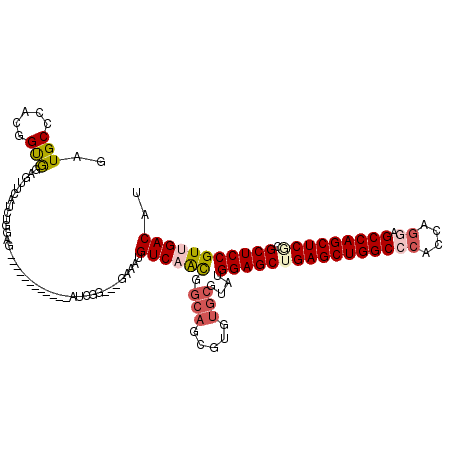
>2R_DroMel_CAF1 9470609 105 + 20766785 GAUGCCCACGGUGGAGGUCAUCUGGAG------------AUCCG---GAAAAGUCAGCGGCAGCGUGUGCGGAUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCACGCUCCGUGGACAU (((..((.....))..))).(((((..------------..)))---))...(((..(.(((.....))).)((((((((((((((((((....)).))))))))).))))))).))).. ( -48.90) >DroVir_CAF1 8759 111 + 1 AAUGCCCACCGCAGAGUUCAGCUGCAGCGCAGUGCCCAAAUCGGGUGAAAAUGUCAACGGCAGCGU------AUGGAGCUGAGCUGGCCCGCCG---GCCAGCUCCCGCUCCGUUGACAU ..(((.....)))...(((.(((((...)))))((((.....))))))).((((((((((.((((.------..(((((((.(((((....)))---))))))))))))))))))))))) ( -56.30) >DroPse_CAF1 6356 105 + 1 GAUGCCCACGGUGGUGUUGAUCUGGAG------------AUCGG---GAAAUGUCAACGGCAGCGUGUGCGUCAGGAGCUGAGCUGGCCCACCAGAAGCCAGCUCGCGCUCCGUUGACAU (((((.((((.((.(((((((((.(..------------..).)---))....)))))).)).)))).))))).((((((((((((((.........))))))))).)))))........ ( -53.20) >DroWil_CAF1 31126 105 + 1 UAUGCCCACGGUAGAGCUCAGCUGGAG------------AUCGG---GAAAUGUCAACGGCAGCGUAUGCGUAUGGAGCUGAGCUGGCCCACCAGGUGCCAGCUCCCGCUCCGUUGACAU ....(((.((((.(....).))))(..------------..)))---)..((((((((.(((.....)))....(((((.((((((((((....)).))))))))..))))))))))))) ( -47.80) >DroAna_CAF1 6588 105 + 1 GAUGCCCACCGCGGAGGUGAUCUGCAG------------GUCCG---GGAACGUCAAUGGCAACGUGUGCGGGUGGAGCUGAGCUGGCCCCCCGGGAGCCAGCUCGCGCUCCGUAGACAU (((((((((((((((.....))))).)------------))..)---))..))))...(....)((.(((....((((((((((((((((....)).))))))))).)))))))).)).. ( -51.60) >DroPer_CAF1 6294 105 + 1 GAUGCCCACGGUGGUGUUGAUCUGGAG------------AUCGG---GAAAUGUCAACGGCAGCGUGUGCGUCAGGAGCUGAGCUGGCCCGCCAGGAGCCAGCUCGCGCUCCGUUGACAU (((((.((((.((.(((((((((.(..------------..).)---))....)))))).)).)))).))))).((((((((((((((((....)).))))))))).)))))........ ( -58.00) >consensus GAUGCCCACGGUGGAGUUCAUCUGGAG____________AUCGG___GAAAUGUCAACGGCAGCGUGUGCGUAUGGAGCUGAGCUGGCCCACCAGGAGCCAGCUCGCGCUCCGUUGACAU ..(((.....))).......................................((((((.(((.....)))....((((((((((((((((....)).))))))))).))))))))))).. (-35.07 = -35.93 + 0.86)
| Location | 9,470,609 – 9,470,714 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.55 |
| Mean single sequence MFE | -49.40 |
| Consensus MFE | -35.11 |
| Energy contribution | -36.20 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.34 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.964596 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
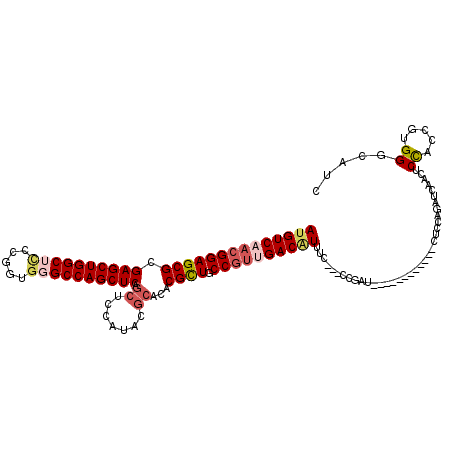
>2R_DroMel_CAF1 9470609 105 - 20766785 AUGUCCACGGAGCGUGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUCCGCACACGCUGCCGCUGACUUUUC---CGGAU------------CUCCAGAUGACCUCCACCGUGGGCAUC (((((((((((((.(((((((((((.....)))))))))))))).((((.(((.....)))............---.((..------------..)).)))).......)))))))))). ( -47.00) >DroVir_CAF1 8759 111 - 1 AUGUCAACGGAGCGGGAGCUGGC---CGGCGGGCCAGCUCAGCUCCAU------ACGCUGCCGUUGACAUUUUCACCCGAUUUGGGCACUGCGCUGCAGCUGAACUCUGCGGUGGGCAUU (((((((((((((((((((((((---.((....)).)).)))))))..------.)))).)))))))))).....(((.....))).....((((((((.......))))))))...... ( -53.70) >DroPse_CAF1 6356 105 - 1 AUGUCAACGGAGCGCGAGCUGGCUUCUGGUGGGCCAGCUCAGCUCCUGACGCACACGCUGCCGUUGACAUUUC---CCGAU------------CUCCAGAUCAACACCACCGUGGGCAUC ((((((((((((((.((((((((((.....)))))))))).((.......))...)))).))))))))))..(---(((((------------(....)))).((......))))).... ( -46.70) >DroWil_CAF1 31126 105 - 1 AUGUCAACGGAGCGGGAGCUGGCACCUGGUGGGCCAGCUCAGCUCCAUACGCAUACGCUGCCGUUGACAUUUC---CCGAU------------CUCCAGCUGAGCUCUACCGUGGGCAUA ((((((((((((((.((((((((.((....)))))))))).((.......))...)))).))))))))))...---.....------------..........((((......))))... ( -48.00) >DroAna_CAF1 6588 105 - 1 AUGUCUACGGAGCGCGAGCUGGCUCCCGGGGGGCCAGCUCAGCUCCACCCGCACACGUUGCCAUUGACGUUCC---CGGAC------------CUGCAGAUCACCUCCGCGGUGGGCAUC (((((((((((((..(((((((((((...))))))))))).)))))...(((..(((((......)))))...---.(((.------------.((.....))..)))))))))))))). ( -52.40) >DroPer_CAF1 6294 105 - 1 AUGUCAACGGAGCGCGAGCUGGCUCCUGGCGGGCCAGCUCAGCUCCUGACGCACACGCUGCCGUUGACAUUUC---CCGAU------------CUCCAGAUCAACACCACCGUGGGCAUC ((((((((((((((.((((((((((.....)))))))))).((.......))...)))).))))))))))..(---(((((------------(....)))).((......))))).... ( -48.60) >consensus AUGUCAACGGAGCGCGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUACGCACACGCUGCCGUUGACAUUUC___CCGAU____________CUCCAGAUCAACUCCACCGUGGGCAUC ((((((((((((((.((((((((((.....)))))))))).((.......))...)))).))))))))))....................................((.....))..... (-35.11 = -36.20 + 1.09)

| Location | 9,470,637 – 9,470,751 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.88 |
| Mean single sequence MFE | -55.47 |
| Consensus MFE | -37.72 |
| Energy contribution | -40.25 |
| Covariance contribution | 2.53 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938429 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
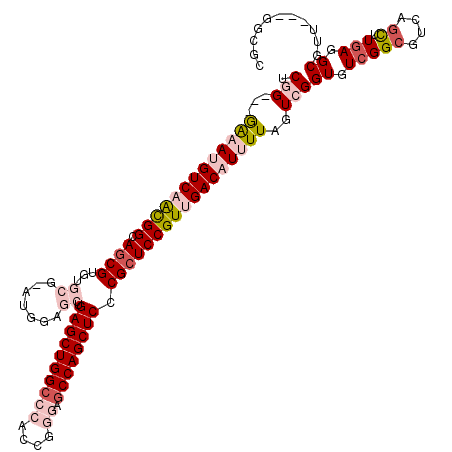
>2R_DroMel_CAF1 9470637 114 + 20766785 UCCG---GAAAAGUCAGCGGCAGCGUGUGCGGAUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCACGCUCCGUGGACAUUUUAGUCGGUGUCGGCGUCAGUUUGAGGCGGC---GUCGC ....---.....(.((.((((((.((((.(..((((((((((((((((((....)).))))))))).)))))))).)))).)).)))).)).)((((((.((.....)))))---))).. ( -53.00) >DroVir_CAF1 8799 105 + 1 UCGGGUGAAAAUGUCAACGGCAGCGU------AUGGAGCUGAGCUGGCCCGCCG---GCCAGCUCCCGCUCCGUUGACAUUUUAGUCGGUGUCGGCGUCAGCUUGAGGCGUU------GC ((((.(((((.(((((((((.((((.------..(((((((.(((((....)))---))))))))))))))))))))))))))).))))...(((((((.......))))))------). ( -57.00) >DroGri_CAF1 7377 108 + 1 UCGGGUGGAAAUGUCAACGGCAGCGU------AUGGAGCUGAGCUGGGCCCCCC---GCCAGCUCCCGCUCCGUUGACAUCUUAGUCGGUGUCGGCGUCAGCUUGAGGCGUU---GCUUC ((((.(((..((((((((((.((((.------..(((((((.((.((....)).---))))))))))))))))))))))).))).)))).(.(((((((.......))))))---))... ( -51.30) >DroWil_CAF1 31154 113 + 1 UCGG---GAAAUGUCAACGGCAGCGUAUGCGUAUGGAGCUGAGCUGGCCCACCAGGUGCCAGCUCCCGCUCCGUUGACAUUUUGGUCGGUGUGGGAGUCAGCUUGAGGCGUU---UGCA- ...(---.(((((((....(((.....)))...(.(((((((((((((((....)).))))))((((((.((((..(....)..).))).)))))).))))))).)))))))---).).- ( -51.50) >DroAna_CAF1 6616 117 + 1 UCCG---GGAACGUCAAUGGCAACGUGUGCGGGUGGAGCUGAGCUGGCCCCCCGGGAGCCAGCUCGCGCUCCGUAGACAUCUUGGUCGGUGUCGGCGUCAGUUUGAGGCUGCUCCGGCGC ..((---(((.(((..(((....)))..)))...((((((((((((((((....)).))))))))).))))).......)))))(((((.(.((((.((.....)).))))).))))).. ( -59.70) >DroPer_CAF1 6322 113 + 1 UCGG---GAAAUGUCAACGGCAGCGUGUGCGUCAGGAGCUGAGCUGGCCCGCCAGGAGCCAGCUCGCGCUCCGUUGACAUUUUGGUCGGUGUCGGCGUCAGUUUGAGGCGUU---GAUG- ((((---((((((((((((((.((....))))).((((((((((((((((....)).))))))))).))))))))))))))))..)))).(((((((((.......))))))---))).- ( -60.30) >consensus UCGG___GAAAUGUCAACGGCAGCGUGUGCG_AUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCCCGCUCCGUUGACAUUUUAGUCGGUGUCGGCGUCAGCUUGAGGCGUU___GGCGC .(((...(((((((((((((.((((...((.......)).((((((((((....)).)))))))).)))))))))))))))))..)))((.(((((....)).))).))........... (-37.72 = -40.25 + 2.53)

| Location | 9,470,637 – 9,470,751 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.88 |
| Mean single sequence MFE | -45.70 |
| Consensus MFE | -34.15 |
| Energy contribution | -36.35 |
| Covariance contribution | 2.20 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925209 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9470637 114 - 20766785 GCGAC---GCCGCCUCAAACUGACGCCGACACCGACUAAAAUGUCCACGGAGCGUGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUCCGCACACGCUGCCGCUGACUUUUC---CGGA (((..---((.((.((.....)).((.((....(((......)))...(((((.(((((((((((.....)))))))))))))))).)).))....)).))))).........---.... ( -42.40) >DroVir_CAF1 8799 105 - 1 GC------AACGCCUCAAGCUGACGCCGACACCGACUAAAAUGUCAACGGAGCGGGAGCUGGC---CGGCGGGCCAGCUCAGCUCCAU------ACGCUGCCGUUGACAUUUUCACCCGA ..------...((.((.....)).))......((...((((((((((((((((((((((((((---.((....)).)).)))))))..------.)))).)))))))))))))....)). ( -45.10) >DroGri_CAF1 7377 108 - 1 GAAGC---AACGCCUCAAGCUGACGCCGACACCGACUAAGAUGUCAACGGAGCGGGAGCUGGC---GGGGGGCCCAGCUCAGCUCCAU------ACGCUGCCGUUGACAUUUCCACCCGA .....---...((.((.....)).))......((....(((((((((((((((((((((((((---.((....)).)).)))))))..------.)))).)))))))))))).....)). ( -44.60) >DroWil_CAF1 31154 113 - 1 -UGCA---AACGCCUCAAGCUGACUCCCACACCGACCAAAAUGUCAACGGAGCGGGAGCUGGCACCUGGUGGGCCAGCUCAGCUCCAUACGCAUACGCUGCCGUUGACAUUUC---CCGA -....---...((.....))............((....((((((((((((((((.((((((((.((....)))))))))).((.......))...)))).)))))))))))).---.)). ( -45.10) >DroAna_CAF1 6616 117 - 1 GCGCCGGAGCAGCCUCAAACUGACGCCGACACCGACCAAGAUGUCUACGGAGCGCGAGCUGGCUCCCGGGGGGCCAGCUCAGCUCCACCCGCACACGUUGCCAUUGACGUUCC---CGGA ...((((.((.((.((.....)).)).((((.(......).))))...(((((..(((((((((((...))))))))))).)))))....))..(((((......)))))..)---))). ( -51.10) >DroPer_CAF1 6322 113 - 1 -CAUC---AACGCCUCAAACUGACGCCGACACCGACCAAAAUGUCAACGGAGCGCGAGCUGGCUCCUGGCGGGCCAGCUCAGCUCCUGACGCACACGCUGCCGUUGACAUUUC---CCGA -....---...((.((.....)).))......((....((((((((((((((((.((((((((((.....)))))))))).((.......))...)))).)))))))))))).---.)). ( -45.90) >consensus GCGCC___AACGCCUCAAACUGACGCCGACACCGACCAAAAUGUCAACGGAGCGGGAGCUGGCUCCCGGCGGGCCAGCUCAGCUCCAU_CGCACACGCUGCCGUUGACAUUUC___CCGA ...........((.((.....)).))............((((((((((((((((.((((((((.((....)))))))))).((.......))...)))).))))))))))))........ (-34.15 = -36.35 + 2.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:36:20 2006