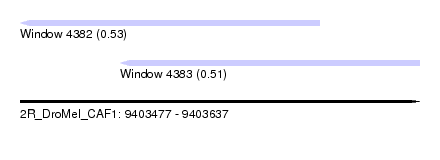
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,403,477 – 9,403,637 |
| Length | 160 |
| Max. P | 0.530727 |

| Location | 9,403,477 – 9,403,597 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.11 |
| Mean single sequence MFE | -46.05 |
| Consensus MFE | -35.42 |
| Energy contribution | -34.70 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.77 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.530727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9403477 120 - 20766785 ACACACAGACUGGACAGGUGGUGCUUGACGAUGGUCGGGUUUGCUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGACAUUCAGUCGAUAUCGGUGCGCGACCCAGUCAGCGGUGAGG .(((...((((((....((.((((((((((((.(((.(((((((..(((((((((.....)))))))))))))))).))).....))))...)))).)))).))))))))....)))... ( -49.60) >DroVir_CAF1 16011 120 - 1 ACUCACAGACAGGAAAAGUGGUACUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAGGCUAUCAACCGUGGCAAACUGGACAUACAGUCGAUAUCGGUGCGCGACCCAGUUAGCGGUGAGG .(((((..........(.(((((.(((((....(((.((((((..((((.(((((.....))))).)))))))))).))).....)))))))))).).(((..........)))))))). ( -38.80) >DroGri_CAF1 16038 120 - 1 AUCCGCAGACAGGUCAAGUGGUGCUAGACGAUGGUCAAGUUUGUUCCAUUGUUGAUGCGAUCAACAGUGGCAAGCUGGACAUUCAGUCGAUAUCGGUGCGCGACCCAGUUAGCGGUGAGG ..((((.(((.((....((.((((....(((((.((.((((((..(((((((((((...))))))))))))))))).(((.....))))))))))..)))).)))).))).))))..... ( -44.50) >DroWil_CAF1 15351 120 - 1 ACUCACAAACCGGUCAGGUAAUACUUGAUGAUGGUCGAGUUUGUUCCAUUGUUGAGGCCAUCAAUAGUGGCAAGCUGGACAUUCAGUCGAUAUCGGUGCGCGACCCAGUUAGCGGUGAAG ..((((..((((((((((.....))))))(((.(((.((((((..((((((((((.....)))))))))))))))).))).....))).....)))).(((..........))))))).. ( -42.60) >DroMoj_CAF1 15926 120 - 1 AUCACCGGUCAGGCAAAGUGGUGCUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAUGCUAUCAACAGUGGCAAACUCGACAUACAGUCGAUAUCGGUGCGCGACCCUGUUAGCGGUGAGG .((((((((((((((......))))))))...((((..(((((..(((((((((((...))))))))))))))))(((((.....)))))...........)))).......)))))).. ( -49.70) >DroPer_CAF1 16161 120 - 1 ACACGGAGACGGGCCAGGUGGUGCUGGACGAUGGUCGGGCUUGCUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGACAUCCAGUCGAUAUCGGUGCGCGACCCAGUUAGCGGCGAGG ...((....)).(((.(((.((((((((.....(((.(((((((..(((((((((.....)))))))))))))))).))).)))).(((....))).)))).)))..(....)))).... ( -51.10) >consensus ACACACAGACAGGACAAGUGGUGCUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGACAUUCAGUCGAUAUCGGUGCGCGACCCAGUUAGCGGUGAGG ...(((..........(.(((((.(((((....(((.((((((..((((((((((.....)))))))))))))))).))).....)))))))))).).((((((...))).))))))... (-35.42 = -34.70 + -0.72)

| Location | 9,403,517 – 9,403,637 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.22 |
| Mean single sequence MFE | -38.01 |
| Consensus MFE | -22.65 |
| Energy contribution | -22.35 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.506015 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9403517 120 - 20766785 GAAGAGCAAAAGCCAUCGGGCAUCGAGAACGUGACGAUAGACACACAGACUGGACAGGUGGUGCUUGACGAUGGUCGGGUUUGCUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGAC ...........(((((((((((((......(((.(....).)))....(((.....)))))))))...)))))))(.(((((((..(((((((((.....)))))))))))))))).).. ( -40.10) >DroVir_CAF1 16051 120 - 1 GAAGAACAGCGACCCGCUCCCAUUGAAAAUGUUACUAUAGACUCACAGACAGGAAAAGUGGUACUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAGGCUAUCAACCGUGGCAAACUGGAC ......(((....((((..(((((.....((((..............)))).....)))))..((..(((((((.((....))..)))))))..))..........))))....)))... ( -27.44) >DroGri_CAF1 16078 120 - 1 GAAGAGCAGCGACCUGCUCCAAUAGAGAAUAUUUGCAUAGAUCCGCAGACAGGUCAAGUGGUGCUAGACGAUGGUCAAGUUUGUUCCAUUGUUGAUGCGAUCAACAGUGGCAAGCUGGAC ....((((.((((((((((.....)))....(((((........)))))))))))....).))))........(((.((((((..(((((((((((...))))))))))))))))).))) ( -39.70) >DroWil_CAF1 15391 120 - 1 GAGGUGCAGCGUCCUGCUGCUAUUGAGAAUGUUGUAAUUGACUCACAAACCGGUCAGGUAAUACUUGAUGAUGGUCGAGUUUGUUCCAUUGUUGAGGCCAUCAAUAGUGGCAAGCUGGAC ...((.((((....(((..(((((((............(((((........)))))(((....((..(..((((...........))))..)..))))).)))))))..))).)))).)) ( -40.00) >DroMoj_CAF1 15966 120 - 1 GAAGAGCAGAGACCUGCAGCAAUUGAGAAUGUUACAAUAGAUCACCGGUCAGGCAAAGUGGUGCUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAUGCUAUCAACAGUGGCAAACUCGAC .....((((....)))).....((((.............(((((.((.(((((((......))))))))).)))))..(((((..(((((((((((...)))))))))))))))))))). ( -39.90) >DroAna_CAF1 15412 120 - 1 GAGGAACAGAAACCGACGGGCAUGGAGAACGUGACCAUUGACCCACAGACCGGACAGGUGAUACUUGACGAUGGUCGGGUUUGCUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGAC ..((........))..(.(((.((..((((.(((((((((..((.......)).((((.....)))).))))))))).))))...((((((((((.....)))))))))))).))).).. ( -40.90) >consensus GAAGAGCAGAGACCUGCUGCCAUUGAGAAUGUUACAAUAGACCCACAGACAGGACAAGUGGUACUUGACGAUGGUCAAGUUUGUUCCAUUGUUGAGGCCAUCAACAGUGGCAAGCUGGAC ....................................................(.((((.....)))).)....(((.((((((..((((((((((.....)))))))))))))))).))) (-22.65 = -22.35 + -0.30)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:35:44 2006