
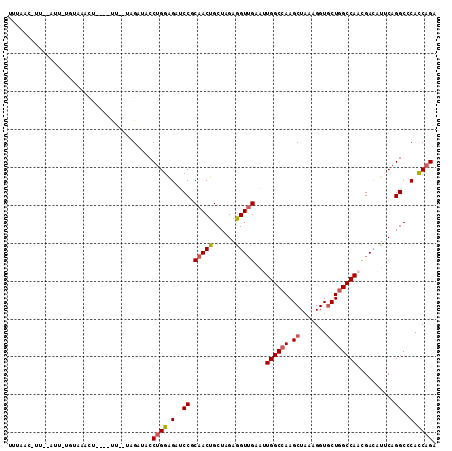
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 9,389,996 – 9,390,242 |
| Length | 246 |
| Max. P | 0.741573 |

| Location | 9,389,996 – 9,390,097 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 77.12 |
| Mean single sequence MFE | -27.60 |
| Consensus MFE | -20.99 |
| Energy contribution | -21.38 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.10 |
| Mean z-score | -0.79 |
| Structure conservation index | 0.76 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.521152 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9389996 101 - 20766785 U------UUCCAUUAUGUAAACU----UU--UAGAUUCCUGGAGAUCCGCAACUGCUAGAGGUUGAAUUGGCCAAGCUAAAGGUGCUGGCCAACGACAUUCAGGCCCACCAGA .------................----..--.......((((.(..((((....))..(((((((..(((((((.((.......))))))))))))).))).))..).)))). ( -26.90) >DroGri_CAF1 1881 99 - 1 ACUAAC-------U-----GAUUUAACUA--CAGAUACCUGGAGACCCGCAACUGCUUGAAGUGGAGCUGGCCAAGCUGAAGGUGCUUGCCAAUGAUAUACAGGCGCAUCAGA .....(-------(-----(((....((.--(((.....(((((..((((..(.....)..))))..))..)))..))).))(((((((...........)))))))))))). ( -24.10) >DroSec_CAF1 1881 107 - 1 UUUAACUUUCCAUUGUGUAAACU----UU--UAGAUUCCUGGAGAUCCGCAACUGCUAGAGGUUGAAUUGGCCAAGCUAAAGGUGCUGGCCAAUGACAUUCAGGCCCACCAGA .........((...((((.((((----((--(((.....(((....)))......)))))))))..((((((((.((.......)))))))))).))))...))......... ( -28.10) >DroEre_CAF1 1881 107 - 1 UUAAACUUUUUAUUUUGUAAACA----CU--UAGAUCCCUGGAGAUCCGCAACUGCUGGAGGUUGAAUUGGCCAAGCUAAAGGUUCUGGCCAACGACAUUCAGGCCCACCAGA .......................----..--.......((((.(..((((....))((((.((((..((((((((((.....))).))))))))))).))))))..).)))). ( -26.50) >DroYak_CAF1 1881 102 - 1 UUCAACAU-----UCCAUAAACU----UU--UAGAUCCCUGGUGAUCCACAACUGCUGGAGGUUGAACUGGCCAAGCUAAAGGUGCUGGCCAACGACAUUCAGGCCCACCAGA ........-----..........----..--.......((((((..((.(((((......)))))...((((((.((.......))))))))..........))..)))))). ( -29.50) >DroPer_CAF1 1881 106 - 1 UGUAACCUUUGGUU---GAAUCC----UUUACAGAUACCCGGGGACCCACAACUGCUCGAGGUUGAGUUGGCCAAGCUGAAGGUGCUGGCCAACGAUAUACAGGCGCACCAGA ..((((((((((((---(.(((.----......)))....((....)).))))))...))))))).((((((((.((.......))))))))))........((....))... ( -30.50) >consensus UUUAAC_UU__AUU_UGUAAACU____UU__UAGAUACCUGGAGAUCCGCAACUGCUAGAGGUUGAAUUGGCCAAGCUAAAGGUGCUGGCCAACGACAUUCAGGCCCACCAGA ......................................((((.(..((.(((((......)))))...((((((.((.......))))))))..........))..).)))). (-20.99 = -21.38 + 0.39)


| Location | 9,390,097 – 9,390,202 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 73.82 |
| Mean single sequence MFE | -26.59 |
| Consensus MFE | -18.39 |
| Energy contribution | -18.12 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.29 |
| Mean z-score | -0.87 |
| Structure conservation index | 0.69 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.512257 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9390097 105 + 20766785 AUGAUUUGGG-UUUCU--CUCAAGCAUGAA--AUAUAU----AUCUACGUACCGGCUUCAGUUGGUCCAGUGUGCUGUCGGUCUUGUUGAUCCAUACCAGCAGCUCGUUGAGAG ..........-...((--(((((.......--......----......(.((((((....)))))).)...(.(((((.(((..((......)).))).))))).).))))))) ( -26.30) >DroVir_CAF1 1980 103 + 1 UAGAUUUUGG-CCAGU--UGGAAGCUA-----CUCCUUCGCUAU---CUUACCGGUUUCAACUGAUCCAAUGUGCUGUCUGUUUUGUUAAUCCAGACCAACAGCUCGUUGAGUG ........(.-.((((--(((((.(..-----............---......).)))))))))..)(((((.(((((.((.((((......)))).))))))).))))).... ( -25.65) >DroGri_CAF1 1980 112 + 1 UCGAUUUUA--CUCUCAUUCAAAGAAAUAACUCUUCUUUGCUUUCUUCUUACCGGCUUCAGCUGAUCCAGAGUGCUGUCUGUCUUGUUUAUCCAGACCAACAGCUCGUUGAGCG (((((...(--((((....(((((((.......)))))))............((((....))))....)))))(((((..((((.(.....).))))..)))))..)))))... ( -26.30) >DroSec_CAF1 1988 105 + 1 ACGAUUUGGU-UUUCU--UACCAGAAUUAA--AUAUAU----AUCUACGUACCGGCUUAAGUUGGUCCAGCGUGCUGUCGGUCUUGUUGAUCCAGACCAGUAGCUCAUUGAGGG ....((((((-.....--.)))))).....--......----.(((((((((((((....))))))...))))((((..(((((.(.....).)))))..))))......))). ( -27.50) >DroEre_CAF1 1988 103 + 1 AAGAAUUGAUUUUUUU--UAUAAGAAUUAU-----UAU----AGUUACGUACCGGCUUCAGUUGGUCCAGUGUGCUGUCGGUCUUGUUGAUCCAGACCAGCAGCUCGUUGAGGG ......((((((((..--...)))))))).-----...----........((((((....)))))).(((((.(((((.(((((.(.....).))))).))))).))))).... ( -26.70) >DroYak_CAF1 1983 103 + 1 ACGAUUUG---UUGUU--UACAAGAAUUAU--AUAUAU----AGUUACGUACCGGCUUCAGUUGGUCCAGAGUGCUGUCGGUCUUGUUGAUCCAGACCAGCAGCUCGUUGAGGG (((.((((---(....--.)))))((((((--....))----)))).)))((((((....)))))).(((.(.(((((.(((((.(.....).))))).))))).).))).... ( -27.10) >consensus ACGAUUUGG__UUUUU__UACAAGAAUUAA__AUAUAU____AUCUACGUACCGGCUUCAGUUGGUCCAGUGUGCUGUCGGUCUUGUUGAUCCAGACCAGCAGCUCGUUGAGGG ..................................................((((((....)))))).(((.(.(((((.(((((.(.....).))))).))))).).))).... (-18.39 = -18.12 + -0.27)


| Location | 9,390,126 – 9,390,242 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.12 |
| Mean single sequence MFE | -38.20 |
| Consensus MFE | -27.67 |
| Energy contribution | -26.98 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.741573 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
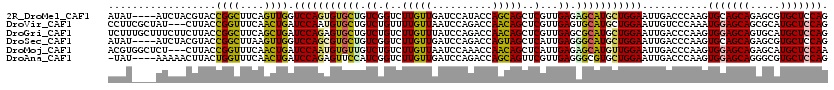
>2R_DroMel_CAF1 9390126 116 + 20766785 AUAU----AUCUACGUACCGGCUUCAGUUGGUCCAGUGUGCUGUCGGUCUUGUUGAUCCAUACCAGCAGCUCGUUGAGAGCAUGCUGGAAUUGACCCAAGUGCAGCAGAGCGUGCUCCAG ....----......(((((.(((.((.((((..((((....(((.((((.....)))).)))((((((((((.....)))).)))))).))))..)))).)).)))...).))))..... ( -39.90) >DroVir_CAF1 2006 117 + 1 CCUUCGCUAU---CUUACCGGUUUCAACUGAUCCAAUGUGCUGUCUGUUUUGUUAAUCCAGACCAACAGCUCGUUGAGUGCAUGCUGGAAUUGUCCCAAAUGGAGCAGCGCAUGCUCCAG ..........---....(((((....(((....(((((.(((((.((.((((......)))).))))))).))))))))....)))))............(((((((.....))))))). ( -34.30) >DroGri_CAF1 2012 120 + 1 UCUUUGCUUUCUUCUUACCGGCUUCAGCUGAUCCAGAGUGCUGUCUGUCUUGUUUAUCCAGACCAACAGCUCGUUGAGCGCAUGCUGGAAUUGACCCAAGUGGAGCAGUGCAUGCUCCAG .....((((...((....((((....)))).(((((.(((((((..((((.(.....).))))..)))((((...)))))))).)))))...))...))))((((((.....)))))).. ( -38.30) >DroSec_CAF1 2017 116 + 1 AUAU----AUCUACGUACCGGCUUAAGUUGGUCCAGCGUGCUGUCGGUCUUGUUGAUCCAGACCAGUAGCUCAUUGAGGGCAUGCUGGAAUUGACCCAAGUGCAGCAGAGCGUGCUCCAG ....----......((((..((((..((..((((((((((((.(((((((.(.....).))))).(.....)...)).)))))))))).))..))..))))((......))))))..... ( -40.00) >DroMoj_CAF1 2002 117 + 1 ACGUGGCUCU---CUUACCGGUUUCAACUGAUCCAAUGUGUUGUCUGUCUUGUUAAUCCAAACCAACAGCUCAUUGAGAGCAUGUUGGAAUUGACCCAAGUGGAGCAGAGCAUGCUCCAA ..........---.....((((((((((((.(((((((.(((((.((..(((......)))..))))))).))))).)).)).)))))))))).......(((((((.....))))))). ( -37.20) >DroAna_CAF1 2014 115 + 1 -UAU----AAAAACUUACUGGUUUCAACUGAUCCAGAGUUCCAUCGGUCUUGUUGAUCCAGACCAGCAGUUCGUUGAGGGCGUGCUGGAAUUGACCCAAGUGGAGCAGGGCGUGCUCCAG -...----....((((.((((..(((((.((.((.((......))))))..))))).)))).((((((((((.....)))).)))))).........))))((((((.....)))))).. ( -39.50) >consensus ACAU____AUCUACUUACCGGCUUCAACUGAUCCAGUGUGCUGUCGGUCUUGUUGAUCCAGACCAACAGCUCGUUGAGAGCAUGCUGGAAUUGACCCAAGUGGAGCAGAGCAUGCUCCAG ..................((((....)))).(((((((((((.((.(..(((((..........)))))..)...)).)))))))))))...........(((((((.....))))))). (-27.67 = -26.98 + -0.69)
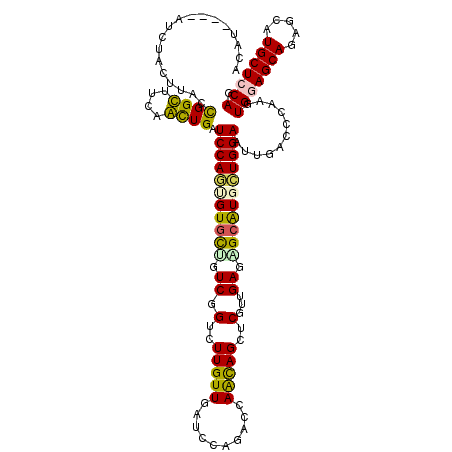
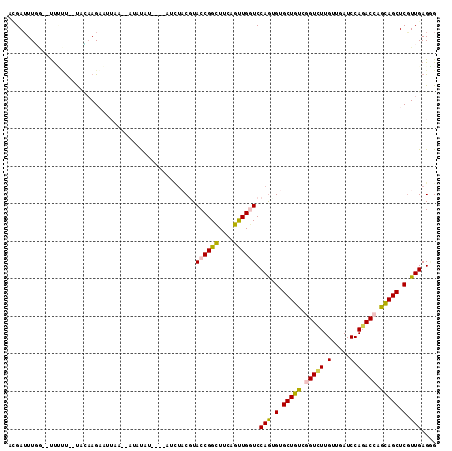
| Location | 9,390,126 – 9,390,242 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.12 |
| Mean single sequence MFE | -34.62 |
| Consensus MFE | -25.11 |
| Energy contribution | -25.17 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.14 |
| Structure conservation index | 0.73 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 9390126 116 - 20766785 CUGGAGCACGCUCUGCUGCACUUGGGUCAAUUCCAGCAUGCUCUCAACGAGCUGCUGGUAUGGAUCAACAAGACCGACAGCACACUGGACCAACUGAAGCCGGUACGUAGAU----AUAU ..((((....))))(((((.((((((((....((((((.((((.....))))))))))....))))..))))...).))))..(((((.(........))))))........----.... ( -36.60) >DroVir_CAF1 2006 117 - 1 CUGGAGCAUGCGCUGCUCCAUUUGGGACAAUUCCAGCAUGCACUCAACGAGCUGUUGGUCUGGAUUAACAAAACAGACAGCACAUUGGAUCAGUUGAAACCGGUAAG---AUAGCGAAGG .(((((((.....))))))).((((..(((((((((..(((...((((.....))))(((((...........))))).)))..)))))...))))...))))....---.......... ( -32.10) >DroGri_CAF1 2012 120 - 1 CUGGAGCAUGCACUGCUCCACUUGGGUCAAUUCCAGCAUGCGCUCAACGAGCUGUUGGUCUGGAUAAACAAGACAGACAGCACUCUGGAUCAGCUGAAGCCGGUAAGAAGAAAGCAAAGA .(((((((.....)))))))(((.(((....(((((..(((((((...))))((((.(((((......).)))).)))))))..)))))...))).)))..................... ( -37.30) >DroSec_CAF1 2017 116 - 1 CUGGAGCACGCUCUGCUGCACUUGGGUCAAUUCCAGCAUGCCCUCAAUGAGCUACUGGUCUGGAUCAACAAGACCGACAGCACGCUGGACCAACUUAAGCCGGUACGUAGAU----AUAU ((((.(((.((...))))).(((((((....((((((.(((.(((...)))....(((((((......).))))))...))).))))))...))))))))))).........----.... ( -33.80) >DroMoj_CAF1 2002 117 - 1 UUGGAGCAUGCUCUGCUCCACUUGGGUCAAUUCCAACAUGCUCUCAAUGAGCUGUUGGUUUGGAUUAACAAGACAGACAACACAUUGGAUCAGUUGAAACCGGUAAG---AGAGCCACGU .(((((((.....)))))))((((.(((....((((((.((((.....))))))))))....)))...)))).....((((...........)))).....(((...---...))).... ( -35.80) >DroAna_CAF1 2014 115 - 1 CUGGAGCACGCCCUGCUCCACUUGGGUCAAUUCCAGCACGCCCUCAACGAACUGCUGGUCUGGAUCAACAAGACCGAUGGAACUCUGGAUCAGUUGAAACCAGUAAGUUUUU----AUA- .(((((((.....)))))))((((((((....((((((((.......))...))))))....))))..))))......((((((((((.((....))..))))..)))))).----...- ( -32.10) >consensus CUGGAGCACGCUCUGCUCCACUUGGGUCAAUUCCAGCAUGCCCUCAACGAGCUGCUGGUCUGGAUCAACAAGACAGACAGCACACUGGAUCAGCUGAAACCGGUAAGUAGAU____AUAU .(((((((.....)))))))((((.(((....((((((.((.(.....).))))))))....)))...))))...........(((((...........)))))................ (-25.11 = -25.17 + 0.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:35:39 2006