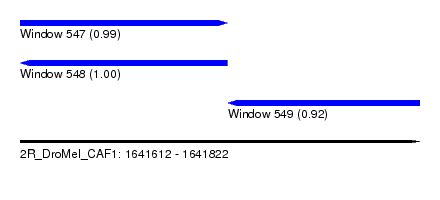
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,641,612 – 1,641,822 |
| Length | 210 |
| Max. P | 0.999377 |

| Location | 1,641,612 – 1,641,721 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 71.92 |
| Mean single sequence MFE | -33.46 |
| Consensus MFE | -10.48 |
| Energy contribution | -10.32 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.31 |
| SVM decision value | 2.47 |
| SVM RNA-class probability | 0.994333 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1641612 109 + 20766785 UGUUGCAGUGCACAGAUAGAUUACACAUAAUGUAUCUG------AGCAUCA-----CUCUCCAGCUAUUUAGUCUACGUGUGUGUAUCUAUGUGCAUUUAGCGAGCGAGAUAGCUGGCUA .......((((.((((((.((((....)))).))))))------.))))..-----....((((((((((.(.((.(((..((((((....))))))...)))))).))))))))))... ( -36.40) >DroPse_CAF1 54539 107 + 1 GCUUACAACUCACAGA-------CACAUUAUGUAUCUGUAUCUUUGCACCAGCUAUCUCUCCAUCUGUGCA-UGUGUAUGUGGGCAUCUGUGUGCACUCGACGAGGGAGAUAGU-----G ((.........(((((-------.((.....)).)))))......))....(((((((((((.((.(((((-..(..(((....)))..)..)))))..)).).))))))))))-----. ( -33.06) >DroSec_CAF1 27453 109 + 1 UGUUGCAGUGCACAGAUAGAUAACACAUAAUGUAUCUG------AGCAUCA-----CUCUCCAGCUAUAUAGUCUACGUGUGUGUAUCUAUGUGCAUUUAGCGAGCGAGAUAGCUGGCUA ......((((((((..((((((.((((((.((((.(((------(((....-----.......)))...)))..))))))))))))))))))))))))((((.(((......))).)))) ( -34.50) >DroEre_CAF1 30267 107 + 1 UGUUGCAGUGCACAGAUAGAUAACACAUAAUGUAUCUG------AGCAUCA-----CUCUCCAGCUAUAUUGUCUACGUGUGUGUAUCUAU--GCAUUUAGCGAGCGAGAUAGCUAGCUA ......((((((.(((((.(((.(((((((((((.(((------((.....-----..)).))).))))))))....)))))).))))).)--)))))((((.(((......))).)))) ( -30.30) >DroPer_CAF1 48968 107 + 1 GCUUACAACUCACAGA-------CACAUUAUGUAUCUGUAUCUUUGCACCAGCUAUCUCUCCAUCUGUGCA-UGUGUAUGUGGGCAUCUGUGUGCACUCGACGAGGGAGAUAGU-----G ((.........(((((-------.((.....)).)))))......))....(((((((((((.((.(((((-..(..(((....)))..)..)))))..)).).))))))))))-----. ( -33.06) >consensus UGUUGCAGUGCACAGAUAGAU_ACACAUAAUGUAUCUG______AGCAUCA_____CUCUCCAGCUAUAUAGUCUACGUGUGUGUAUCUAUGUGCAUUUAGCGAGCGAGAUAGCU_GCUA ....((......((((((.((........)).))))))...................((((..(((.......(((..(((..((....))..)))..)))..)))))))..))...... (-10.48 = -10.32 + -0.16)

| Location | 1,641,612 – 1,641,721 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 71.92 |
| Mean single sequence MFE | -32.80 |
| Consensus MFE | -8.89 |
| Energy contribution | -9.49 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.24 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.27 |
| SVM decision value | 3.55 |
| SVM RNA-class probability | 0.999377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1641612 109 - 20766785 UAGCCAGCUAUCUCGCUCGCUAAAUGCACAUAGAUACACACACGUAGACUAAAUAGCUGGAGAG-----UGAUGCU------CAGAUACAUUAUGUGUAAUCUAUCUGUGCACUGCAACA ...((((((((.......((.....))...(((.(((......)))..))).))))))))...(-----(..(((.------((((((.((((....)))).)))))).)))..)).... ( -30.40) >DroPse_CAF1 54539 107 - 1 C-----ACUAUCUCCCUCGUCGAGUGCACACAGAUGCCCACAUACACA-UGCACAGAUGGAGAGAUAGCUGGUGCAAAGAUACAGAUACAUAAUGUG-------UCUGUGAGUUGUAAGC (-----((((((((.(.((((..(((((.....(((....))).....-))))).))))).))))))).)).(((((...(((((((((.....)))-------))))))..)))))... ( -37.30) >DroSec_CAF1 27453 109 - 1 UAGCCAGCUAUCUCGCUCGCUAAAUGCACAUAGAUACACACACGUAGACUAUAUAGCUGGAGAG-----UGAUGCU------CAGAUACAUUAUGUGUUAUCUAUCUGUGCACUGCAACA ((((.(((......))).))))..((((((((((((...((((((((.(((......))).(((-----.....))------).......))))))))))))))..))))))........ ( -29.70) >DroEre_CAF1 30267 107 - 1 UAGCUAGCUAUCUCGCUCGCUAAAUGC--AUAGAUACACACACGUAGACAAUAUAGCUGGAGAG-----UGAUGCU------CAGAUACAUUAUGUGUUAUCUAUCUGUGCACUGCAACA ((((.(((......))).))))..(((--(((((((...((((((((.((.......))..(((-----.....))------).......))))))))....))))))))))........ ( -29.30) >DroPer_CAF1 48968 107 - 1 C-----ACUAUCUCCCUCGUCGAGUGCACACAGAUGCCCACAUACACA-UGCACAGAUGGAGAGAUAGCUGGUGCAAAGAUACAGAUACAUAAUGUG-------UCUGUGAGUUGUAAGC (-----((((((((.(.((((..(((((.....(((....))).....-))))).))))).))))))).)).(((((...(((((((((.....)))-------))))))..)))))... ( -37.30) >consensus UAGC_AGCUAUCUCGCUCGCUAAAUGCACAUAGAUACACACACGUAGACUACAUAGCUGGAGAG_____UGAUGCU______CAGAUACAUUAUGUGU_AUCUAUCUGUGCACUGCAACA ......(((((((.....((.....))....)))))..((((.(((((..(((((..((.............................))...)))))..))))).))))....)).... ( -8.89 = -9.49 + 0.60)

| Location | 1,641,721 – 1,641,822 |
|---|---|
| Length | 101 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.55 |
| Mean single sequence MFE | -29.22 |
| Consensus MFE | -23.73 |
| Energy contribution | -24.29 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918261 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1641721 101 - 20766785 UUGUCG--GUCGGUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUUGAGCGACAGGC---------AUGUAGACAAGUUUUUCUCCUUUUU--UUU------CUGUUUUC (((((.--(((.(((((((((((((((((((((....)))))))))))....)).)))))))).)))---------.....)))))...............--...------........ ( -29.50) >DroPse_CAF1 54646 118 - 1 UUGUCGGGGUCUCUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUUGUACGACAGGCAUCGGAGCCAUGUAGACAAGUUUUUCUUUUUCUU--UUCCUUUAGCUGUUCUC .....(((....)))(..(((((((((((((((....)))))))))))...(((((((...((((((......))).)))..)))))))............--........))))..).. ( -28.80) >DroSec_CAF1 27562 103 - 1 UUGUCG--GUCGGUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUCGAGCGACAGGC---------AUGUAGACAAGUUUUUCUCCUUUUUUGUUU------CUGUUUUC (((((.--(((.(((((((((((((((((((((....)))))))))))....))).))))))).)))---------.....)))))....................------........ ( -29.50) >DroEre_CAF1 30374 103 - 1 UUGUCG--GUCGGUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUUGAGCGACAGGC---------AUGUAGACAAGUUUUUCUCCACUUUUGUUU------CUAUUUUU (((((.--(((.(((((((((((((((((((((....)))))))))))....)).)))))))).)))---------.....)))))....................------........ ( -29.50) >DroPer_CAF1 49075 118 - 1 UUGUCGGGGUCUCUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUUGUACGACAGGCAUCGGAGCCAUGUAGACAAGUUUUUCUUUUUCUU--UUCCUUUAGCUGUUCUC .....(((....)))(..(((((((((((((((....)))))))))))...(((((((...((((((......))).)))..)))))))............--........))))..).. ( -28.80) >consensus UUGUCG__GUCGGUCGUUCAGUGGAUUUGUGUGUCUUCGCAUAAAUUUUGCAGCUUGAGCGACAGGC_________AUGUAGACAAGUUUUUCUCCUUUUU__UUU______CUGUUUUC (((((...(((.(((((((((((((((((((((....)))))))))))....))).))))))).)))..............))))).................................. (-23.73 = -24.29 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:33:46 2006