
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 8,964,514 – 8,964,623 |
| Length | 109 |
| Max. P | 0.941577 |

| Location | 8,964,514 – 8,964,623 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 75.29 |
| Mean single sequence MFE | -32.95 |
| Consensus MFE | -20.00 |
| Energy contribution | -21.06 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.936722 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
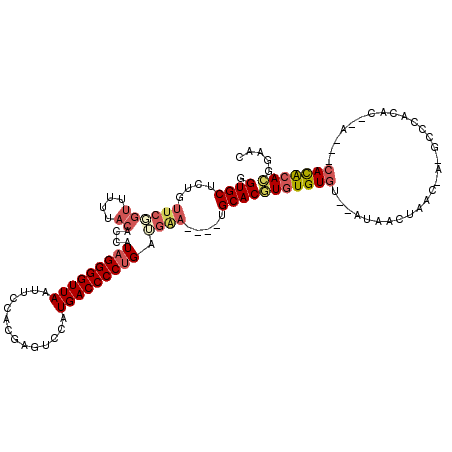
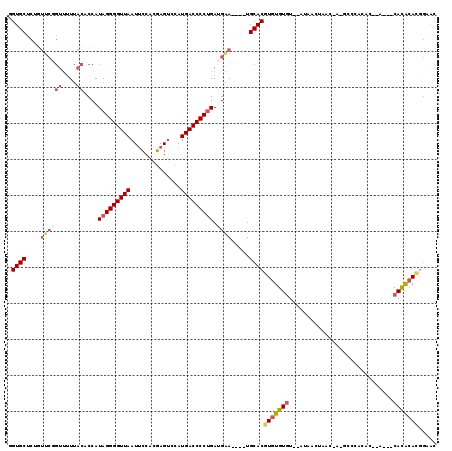
>2R_DroMel_CAF1 8964514 109 + 20766785 GGUGCUCUGUUCGGUUUUUACCCCAUAGGGGUUAAUUCCACGAGUCCAUGACCCCUGAUGAA----UGCACAUGUGUAU--AUAACUAACUA-ACCCACACACA---CACACAUGGAGC ...((((.(((((((....)))...(((((((((((((...))))...)))))))))..)))----)...(((((((..--...........-...........---.))))))))))) ( -27.51) >DroSec_CAF1 11845 101 + 1 GGUGCUCUGUUCGGUUUUUACACCAUAGGGGUUAAUUCCACGAGUCCAUGACCCCUGAUGAA----UGCACGUGUGUGU--AUAACUAAC-------CCAC--A---CACACACGGAAC .((((...(((((((......))).(((((((((((((...))))...)))))))))..)))----)))))((((((((--.........-------..))--)---)))))....... ( -33.10) >DroSim_CAF1 8467 107 + 1 GGUGCUCUGUUCGGUUUUUACACCAUAGGGGUUAAUUCCACGAGUCCAUGACCCCUGAUGAA----UGCACGUGUGUGU--AUAACUAACUA-GCCCCCAC--A---CACACACGGAAC .((((...(((((((......))).(((((((((((((...))))...)))))))))..)))----)))))((((((((--....((....)-).....))--)---)))))....... ( -32.80) >DroEre_CAF1 8813 102 + 1 GGUGCUCAGUUCCGUUUUUACCCCAUAGGGGUUAAU-CCACGAGUCCAUGACCCCUGAGGAA----UGCACGUGUGUGU--GUAACU----A-GCCCACAU--G---CACGCACGGAGC ........(((((((.......((.(((((((((..-...........))))))))).))..----....(((((((((--(.....----.-...)))))--)---)))).))))))) ( -35.72) >DroYak_CAF1 8662 102 + 1 GGUGCUCUUUUCCGUU-UUACCCCAUAGGGGUUAAUUCCACGAGGCCAUGACCCCUGAUGGA----UGCACGUGUGUGUGUGCAACU----A-GCCCACA-------CACACACGGGGC ...((((.........-.....((((((((((((...((....))...)))))))).)))).----....((((((((((((.....----.-...))))-------)))))))))))) ( -42.00) >DroAna_CAF1 8536 109 + 1 AGUGCUCC------UGUUACCACCAUAGGGGUUAAUUUGGUGAGUAAAUGACCCCCGACGUAGGCCUGCACGUGUGUGU--GCCAGUGAGUGUGCUUCUAU--AAAUCAUAAAACAAAU .(..((((------((.((((((....(((((((.((((.....)))))))))))..((((........))))))).))--).))).)))..)........--................ ( -26.60) >consensus GGUGCUCUGUUCGGUUUUUACACCAUAGGGGUUAAUUCCACGAGUCCAUGACCCCUGAUGAA____UGCACGUGUGUGU__AUAACUAAC_A_GCCCACAC__A___CACACACGGAAC .((((....((((((....))....(((((((((..............))))))))).)))).....))))(((((((.............................)))))))..... (-20.00 = -21.06 + 1.06)

| Location | 8,964,514 – 8,964,623 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 75.29 |
| Mean single sequence MFE | -36.40 |
| Consensus MFE | -18.11 |
| Energy contribution | -18.87 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.50 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941577 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
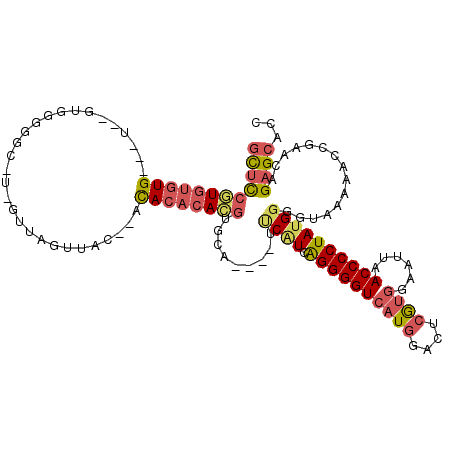
>2R_DroMel_CAF1 8964514 109 - 20766785 GCUCCAUGUGUG---UGUGUGUGGGU-UAGUUAGUUAU--AUACACAUGUGCA----UUCAUCAGGGGUCAUGGACUCGUGGAAUUAACCCCUAUGGGGUAAAAACCGAACAGAGCACC ((((((((((((---((((((.....-........)))--)))))))))))..----(((.(((.(((((...))))).))).....(((((...))))).......)))..))))... ( -35.62) >DroSec_CAF1 11845 101 - 1 GUUCCGUGUGUG---U--GUGG-------GUUAGUUAU--ACACACACGUGCA----UUCAUCAGGGGUCAUGGACUCGUGGAAUUAACCCCUAUGGUGUAAAAACCGAACAGAGCACC ((((((((((((---(--((((-------.....))))--)))))))))((((----(.(((.((((((((((....))))......))))))))))))))......))))........ ( -36.40) >DroSim_CAF1 8467 107 - 1 GUUCCGUGUGUG---U--GUGGGGGC-UAGUUAGUUAU--ACACACACGUGCA----UUCAUCAGGGGUCAUGGACUCGUGGAAUUAACCCCUAUGGUGUAAAAACCGAACAGAGCACC ((((.(..((((---(--(((..(((-(....))))..--.))))))))..).----(((...((((((((((....))))......))))))..(((......))))))..))))... ( -37.10) >DroEre_CAF1 8813 102 - 1 GCUCCGUGCGUG---C--AUGUGGGC-U----AGUUAC--ACACACACGUGCA----UUCCUCAGGGGUCAUGGACUCGUGG-AUUAACCCCUAUGGGGUAAAAACGGAACUGAGCACC ((((.(..((((---.--.((((...-.----.....)--)))..))))..).----(((((((.(((((...))))).)))-....(((((...)))))......))))..))))... ( -38.40) >DroYak_CAF1 8662 102 - 1 GCCCCGUGUGUG-------UGUGGGC-U----AGUUGCACACACACACGUGCA----UCCAUCAGGGGUCAUGGCCUCGUGGAAUUAACCCCUAUGGGGUAA-AACGGAAAAGAGCACC .....(((((((-------((((.(.-.----...).)))))))))))((((.----(((((..(((((....))))))))))....(((((...)))))..-...........)))). ( -44.10) >DroAna_CAF1 8536 109 - 1 AUUUGUUUUAUGAUUU--AUAGAAGCACACUCACUGGC--ACACACACGUGCAGGCCUACGUCGGGGGUCAUUUACUCACCAAAUUAACCCCUAUGGUGGUAACA------GGAGCACU ...((((((.......--...))))))..(((.(((((--((......))))..(((..(((.((((((.((((.......))))..)))))))))..)))..))------)))).... ( -26.80) >consensus GCUCCGUGUGUG___U__GUGGGGGC_U_GUUAGUUAC__ACACACACGUGCA____UUCAUCAGGGGUCAUGGACUCGUGGAAUUAACCCCUAUGGGGUAAAAACCGAACAGAGCACC ((((((((((((.............................)))))))).........((((.((((((((((....))))......))))))))))...............))))... (-18.11 = -18.87 + 0.76)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:32:01 2006