
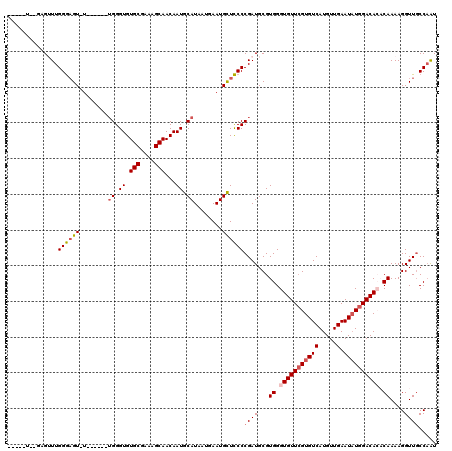
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 8,609,712 – 8,609,954 |
| Length | 242 |
| Max. P | 0.999352 |

| Location | 8,609,712 – 8,609,815 |
|---|---|
| Length | 103 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.31 |
| Mean single sequence MFE | -33.42 |
| Consensus MFE | -25.05 |
| Energy contribution | -25.97 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.540755 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609712 103 + 20766785 ----------GGUUGGGAGU-U------GGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGCUCCCCGAUGCGUGGGUGUUCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAU ----------.((((((.((-(------(((((((((....)).)))..((((.....)))).))))))).).((.(((((((((((.....).)))))))))).))........))))) ( -35.50) >DroSec_CAF1 10600 106 + 1 -----A--UUGGUUGGGAGU-U------UGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGCUCCCCGAUGCGUGGGUGUUCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAU -----(--(((((.((((((-.------((.((.(((....)))...)).))........)))))).......((.(((((((((((.....).)))))))))).)).......)))))) ( -32.80) >DroSim_CAF1 7443 106 + 1 -----C--UCGUUUGGGAGU-G------UGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGUUCCCCGAUGCGUGGGUGUUGGUGUCAUGUUGAAUCUGGACACACAAAAGGUUGCCAAU -----(--.((((((((..(-(------((.((.(((....)))...)).))))..((....))))))).))).)(((.((((((((..(......)..))))).)))......)))... ( -26.60) >DroEre_CAF1 9923 114 + 1 -GUUUU--GAGUUUGGGUGU-UGGGUGCUGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGCUCCCCGAUGCGUGGGUGUUCGUGUCAUGUUGAAUAUGGACA--CAAAAGGUUGCCAGU -.....--....((((((((-((((......((.(((....)))))...((((.....))))..))))))))....(((((((((((.....).)))))))))--).........)))). ( -33.90) >DroYak_CAF1 5175 118 + 1 GGUGUUGGGAGUUUGGAAGUUUGGGUGUUGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGCUUCCCGAUGCGUGGGUGUUCGUGUCAUGUUGAAUAUGGACA--CAAAAGGUUCCCUGU ......(((((..(..........(((((((((((((....)).)))..((((.....))))..))))))))....(((((((((((.....).)))))))))--)...)..)))))... ( -38.30) >consensus _____U__GAGUUUGGGAGU_U______UGGGUGUGCGAAAGCAACAAUGCAUAAUGAAUGCUCCCCGAUGCGUGGGUGUUCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAU ..............((((((........((.((.(((....)))...)).))........))))))((((...((.(((((((((((.....).)))))))))).))....))))..... (-25.05 = -25.97 + 0.92)


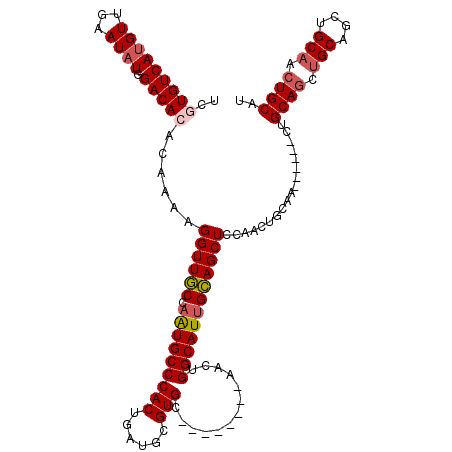
| Location | 8,609,775 – 8,609,881 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.10 |
| Mean single sequence MFE | -36.62 |
| Consensus MFE | -26.79 |
| Energy contribution | -27.43 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.871580 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609775 106 + 20766785 UCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAUGCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAACUGCAA------CUGCAGCUGCAACUGCAACUGCAU ..(((((((((...)))).)))))......((((((..((((........))))))--------)))).(((((((((...((.((((..------..)))).))...)))))).))).. ( -33.70) >DroSec_CAF1 10666 112 + 1 UCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAUGCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAACUGCAAAUGCGACUGCAGCUGCAGCUGCAACUGCAU ..(((((((((...)))).)))))......((((((..((((........))))))--------)))).(((((((((((.((.(((((........))))).)).)))))))).))).. ( -40.90) >DroSim_CAF1 7509 112 + 1 UGGUGUCAUGUUGAAUCUGGACACACAAAAGGUUGCCAAUGCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAAUUGCAAAUGCGACUGCAGGUGCAGCUGCAACUGCAA (((((((..(......)..))))).))...((((((..((((........))))))--------)))).(((((((((((.((.(((((........))))).)).)))))))).))).. ( -37.90) >DroEre_CAF1 9999 104 + 1 UCGUGUCAUGUUGAAUAUGGACA--CAAAAGGUUGCCAGUGCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCCACUGCAG------CUGCAGCUGCAGCUGCAACUGCAA ..(((((((((...)))).))))--).......(((((((...(((......))).--------.)))))))((((((.......(((((------((((....))))))))).)))))) ( -41.70) >DroYak_CAF1 5255 112 + 1 UCGUGUCAUGUUGAAUAUGGACA--CAAAAGGUUCCCUGUGCCCACUGAUGCGUGCAACUGUGCAACUGGCAUUGGAGCUCCAACUGCAA------CAGCAACAGCAACUGCAACUGCAU ..(((((((((...)))).))))--)....((((((..(((((....(.((((........)))).).))))).)))))).....((((.------..(((........)))...)))). ( -28.90) >consensus UCGUGUCAUGUUGAAUAUGGACACACAAAAGGUUGCCAAUGCCCACUGAUGCGUGC________AACUGGCAUUGCAGCUCCAACUGCAA______CUGCAGCUGCAGCUGCAACUGCAU ..(((((((((...)))).)))))......((((((.(((((((((......))).............))))))))))))..................((((.(((....))).)))).. (-26.79 = -27.43 + 0.64)

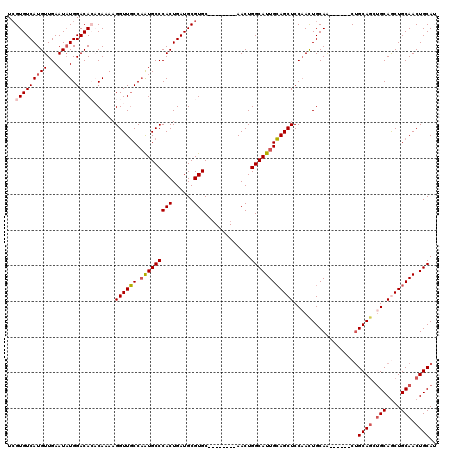
| Location | 8,609,775 – 8,609,881 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.10 |
| Mean single sequence MFE | -44.76 |
| Consensus MFE | -34.54 |
| Energy contribution | -35.78 |
| Covariance contribution | 1.24 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.79 |
| Structure conservation index | 0.77 |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.998217 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609775 106 - 20766785 AUGCAGUUGCAGUUGCAGCUGCAG------UUGCAGUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGCAUUGGCAACCUUUUGUGUGUCCAUAUUCAACAUGACACGA ..((((((((((((.(((((((..------..))))))).)))))))))((((((.--------((.(((....))).)))))))).......)))(((((............))))).. ( -44.80) >DroSec_CAF1 10666 112 - 1 AUGCAGUUGCAGCUGCAGCUGCAGUCGCAUUUGCAGUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGCAUUGGCAACCUUUUGUGUGUCCAUAUUCAACAUGACACGA ..((((((((((((.(((((((((......))))))))).)))))))))((((((.--------((.(((....))).)))))))).......)))(((((............))))).. ( -49.40) >DroSim_CAF1 7509 112 - 1 UUGCAGUUGCAGCUGCACCUGCAGUCGCAUUUGCAAUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGCAUUGGCAACCUUUUGUGUGUCCAGAUUCAACAUGACACCA ((((((((.(((.((((..(((....)))..)))).))).))))))))(((((((.--------((.(((....))).)))))))))......((.(((((............))))))) ( -41.50) >DroEre_CAF1 9999 104 - 1 UUGCAGUUGCAGCUGCAGCUGCAG------CUGCAGUGGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGCACUGGCAACCUUUUG--UGUCCAUAUUCAACAUGACACGA ((((((((.(.((((((((....)------))))))).).))))))))(((((((.--------((.(((....))).))))))))).....(((--((((............))))))) ( -49.80) >DroYak_CAF1 5255 112 - 1 AUGCAGUUGCAGUUGCUGUUGCUG------UUGCAGUUGGAGCUCCAAUGCCAGUUGCACAGUUGCACGCAUCAGUGGGCACAGGGAACCUUUUG--UGUCCAUAUUCAACAUGACACGA .((((((((..(.((((((.((((------(.(((..(((..........)))..)))))))).))).))).).((((((((((((....)))))--)))))))...)))).)).))... ( -38.30) >consensus AUGCAGUUGCAGCUGCAGCUGCAG______UUGCAGUUGGAGCUGCAAUGCCAGUU________GCACGCAUCAGUGGGCAUUGGCAACCUUUUGUGUGUCCAUAUUCAACAUGACACGA .....(((((((((.(((((((((......))))))))).)))))))))((((((.........((.(((....))).))))))))..........(((((............))))).. (-34.54 = -35.78 + 1.24)

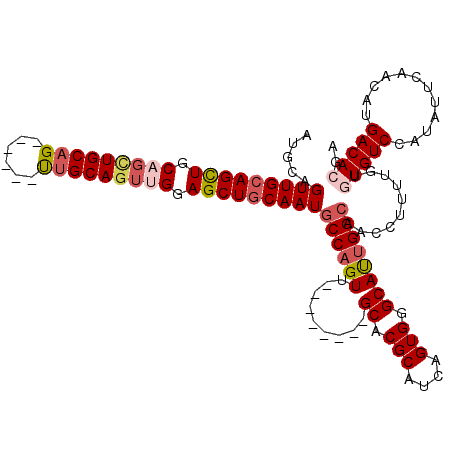

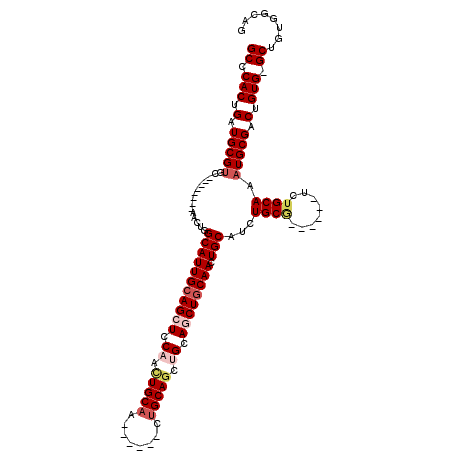
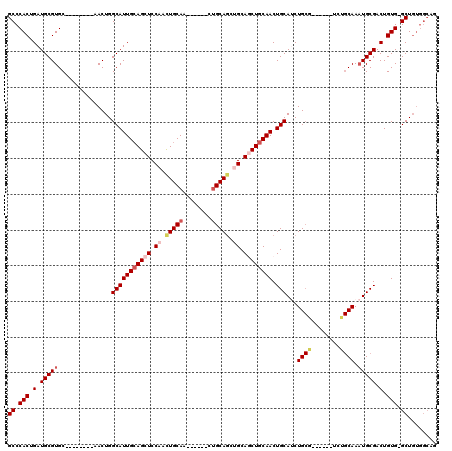
| Location | 8,609,815 – 8,609,914 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.95 |
| Mean single sequence MFE | -40.68 |
| Consensus MFE | -28.04 |
| Energy contribution | -29.74 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967081 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609815 99 + 20766785 GCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAACUGCAA------CUGCAGCUGCAACUGCAACUGCAUCUGCG------UCUGCAAAUGCGACUGUG-GCUGUGGCAU (((....(((((....--------.....)))))(((((..((..((((.------.(((((.((((..(((....)))..))))------.)))))..))))..))..-)))))))).. ( -39.00) >DroSec_CAF1 10706 105 + 1 GCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAACUGCAAAUGCGACUGCAGCUGCAGCUGCAACUGCAUCUGCG------UCUGCAAAUGCGACUGUG-GCUGUGGCAG ((........)).(((--------(..(.(((((((((......)))).))))).).))))((..((((..((..(((((.(((.------...))).)))))..))..-))))..)).. ( -43.40) >DroSim_CAF1 7549 112 + 1 GCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCAAUUGCAAAUGCGACUGCAGGUGCAGCUGCAACUGCAACUGCAUGGGGUUCUGCAAAUGCGACUGUGGGCUGUGGCAG ((((((.(.(((((((--------.....)).(((((((((((.((((....)))).(((((.(((((......))))).))))))))))..))))))))))).).))))))........ ( -48.60) >DroEre_CAF1 10037 86 + 1 GCCCACUGAUGCGUGC--------AACUGGCAUUGCAGCUCCCACUGCAG------CUGCAGCUGCAGCUGCAACUGCAACUG------------CAACUGCGACUGUG-GCU------- (((.((.(.(((.(((--------(....(((((((((((......((((------(....))))))))))))).)))...))------------))...))).).)))-)).------- ( -35.80) >DroYak_CAF1 5293 107 + 1 GCCCACUGAUGCGUGCAACUGUGCAACUGGCAUUGGAGCUCCAACUGCAA------CAGCAACAGCAACUGCAACUGCAUCUGCG------UCGGCAAAUGCGACUGUG-GCUGUGGCUG (((..(..((((.((((....))))....))))..).((.......)).(------((((.((((((..(((....)))..)))(------(((.(....)))))))).-)))))))).. ( -36.60) >consensus GCCCACUGAUGCGUGC________AACUGGCAUUGCAGCUCCAACUGCAA______CUGCAGCUGCAGCUGCAACUGCAUCUGCG______UCUGCAAAUGCGACUGUG_GCUGUGGCAG ((.(((.(.(((((...............(((((((((((.((.(((((........))))).)).)))))))).)))...((((........)))).))))).).))).))........ (-28.04 = -29.74 + 1.70)

| Location | 8,609,815 – 8,609,914 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.95 |
| Mean single sequence MFE | -46.68 |
| Consensus MFE | -36.47 |
| Energy contribution | -37.16 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.13 |
| Mean z-score | -3.83 |
| Structure conservation index | 0.78 |
| SVM decision value | 3.53 |
| SVM RNA-class probability | 0.999352 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609815 99 - 20766785 AUGCCACAGC-CACAGUCGCAUUUGCAGA------CGCAGAUGCAGUUGCAGUUGCAGCUGCAG------UUGCAGUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGC ..(((...((-.((....((((((((...------.)))))))).(((((((((.(((((((..------..))))))).)))))))))....)).--------)).(((....)))))) ( -46.60) >DroSec_CAF1 10706 105 - 1 CUGCCACAGC-CACAGUCGCAUUUGCAGA------CGCAGAUGCAGUUGCAGCUGCAGCUGCAGUCGCAUUUGCAGUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGC ..(((...((-.((....((((((((...------.)))))))).(((((((((.(((((((((......))))))))).)))))))))....)).--------)).(((....)))))) ( -51.20) >DroSim_CAF1 7549 112 - 1 CUGCCACAGCCCACAGUCGCAUUUGCAGAACCCCAUGCAGUUGCAGUUGCAGCUGCACCUGCAGUCGCAUUUGCAAUUGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGC ........((((((....(((..(((.((......(((((.((((((....)))))).))))).)))))..)))...((..((((((((....)))--------))).))..)))))))) ( -47.00) >DroEre_CAF1 10037 86 - 1 -------AGC-CACAGUCGCAGUUG------------CAGUUGCAGUUGCAGCUGCAGCUGCAG------CUGCAGUGGGAGCUGCAAUGCCAGUU--------GCACGCAUCAGUGGGC -------..(-(((....(((((((------------((((((((((....)))))))))))))------)))).....((((((((((....)))--------))).)).)).)))).. ( -48.30) >DroYak_CAF1 5293 107 - 1 CAGCCACAGC-CACAGUCGCAUUUGCCGA------CGCAGAUGCAGUUGCAGUUGCUGUUGCUG------UUGCAGUUGGAGCUCCAAUGCCAGUUGCACAGUUGCACGCAUCAGUGGGC ...(((((((-..(((..((((((((...------.))))))))..)))..)))(((((.((((------(.(((..(((..........)))..)))))))).))).))....)))).. ( -40.30) >consensus CUGCCACAGC_CACAGUCGCAUUUGCAGA______CGCAGAUGCAGUUGCAGCUGCAGCUGCAG______UUGCAGUUGGAGCUGCAAUGCCAGUU________GCACGCAUCAGUGGGC ........((.(((....((((((((..........)))))))).(((((((((.(((((((((......))))))))).))))))))).........................))).)) (-36.47 = -37.16 + 0.69)

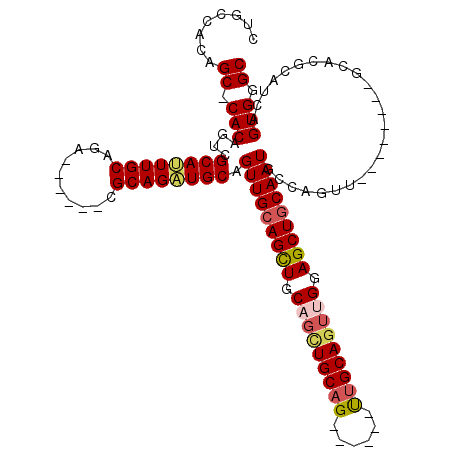
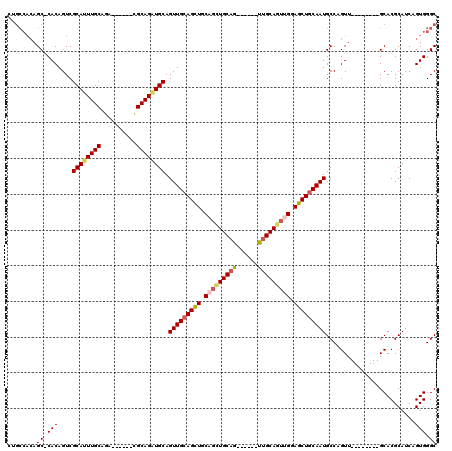
| Location | 8,609,847 – 8,609,954 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.19 |
| Mean single sequence MFE | -40.98 |
| Consensus MFE | -28.28 |
| Energy contribution | -29.12 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.738395 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8609847 107 - 20766785 CUGGACGGCAGAUGGAAUUGCUCAGUCAGUCACACAGGCGAUGCCACAGC-CACAGUCGCAUUUGCAGA------CGCAGAUGCAGUUGCAGUUGCAGCUGCAG------UUGCAGUUGG ...((((((.(.(((.((((((..((.....))...)))))).)))).))-)...(((((....)).))------)((((.(((((((((....))))))))).------)))).))).. ( -39.90) >DroSec_CAF1 10738 113 - 1 CUGGACGGCAGAUGGAAUUGCUCACUCAGUCACACAGGCGCUGCCACAGC-CACAGUCGCAUUUGCAGA------CGCAGAUGCAGUUGCAGCUGCAGCUGCAGUCGCAUUUGCAGUUGG .(((.((((.(((((......))).)).(((.....))))))))))((((-....(((((....)).))------)((((((((..(((((((....)))))))..)))))))).)))). ( -45.40) >DroSim_CAF1 7581 120 - 1 CUGGACGGCAGAUGGAAUUGCUCACUCAGUCACACAGGCUCUGCCACAGCCCACAGUCGCAUUUGCAGAACCCCAUGCAGUUGCAGUUGCAGCUGCACCUGCAGUCGCAUUUGCAAUUGG (((...((((((((..((((......))))..)).....)))))).)))....((((.(((..(((.((......(((((.((((((....)))))).))))).)))))..))).)))). ( -41.00) >DroEre_CAF1 10069 89 - 1 CUGGACGGCAGAUGGAAUUGCUCACUCAGUCACAC------------AGC-CACAGUCGCAGUUG------------CAGUUGCAGUUGCAGCUGCAGCUGCAG------CUGCAGUGGG ((((..(((((......)))))...))))......------------..(-(((....(((((((------------((((((((((....)))))))))))))------)))).)))). ( -41.70) >DroYak_CAF1 5333 107 - 1 CUGGCCGGCAGAUGGAAUUGCUCACUCAGUCACACAGGCGCAGCCACAGC-CACAGUCGCAUUUGCCGA------CGCAGAUGCAGUUGCAGUUGCUGUUGCUG------UUGCAGUUGG ....(((((...((..((((......))))..))...(((((((.(((((-..(((..((((((((...------.))))))))..))).....))))).))))------.))).))))) ( -36.90) >consensus CUGGACGGCAGAUGGAAUUGCUCACUCAGUCACACAGGCGCUGCCACAGC_CACAGUCGCAUUUGCAGA______CGCAGAUGCAGUUGCAGCUGCAGCUGCAG______UUGCAGUUGG ((((..(((((......)))))...)))).......(((...))).((((...(((..((((((((..........))))))))..)))..))))(((((((((......))))))))). (-28.28 = -29.12 + 0.84)

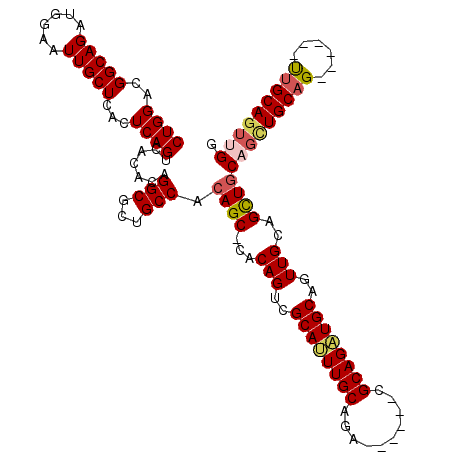
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:28:21 2006