
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 8,394,381 – 8,394,517 |
| Length | 136 |
| Max. P | 0.991053 |

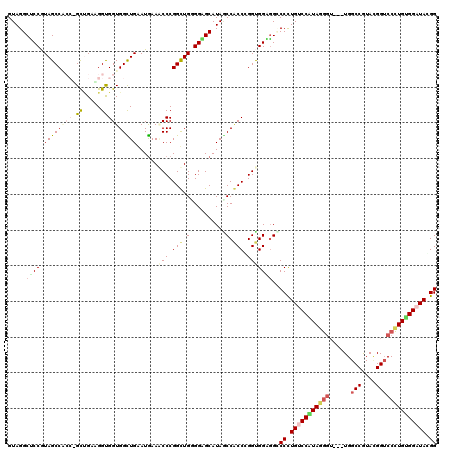
| Location | 8,394,381 – 8,394,483 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.62 |
| Mean single sequence MFE | -57.30 |
| Consensus MFE | -33.80 |
| Energy contribution | -34.93 |
| Covariance contribution | 1.13 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.77 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.25 |
| SVM RNA-class probability | 0.991053 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
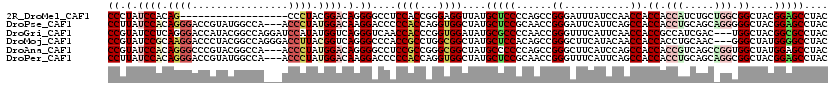
>2R_DroMel_CAF1 8394381 102 + 20766785 GUAGGCUCCGUAGCCGCCAGCAGAUGGUGGUGGUUGGAUAAAUCCCGGCUGGGGAGCAUAACCUCCCGGUGGAGGCCCCUGUCCGUAGGG------------------CUGUGGAUAGGG ....(((((.(((((((((.....))))((.((((.....))))))))))).)))))....(((((....))))).(((((((((((...------------------.))))))))))) ( -50.10) >DroPse_CAF1 5540 117 + 1 GUAGGCUCCGUAGCCCCCUGCUGCAGGUGGUGGCUGAAUGAAUCCCGGUUGCGGAGCAUAGCCACCUGGUGGGGGUCCUUGUCCAUAGGGU---UGGCCAUACGGUCCCUGUGGAUAAGG ...((((....))))(((..(..((((((((.(((((.....))(((....))))))...)))))))))..)))..((((((((((((((.---..(((....))))))))))))))))) ( -58.40) >DroGri_CAF1 10135 117 + 1 GUAGGCGCCGUAGCCA---GUCGAUGGCGGUGGUUGAAUGAAACCCGGUUGGGGCGCAUAUCCACCGGGUGGUUGACCCUGACCAUAUGGAUCCUGGCCGUAUGGUCCCUGAGGAUACGG ....(((((.(((((.---((.....)).(.((((......)))))))))).))))).(((((...((((.....)))).((((((((((.......)))))))))).....)))))... ( -49.90) >DroMoj_CAF1 10034 117 + 1 GUAGGCCCCAUAGCCC---GUUGCAGGUGGUGGUUGUAUGAAGCCCGGCUGUGGAGCAUAGCCGCCAGGCGGUGGGCCCUGACCGUAAGGUCCCUGGCCGUAGGGUCCUUGCGGAUACGG .((((.(((((.((((---......).((((((((......)))).(((((((...)))))))))))))).))))).)))).((((((((.(((((....))))).))))))))...... ( -56.30) >DroAna_CAF1 6094 117 + 1 GUAGGCUCCAUAGCCACCGGCUGACGGUGGUGGCUGGAUGAAGCCCGGCUGGGGGGCAUAGCCGCCCGGCGGAGGCCCCUGUCCAUAGGGU---UGGCCGUACGGGCCCUGUGGAUACGG ...((((((...(((((((.....)))))))((((......))))..((((((.(((...))).))))))))).)))((((((((((((((---(.(.....).))))))))))))).)) ( -68.00) >DroPer_CAF1 5545 117 + 1 GUAGGCUCCGUAGCCGCCUGCUGCAGGUGGUGGCUGAAUGAAACCCGGUUGCGGAGCAUAGCCACCUGGUGGGGGUCCUUGUCCAUAGGGU---UGGCCAUACGGUCCCUGUGGAUAAGG ....((((((((((((((((...)))))(((..(.....)..))).)))))))))))...(((.((....)).)))((((((((((((((.---..(((....))))))))))))))))) ( -61.10) >consensus GUAGGCUCCGUAGCCACC_GCUGAAGGUGGUGGCUGAAUGAAACCCGGCUGGGGAGCAUAGCCACCCGGUGGAGGCCCCUGUCCAUAGGGU___UGGCCGUACGGUCCCUGUGGAUACGG .....((((.((((((((.((.....))))))))))......(((.(((.((........)).))).)))))))..((.(((((((((((......(((....)))))))))))))).)) (-33.80 = -34.93 + 1.13)


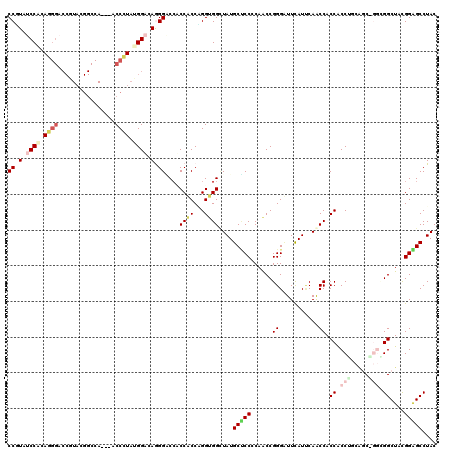
| Location | 8,394,381 – 8,394,483 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.62 |
| Mean single sequence MFE | -44.15 |
| Consensus MFE | -20.24 |
| Energy contribution | -21.44 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.600297 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8394381 102 - 20766785 CCCUAUCCACAG------------------CCCUACGGACAGGGGCCUCCACCGGGAGGUUAUGCUCCCCAGCCGGGAUUUAUCCAACCACCACCAUCUGCUGGCGGCUACGGAGCCUAC ((((.(((....------------------......))).))))((((((....))))))...(((((..((((((((....)))..(((.((.....)).))))))))..))))).... ( -40.30) >DroPse_CAF1 5540 117 - 1 CCUUAUCCACAGGGACCGUAUGGCCA---ACCCUAUGGACAAGGACCCCCACCAGGUGGCUAUGCUCCGCAACCGGGAUUCAUUCAGCCACCACCUGCAGCAGGGGGCUACGGAGCCUAC ((((.((((.((((.((....))...---.)))).)))).))))..........((((((((((.((((....))))...)))..))))))).((((...))))(((((....))))).. ( -42.70) >DroGri_CAF1 10135 117 - 1 CCGUAUCCUCAGGGACCAUACGGCCAGGAUCCAUAUGGUCAGGGUCAACCACCCGGUGGAUAUGCGCCCCAACCGGGUUUCAUUCAACCACCGCCAUCGAC---UGGCUACGGCGCCUAC ..((((((.(...(((((((.((.......)).))))))).((((.....)))).).))))))(((((.......((((......))))...((((.....---))))...))))).... ( -41.10) >DroMoj_CAF1 10034 117 - 1 CCGUAUCCGCAAGGACCCUACGGCCAGGGACCUUACGGUCAGGGCCCACCGCCUGGCGGCUAUGCUCCACAGCCGGGCUUCAUACAACCACCACCUGCAAC---GGGCUAUGGGGCCUAC .(((((((....)))...))))((((((((((....)))).((.....)).))))))((((.((...)).))))((((((((((...((............---))..)))))))))).. ( -48.40) >DroAna_CAF1 6094 117 - 1 CCGUAUCCACAGGGCCCGUACGGCCA---ACCCUAUGGACAGGGGCCUCCGCCGGGCGGCUAUGCCCCCCAGCCGGGCUUCAUCCAGCCACCACCGUCAGCCGGUGGCUAUGGAGCCUAC ((...((((.((((.((....))...---.)))).))))..))(((....)))(((.(((...))).)))....(((((((((..(((((((..(....)..)))))))))))))))).. ( -51.80) >DroPer_CAF1 5545 117 - 1 CCUUAUCCACAGGGACCGUAUGGCCA---ACCCUAUGGACAAGGACCCCCACCAGGUGGCUAUGCUCCGCAACCGGGUUUCAUUCAGCCACCACCUGCAGCAGGCGGCUACGGAGCCUAC ((((.((((.((((.((....))...---.)))).)))).))))..........((((((((((.((((....))))...)))..))))))).((((...)))).((((....))))... ( -40.60) >consensus CCGUAUCCACAGGGACCGUACGGCCA___ACCCUAUGGACAGGGACCACCACCAGGUGGCUAUGCUCCCCAACCGGGAUUCAUUCAACCACCACCUGCAGC_GGCGGCUACGGAGCCUAC ((.(.((((.((((................)))).)))).).))....((((...))))....(((((......((...........)).((.(((.....))).))....))))).... (-20.24 = -21.44 + 1.20)

| Location | 8,394,421 – 8,394,517 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.34 |
| Mean single sequence MFE | -53.70 |
| Consensus MFE | -29.25 |
| Energy contribution | -29.26 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948131 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
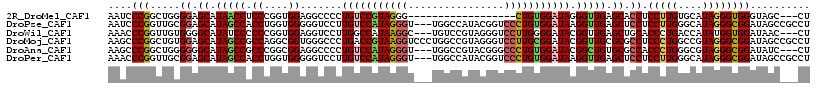
>2R_DroMel_CAF1 8394421 96 + 20766785 AAUCCCGGCUGGGGAGCAUAACCUCCCGGUGGAGGCCCCUGUCCGUAGGG------------------CUGUGGAUAGGGUUGAGCACCUCCUUGUGCAUAGGGUGGGUAGC---CU ..(((..((((((..(.....)..)))))))))((((((((((((((...------------------.)))))))))))....((.((.(((((....))))).)))).))---). ( -39.30) >DroPse_CAF1 5580 114 + 1 AAUCCCGGUUGCGGAGCAUAGCCACCUGGUGGGGGUCCUUGUCCAUAGGGU---UGGCCAUACGGUCCCUGUGGAUAAGGUUGAGCUCCUCCUUGGGCAUAGGGCGGAUAGCCGCCU ...(((((..(.(((((...(((.((....)).)))((((((((((((((.---..(((....)))))))))))))))))....))))).).)))))....((((((....)))))) ( -55.80) >DroWil_CAF1 4969 111 + 1 AAACCCGGUUGUGGGGCAUAUCCCCCCGGUGGAGGUCCUUGGCCAUAAGGC---UGUCCGUAGGGUCCUUGGGGAUACGGUUGAGCUGCACCCUGACCAUAUGGUGGAUAAC---CU ....((((....((((.....)))))))).((..((((..((((....)))---)..(((((.((((...((((...(((.....)))..)))))))).))))).))))..)---). ( -41.00) >DroMoj_CAF1 10071 117 + 1 AAGCCCGGCUGUGGAGCAUAGCCGCCAGGCGGUGGGCCCUGACCGUAAGGUCCCUGGCCGUAGGGUCCUUGCGGAUACGGUUGCGCGCCUCCCUGGCCGUAGGGCGGAUAGCCGCCU ..(((((((((((...)))))))((((((.((((.((((((.((((((((.(((((....))))).)))))))).)).))..)).))))..))))))....))))((....)).... ( -64.80) >DroAna_CAF1 6134 111 + 1 AAGCCCGGCUGGGGGGCAUAGCCGCCCGGCGGAGGCCCCUGUCCAUAGGGU---UGGCCGUACGGGCCCUGUGGAUACGGCUGUGCGCCACCCUGGGCGUAGGGCGGAUAUC---CU ..(((((((((((((((...(((....)))....))))))(((((((((((---(.(.....).)))))))))))).))))).((((((......))))))))))((....)---). ( -65.10) >DroPer_CAF1 5585 114 + 1 AAACCCGGUUGCGGAGCAUAGCCACCUGGUGGGGGUCCUUGUCCAUAGGGU---UGGCCAUACGGUCCCUGUGGAUAAGGUUGAGCUCCUCCUUGGGCAUAGGGCGGAUAGCCGCCU ...(((((..(.(((((...(((.((....)).)))((((((((((((((.---..(((....)))))))))))))))))....))))).).)))))....((((((....)))))) ( -56.20) >consensus AAACCCGGCUGCGGAGCAUAGCCACCCGGUGGAGGUCCCUGUCCAUAGGGU___UGGCCGUACGGUCCCUGUGGAUACGGUUGAGCUCCUCCCUGGGCAUAGGGCGGAUAGC___CU ....(((.....((.((.(((((.((....)).......(((((((((((................))))))))))).))))).)).)).(((((....)))))))).......... (-29.25 = -29.26 + 0.00)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:25:28 2006