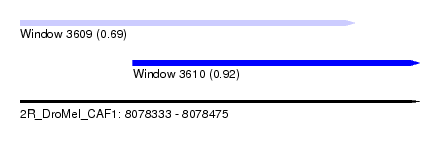
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 8,078,333 – 8,078,475 |
| Length | 142 |
| Max. P | 0.918263 |

| Location | 8,078,333 – 8,078,452 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.75 |
| Mean single sequence MFE | -50.75 |
| Consensus MFE | -37.24 |
| Energy contribution | -37.83 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.692840 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
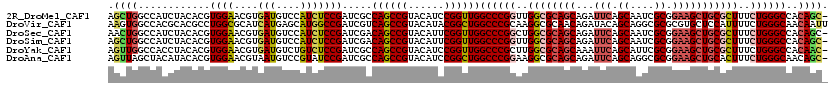
>2R_DroMel_CAF1 8078333 119 + 20766785 AGCUGGCCAUCUACACGUGGAACGUGAUGUCCAUCUCCGAUCGCCAGCCGUACAUCCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGC- .((((((.(((.....(((((........)))))....))).))))))............((((((((..((((((((...(((.((....)).)))))))))))..))))))))....- ( -54.20) >DroVir_CAF1 31991 120 + 1 AAGUGGCCACGCACGCCUGGCGCAUCAUGAGCAUGGCCGAUCGUCAGCCGUACAUACGGCUGGCCCGCAAGGCGCAACAGAUACAGCAGGCGCGCGUGCUCCAUUUUCUGGGCAACAAUU ((((((.(((((.((((((((.((((....).)))))).(((..(((((((....)))))))((((....)).))....))).....))))).)))))..))))))....(....).... ( -50.60) >DroSec_CAF1 43204 119 + 1 AACUGGCCAUCUACACGUGGAACGUGAUGUCCAUCUCCGAUCGACAGCCGUACAUUCGGUUGGCCCGGCUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGC- ..((((((.((((....))))(((.(.((((.(((...))).)))).))))......)))((((((((..((((((((...(((.((....)).)))))))))))..))))))))))).- ( -49.80) >DroSim_CAF1 25666 119 + 1 AGCUGGCCAUCUACACGUGGAACGUGAUGUCCAUCUCCGAUCGACAGCCGUACAUUCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGC- .(((((((.((((....))))(((.(.((((.(((...))).)))).))))......)))((((((((..((((((((...(((.((....)).)))))))))))..))))))))))))- ( -53.30) >DroYak_CAF1 41650 119 + 1 AGUUGGCCACCUACACGUGGAACGUGAUGUCUGUCUCCGAUCGCCAGCCGUACAUCCGGUUGGCCCGCUUGGCGCAGCAAAUUCAGCAUUCGCGGAAGCUGCGCUUUCUGGGCCACAAC- .((.((((.....((.((((..((.(((....)))..))...((((((((......)))))))))))).))(((((((...(((.((....)).))))))))))......))))))...- ( -48.30) >DroAna_CAF1 42059 119 + 1 AGUUAGCUACAUACACGUGGAACGUAAUGUCCGUAUCCGAUCGCCAGCCGUACAUCCGGCUGGCCCGGAAGGCGCAGCAGAUUCAGCAGGCGCGGAAGCUGCACUUUCUGGGCAACAGC- .(((.((..........(((((((.......))).))))...((((((((......))))))))((((((((.(((((...(((.((....)).)))))))).)))))))))))))...- ( -48.30) >consensus AGCUGGCCACCUACACGUGGAACGUGAUGUCCAUCUCCGAUCGCCAGCCGUACAUCCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGC_ .((((............((((....(((....))))))).....((((((......))))))((((((..((((((((...(((.((....)).)))))))))))..))))))..)))). (-37.24 = -37.83 + 0.59)
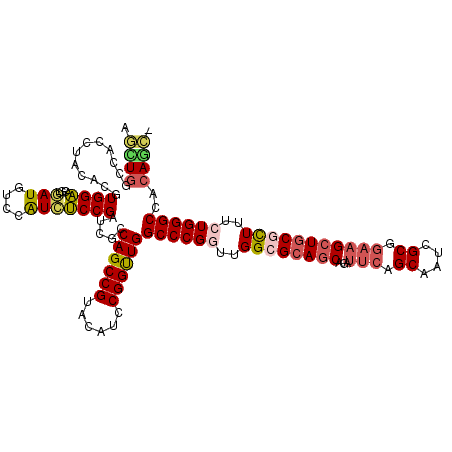
| Location | 8,078,373 – 8,078,475 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 89.54 |
| Mean single sequence MFE | -47.65 |
| Consensus MFE | -38.53 |
| Energy contribution | -39.12 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918263 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 8078373 102 + 20766785 UCGCCAGCCGUACAUCCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGCAGUCGGUCGGUGACCAAGAGGAA (((((.((((.((.....((.(((((((..((((((((...(((.((....)).)))))))))))..)))))))))....)))))).)))))((....)).. ( -50.90) >DroSec_CAF1 43244 102 + 1 UCGACAGCCGUACAUUCGGUUGGCCCGGCUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGCAGUCGGUCGGUGAACAAAAGGAA ((((((((((......)))))(((((((..((((((((...(((.((....)).)))))))))))..)))))))......)))))................. ( -46.10) >DroSim_CAF1 25706 102 + 1 UCGACAGCCGUACAUUCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGCAGUCGGUCGGUGAACAAAAGGAA ((((((((((......)))))(((((((..((((((((...(((.((....)).)))))))))))..)))))))......)))))................. ( -46.90) >DroEre_CAF1 41717 102 + 1 UCGCCAGCCGUACAUCCGGUUGGCCCGGUUGGCGCAGAAGAUUCAGCAAUGGCGGAGGCUGCGCUUUCUGGGCCGCAGCAGUCGGUCGGGGACCAAGCGCAA ..((.(((((......)))))(((((((..(((((((....(((.((....)).))).)))))))..))))))))).((.((.((((...))))..)))).. ( -46.00) >DroYak_CAF1 41690 102 + 1 UCGCCAGCCGUACAUCCGGUUGGCCCGCUUGGCGCAGCAAAUUCAGCAUUCGCGGAAGCUGCGCUUUCUGGGCCACAACGGUCGGUCGGUGAACAAGAGGAG (((((.((((.....(((..(((((((...((((((((...(((.((....)).)))))))))))...)))))))...))).)))).))))).......... ( -49.70) >DroAna_CAF1 42099 102 + 1 UCGCCAGCCGUACAUCCGGCUGGCCCGGAAGGCGCAGCAGAUUCAGCAGGCGCGGAAGCUGCACUUUCUGGGCAACAGCAAUCGGCCGUUGACCAAAAGAAU ..((((((((......))))))))((((((((.(((((...(((.((....)).)))))))).)))))))).((((.((.....)).))))........... ( -46.30) >consensus UCGCCAGCCGUACAUCCGGUUGGCCCGGUUGGCGCAGCAGAUUCAGCAAUCGCGGAAGCUGCGCUUUCUGGGCCACAGCAGUCGGUCGGUGAACAAAAGGAA (((.(.((((.((.....(.((((((((..((((((((...(((.((....)).)))))))))))..)))))))))....)))))).).))).......... (-38.53 = -39.12 + 0.59)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:23:23 2006