
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,954,391 – 7,954,536 |
| Length | 145 |
| Max. P | 0.999082 |

| Location | 7,954,391 – 7,954,496 |
|---|---|
| Length | 105 |
| Sequences | 5 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 93.71 |
| Mean single sequence MFE | -29.82 |
| Consensus MFE | -27.72 |
| Energy contribution | -28.12 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.36 |
| SVM RNA-class probability | 0.999082 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7954391 105 - 20766785 UAUUCGCUGAGCUAAUCAUUUCACCGACUGCCAUAUGGCGGUAUGUGAUCUAAUCUCAAUGAGCAAAAGGUCUAGUUAUUAAGGCAUGCGGCUCAGUUUGAUAUG ((((.((((((((..(((((((((..((((((....))))))..)))).........)))))(((....((((........)))).)))))))))))..)))).. ( -32.80) >DroSec_CAF1 6127 105 - 1 UAUUCGCUGAGCUAAUCAUUUCACCGACUGCCAUAUGGCGGUAUGUGAUCUAAUCUCAAUGAGCAAAAGGUCUAUUAAUUAAGGCACGCGGCUCAGUUUGAUAUG ((((.((((((((..(((((((((..((((((....))))))..)))).........)))))((.....((((........))))..))))))))))..)))).. ( -31.00) >DroSim_CAF1 6112 105 - 1 UAUUCGCUGAGCUAAUCAUUUCACCGACUGCCAUAUGGCGGUAUGUGAUCUAAUCUCAAUGAGCAAAAGGUCUAUUAAUUAAGGCACGCGGCUCAGUUUGAUAUG ((((.((((((((..(((((((((..((((((....))))))..)))).........)))))((.....((((........))))..))))))))))..)))).. ( -31.00) >DroEre_CAF1 6206 105 - 1 UAUUCGCUGAGCUAAUCAUUUCACCGAUUGCCAUAUGGCGGUAAGUGAUCUAAUCUCAAUGAGCAAACGGUCUGUUAAUUAAAAAAUGCGGUUCAGUUUGAUAUG ((((.((((((((..(((((((((..((((((....))))))..)))).........)))))(((.....................)))))))))))..)))).. ( -24.00) >DroYak_CAF1 6212 105 - 1 UAUUCGCUGAGCUAAUCACUUCGCCGAUUGCCAUAUGGCGGUAUGUGAUCUAAUCUCAAUGAGCAAAAGGUCUGUUAAUUAAGAAAUGCGGCUCAGUGUGAUAUG (((((((((((((.(((((.((((((.........))))))...))))).............(((.....(((........)))..)))))))))))).)))).. ( -30.30) >consensus UAUUCGCUGAGCUAAUCAUUUCACCGACUGCCAUAUGGCGGUAUGUGAUCUAAUCUCAAUGAGCAAAAGGUCUAUUAAUUAAGGCAUGCGGCUCAGUUUGAUAUG .....((((((((..(((((((((..((((((....))))))..)))).........)))))(((....((((........)))).)))))))))))........ (-27.72 = -28.12 + 0.40)

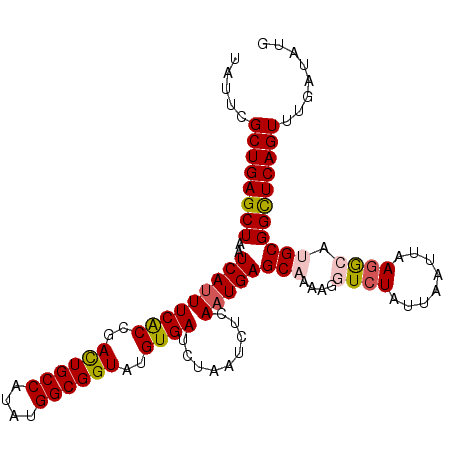

| Location | 7,954,431 – 7,954,536 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.15 |
| Mean single sequence MFE | -28.43 |
| Consensus MFE | -20.88 |
| Energy contribution | -20.69 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.943346 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7954431 105 - 20766785 GUGUGUGUGUGUGUGACUUUUCAUUCGCACGACGCUUAAUUAUUCGCUGAGCUAAUCAUUUCACC--------GACUGCCAUAUGGCGGUAU-------GUGAUCUAAUCUCAAUGAGCA ((((.((((((.((((....)))).)))))))))).......((((.((((.........((((.--------.((((((....))))))..-------))))......)))).)))).. ( -32.26) >DroSec_CAF1 6167 101 - 1 GUGUG----UGUGUGACUUUUCAUUCGCACGACGCUCAAUUAUUCGCUGAGCUAAUCAUUUCACC--------GACUGCCAUAUGGCGGUAU-------GUGAUCUAAUCUCAAUGAGCA (((((----((.((((....)))).))))).))(((((.........(((.....)))..((((.--------.((((((....))))))..-------))))...........))))). ( -31.60) >DroSim_CAF1 6152 103 - 1 GUGUGU--GUGUGUGACUUUUCAUUCGCACCAAGCUCAAUUAUUCGCUGAGCUAAUCAUUUCACC--------GACUGCCAUAUGGCGGUAU-------GUGAUCUAAUCUCAAUGAGCA .((.((--(((.((((....)))).))))))).(((((.........(((.....)))..((((.--------.((((((....))))))..-------))))...........))))). ( -31.40) >DroEre_CAF1 6246 95 - 1 --------GAGUGU--CUUUUCAUUCGCACGGCGCUCAAUUAUUCGCUGAGCUAAUCAUUUCACC--------GAUUGCCAUAUGGCGGUAA-------GUGAUCUAAUCUCAAUGAGCA --------(((((.--.....)))))((.....(((((.........)))))...(((((((((.--------.((((((....))))))..-------)))).........))))))). ( -25.90) >DroYak_CAF1 6252 104 - 1 G-GUAGCUGAGUGUGACUUUUCAUUCGCACGGCGCUCAAUUAUUCGCUGAGCUAAUCACUUCGCC--------GAUUGCCAUAUGGCGGUAU-------GUGAUCUAAUCUCAAUGAGCA .-...(((..((((((........)))))))))(((((.........((((...(((((.(((((--------(.........))))))...-------))))).....)))).))))). ( -28.20) >DroAna_CAF1 5920 111 - 1 ----CCAAGUGUGUGACUUUUAAUUAGCUUGAAAUUCAAU-----AUUGAAUUAAUCACAGUGCCCGCUGUCGCACUGCCAUAUGGCAGUCUAGUAUCUCCGAUCUAAUCUUAAAUAGUG ----.(((((..((........))..))))).((((((..-----..))))))....(((((....)))))(((((((((....)))))).(((.(((...))))))..........))) ( -21.20) >consensus G_GUG___GUGUGUGACUUUUCAUUCGCACGACGCUCAAUUAUUCGCUGAGCUAAUCAUUUCACC________GACUGCCAUAUGGCGGUAU_______GUGAUCUAAUCUCAAUGAGCA ..........((((((........))))))...(((((.........(((.....)))..((((..........((((((....)))))).........))))...........))))). (-20.88 = -20.69 + -0.19)

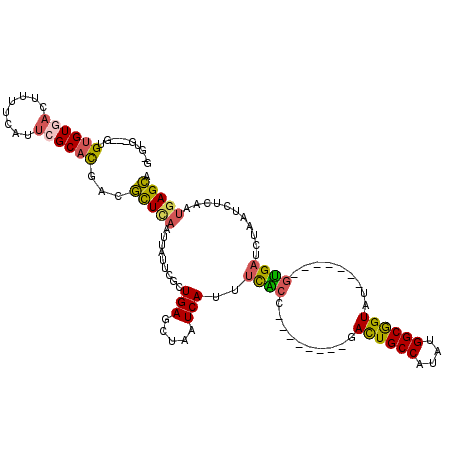
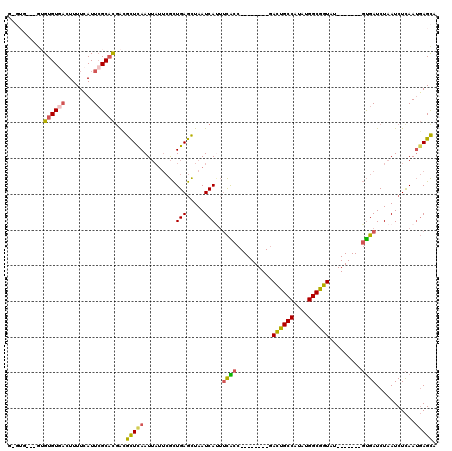
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:22:03 2006