
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,741,826 – 7,741,958 |
| Length | 132 |
| Max. P | 0.897156 |

| Location | 7,741,826 – 7,741,935 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 74.64 |
| Mean single sequence MFE | -42.70 |
| Consensus MFE | -28.07 |
| Energy contribution | -27.93 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.05 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.521343 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
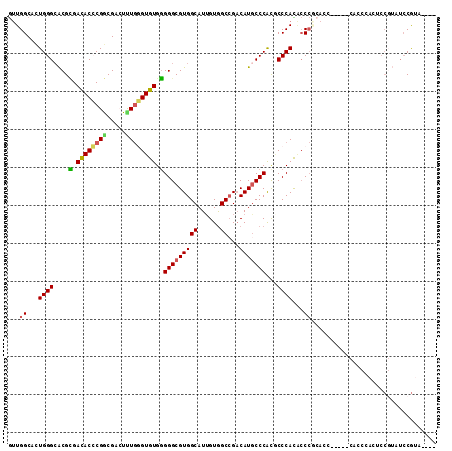
>2R_DroMel_CAF1 7741826 109 + 20766785 GUUGGCACUGGGCACGCGACACCCGGCGACUUUGGGUGUGGGGGCGUGGCAUUGUGGCCGACAUGCCCACACCCAUACCCGCACCGGCGGCACCCACUCCGUAUCCGUA---- ...((...((((..(((........)))....((((((((((.(.(((((......))).)).).))))))))))...((((....))))..))))..)).........---- ( -42.90) >DroSec_CAF1 31825 106 + 1 GUUGGCACUGGGCACGCGACACCCGGCGACUUUGGGUGUGGGGGCGUGGCAUUGUGGCCGACAUGCCCACACCCACACCCGCACAU---GCACCCACUCCGUAUCCGUA---- ...((...((((..(((........))).....(((((((((.(.(.(((((.((.....)))))))).).)))))))))((....---)).))))..)).........---- ( -40.10) >DroEre_CAF1 25269 106 + 1 GUUGGCACUGGGCACGCGACACCCGGCGACUUUGGGUGUGGGGGCGUGGCAUUGUGGCCGACAUGCCCACGCCCACGCCCGCACCC---CCAACCACACCGUAUCCGUA---- (((((...(((((...(.((((((((.....)))))))).)(((((((((((.((.....))))).))))))))..))))).....---)))))...............---- ( -46.30) >DroYak_CAF1 25541 110 + 1 GUUGGCACUGGGCACGCGACACCCGGCGACUCUGGGUGUGGGGGCGUGGCAUUGUGGCCGACAUGCCCACGCCCACACCCACACCC---ACACCUCCACCGUAUCCGUAUCCG .........((..(((.((....(((.(....((((((((((((((((((((.((.....))))).)))))))....)))))))))---)......).)))..)))))..)). ( -45.70) >DroAna_CAF1 23734 82 + 1 AUUGGCACUGGGCGCGGGACACGCGACGCGUUUCAGUGUGUGGGCGUGGCAUUGUGGCCGACAUACCCGC-CCCAUAGC--------------------------CAUA---- ..((((..((((.((((((.((((...)))).)).((((((.(((...........))).))))))))))-))))..))--------------------------))..---- ( -38.40) >DroPer_CAF1 23912 94 + 1 GCUGGCAUUGGGAGCGUGAUACGCGACGCGUCUGUGUGUGGGGGCGUGGCAUGGUAGCAGCCAUGCCCAUGCCCAUUCCC---------------AUUCCCAUUCCGUA---- ..(((...((((((..(.((((((((....)).)))))).)((((((((((((((....)))))).)))))))).)))))---------------)...))).......---- ( -42.80) >consensus GUUGGCACUGGGCACGCGACACCCGGCGACUUUGGGUGUGGGGGCGUGGCAUUGUGGCCGACAUGCCCACGCCCACACCCGCACC_____CACCCACUCCGUAUCCGUA____ ...((...((((....(.((((((((.....)))))))).)(((((((((......))...)))))))...))))...))................................. (-28.07 = -27.93 + -0.14)

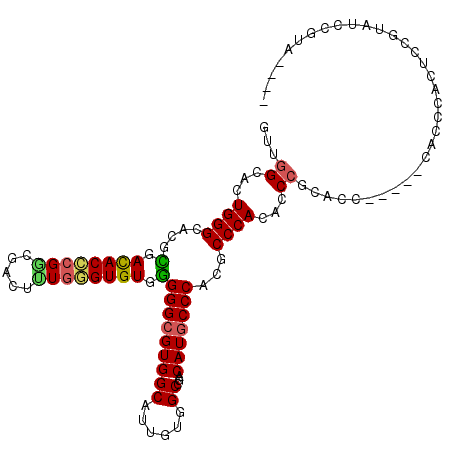
| Location | 7,741,826 – 7,741,935 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 74.64 |
| Mean single sequence MFE | -48.19 |
| Consensus MFE | -24.56 |
| Energy contribution | -25.37 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897156 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7741826 109 - 20766785 ----UACGGAUACGGAGUGGGUGCCGCCGGUGCGGGUAUGGGUGUGGGCAUGUCGGCCACAAUGCCACGCCCCCACACCCAAAGUCGCCGGGUGUCGCGUGCCCAGUGCCAAC ----...((.(((((.(((.(((.((((.(.((((.(.((((((((((..(((.(((......))))))..)))))))))).).))))).)))).))).))))).)))))... ( -55.70) >DroSec_CAF1 31825 106 - 1 ----UACGGAUACGGAGUGGGUGC---AUGUGCGGGUGUGGGUGUGGGCAUGUCGGCCACAAUGCCACGCCCCCACACCCAAAGUCGCCGGGUGUCGCGUGCCCAGUGCCAAC ----...((.(((((.(((.((((---((.(((((.(.((((((((((..(((.(((......))))))..)))))))))).).))).)).))).))).))))).)))))... ( -49.10) >DroEre_CAF1 25269 106 - 1 ----UACGGAUACGGUGUGGUUGG---GGGUGCGGGCGUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCCCACACCCAAAGUCGCCGGGUGUCGCGUGCCCAGUGCCAAC ----...((.(((((((...((((---(.(((.((((((((.(((.(((......)))...))))))))))).))).)))))...))))((((.......)))).)))))... ( -55.60) >DroYak_CAF1 25541 110 - 1 CGGAUACGGAUACGGUGGAGGUGU---GGGUGUGGGUGUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCCCACACCCAGAGUCGCCGGGUGUCGCGUGCCCAGUGCCAAC .((.((((((((((((((...(.(---(((((((((.((((((((.(((......)))...))))).))).)))))))))).).)))))..))))).)))).))......... ( -57.40) >DroAna_CAF1 23734 82 - 1 ----UAUG--------------------------GCUAUGGG-GCGGGUAUGUCGGCCACAAUGCCACGCCCACACACUGAAACGCGUCGCGUGUCCCGCGCCCAGUGCCAAU ----..((--------------------------((..((((-((((((((((.(((......)))((((..............)))).))))).))))).))))..)))).. ( -34.54) >DroPer_CAF1 23912 94 - 1 ----UACGGAAUGGGAAU---------------GGGAAUGGGCAUGGGCAUGGCUGCUACCAUGCCACGCCCCCACACACAGACGCGUCGCGUAUCACGCUCCCAAUGCCAGC ----.......(((.(.(---------------((((..((((...(((((((......)))))))..))))............((((........))))))))).).))).. ( -36.80) >consensus ____UACGGAUACGGAGUGGGUG_____GGUGCGGGUAUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCCCACACCCAAAGUCGCCGGGUGUCGCGUGCCCAGUGCCAAC .................................((((..((((((((.(((..........)))))))))))..((((((.........)))))).....))))......... (-24.56 = -25.37 + 0.81)
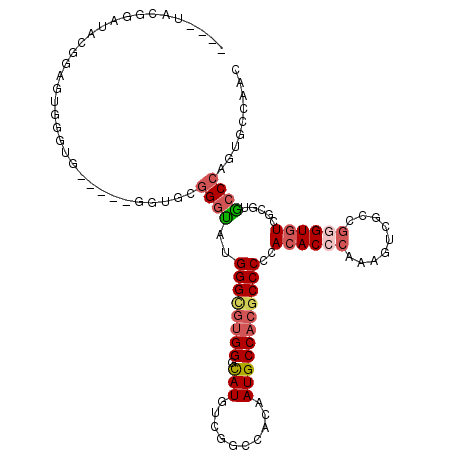
| Location | 7,741,866 – 7,741,958 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -39.00 |
| Consensus MFE | -25.74 |
| Energy contribution | -27.30 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.801985 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7741866 92 - 20766785 ACUCGCUGACGACCAGUCCCACA------UACGGAUACGGAGUGGGUGCCGCCGGUGCGGGUAUGGGUGUGGGCAUGUCGGCCACAAUGCCACGCCCC ((((((...((.(....(((((.------..((....))..)))))....).))..))))))..((((((((.(((((.....)).))))))))))). ( -37.30) >DroSec_CAF1 31865 89 - 1 ACUCGCUGUCUCCAAGUCCCACA------UACGGAUACGGAGUGGGUGC---AUGUGCGGGUGUGGGUGUGGGCAUGUCGGCCACAAUGCCACGCCCC ...((((..((((..((((....------...))))..))))..)))).---......((((((((.(((((.(.....).)))))...)))))))). ( -37.10) >DroSim_CAF1 43397 89 - 1 ACUCGCUGACUCCCAGUCCCACA------UACGGAUACGGAGUGGGUGC---CGGUGCUGGUGUGGGUGUGGGCAUGUCGGCCACAAUGCCACGUCCA ....((((((.((((..((((((------(.(((...(((........)---))...))))))))))..))))...))))))................ ( -35.10) >DroEre_CAF1 25309 89 - 1 ACUCACUGACGCCCAGUCCCACA------UACGGAUACGGUGUGGUUGG---GGGUGCGGGCGUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCC .......(.(((((....(((((------(.((....))))))))....---))))))((((((((.(((.(((......)))...))))))))))). ( -43.60) >DroYak_CAF1 25581 95 - 1 ACUCGCUGACGCCCAGUCCCACUUACGGAUACGGAUACGGUGGAGGUGU---GGGUGUGGGUGUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCC ...((((.(((((((((((((((..((....)).....))))).))).)---)))))).)))).((((((((.(((((.....)).))))))))))). ( -42.60) >DroPer_CAF1 23952 77 - 1 AUUCCCAGACCCCAAGUCCCAGC------UACGGAAUGGGAAU---------------GGGAAUGGGCAUGGGCAUGGCUGCUACCAUGCCACGCCCC (((((((...((((..(((....------...))).))))..)---------------))))))((((...(((((((......)))))))..)))). ( -38.30) >consensus ACUCGCUGACGCCCAGUCCCACA______UACGGAUACGGAGUGGGUGC___CGGUGCGGGUGUGGGCGUGGGCAUGUCGGCCACAAUGCCACGCCCC ...((((.(((((..((((.............))))..))))).))))..........((((((((.(((.(((......)))...))))))))))). (-25.74 = -27.30 + 1.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:20:18 2006