
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,683,855 – 7,684,099 |
| Length | 244 |
| Max. P | 0.885031 |

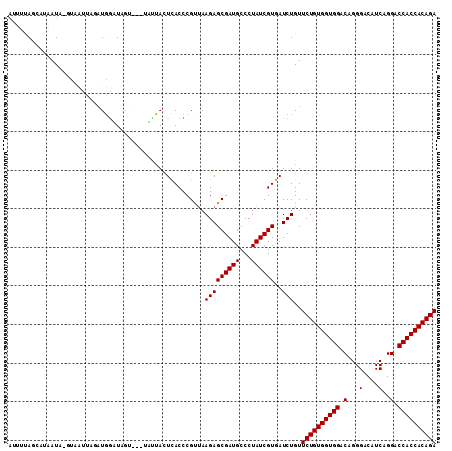
| Location | 7,683,855 – 7,683,966 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 81.80 |
| Mean single sequence MFE | -29.79 |
| Consensus MFE | -21.32 |
| Energy contribution | -21.32 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.72 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.520966 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7683855 111 + 20766785 AUUUUAGCAUAAUAAGUACUUAAAUGGUUAGUUAUUAUUACUCACCCGUCAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA ......((((.....((.(((....(((.(((.......))).)))....))).)).))))...............((((((((((.(...(....)..).)))))))))) ( -26.60) >DroGri_CAF1 5186 101 + 1 AAAGGAA----AUU-GCAAUUUGAUGGUUCG-----GUUAUUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA ..(((..----(((-((..((((((((....-----.........)))))))).)))))..)))............((((((((((.(...(....)..).)))))))))) ( -30.32) >DroSec_CAF1 2827 110 + 1 AUUUUAGCAUAAUA-GUACUUGGAUGAUUAGUUACUAUUACUCACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA .(((((((((....-))....((.(((.((((....)))).))).)))))))))((((((...)))))).......((((((((((.(...(....)..).)))))))))) ( -28.00) >DroEre_CAF1 5329 107 + 1 GUUUUAGGAUAAUA-GCCUUUACAUGGAUCAU---UUUUACUCACCCGCUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA ((...(((......-.)))..))(((((((((---.......((..(((.....))).)).......)))))))))((((((((((.(...(....)..).)))))))))) ( -29.94) >DroYak_CAF1 5305 107 + 1 GUCUUAGAAUGAUA-GUCUUUUCAUGGAUAGU---UCGUACUCACCCGCUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACCGGGACAUCAGGACCACCACAGA .((((((.((((..-......))))((..(((---....)))...)).))))))((((((...)))))).......((((((((((.((..(....).)).)))))))))) ( -32.90) >DroAna_CAF1 5650 106 + 1 AGUUAAUCAGAAUA-GAAACAAG-UUGAUAAA---AUCAACUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA ........((((((-((...(((-(((((...---))))))))...........((((((...))))))..))))))))(((((((.(...(....)..).)))))))... ( -31.00) >consensus AUUUUAGCAUAAUA_GUAAUUAGAUGGAUAGU___UAUUACUCACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGA ...................................................(((((((((...))))))..)))..((((((((((.(...(....)..).)))))))))) (-21.32 = -21.32 + -0.00)


| Location | 7,683,855 – 7,683,966 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 81.80 |
| Mean single sequence MFE | -31.81 |
| Consensus MFE | -24.22 |
| Energy contribution | -24.22 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.885031 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7683855 111 - 20766785 UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUGACGGGUGAGUAAUAAUAACUAACCAUUUAAGUACUUAUUAUGCUAAAAU ((((((((((.(..(......).).))))))))))..............(((.((((((.....))))))(((((((..(((.........)))..))))))))))..... ( -29.80) >DroGri_CAF1 5186 101 - 1 UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAACGGGUAAAUAAC-----CGAACCAUCAAAUUGC-AAU----UUCCUUU ....(((((((...((((.(((((((..................)))))))))))........(((........)-----)).)))))))......-...----....... ( -26.67) >DroSec_CAF1 2827 110 - 1 UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAACGGGUGAGUAAUAGUAACUAAUCAUCCAAGUAC-UAUUAUGCUAAAAU ((((((((((.(..(......).).))))))))))..............(((.((((((.....))))))(((((((((.((.........)))))-)))))))))..... ( -33.80) >DroEre_CAF1 5329 107 - 1 UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAGCGGGUGAGUAAAA---AUGAUCCAUGUAAAGGC-UAUUAUCCUAAAAC ((((((((((.(..(......).).))))))))))((((((((.......((.((((((.....))))))))....---.)))))..)))..(((.-......)))..... ( -30.10) >DroYak_CAF1 5305 107 - 1 UCUGUGGUGGUCCUGAUGUCCCGGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAGCGGGUGAGUACGA---ACUAUCCAUGAAAAGAC-UAUCAUUCUAAGAC ((((((((((.((.(.....).)).)))))))))).....(((.(((((..(.((((((.....))))))....).---.)))))..)))......-.............. ( -35.30) >DroAna_CAF1 5650 106 - 1 UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAACGGGUAAGUUGAU---UUUAUCAA-CUUGUUUC-UAUUCUGAUUAACU ((((((((((.(..(......).).))))))))))...(((((.((((((((...((((.....))))((((((((---...)))))-)))).)))-)))).))))).... ( -35.20) >consensus UCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGAUCACGAUAGGGCAUCGCUCUUAACGGGUGAGUAAAA___ACUAACCAUCUAAAUAC_UAUUAUGCUAAAAU ((((((((((.(...........).))))))))))....(((.((..(((((...)))))...)))))........................................... (-24.22 = -24.22 + -0.00)



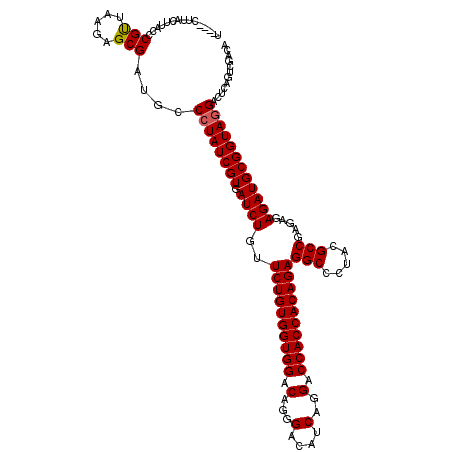
| Location | 7,683,886 – 7,684,006 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.24 |
| Mean single sequence MFE | -41.50 |
| Consensus MFE | -36.96 |
| Energy contribution | -36.98 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.777026 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7683886 120 + 20766785 UUAUUAUUACUCACCCGUCAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGACUCCGAGGACA .........(((...((((......)))).((((((((.((((..((((((((((.(...(....)..).))))))))))(((.....))).....)))))))))))).....))).... ( -42.20) >DroVir_CAF1 4532 115 + 1 -----UUUAUUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGACUCAGUCGACA -----..................(((((..((((((((.((((..((((((((((.(...(....)..).))))))))))(((.....))).....)))))))))))).....)))).). ( -39.50) >DroGri_CAF1 5212 115 + 1 -----GUUAUUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAAGAUUCAGUCGACA -----....((((((((((....((((((...))))))..(((..((((((((((.(...(....)..).))))))))))(((.....))).)))..)))).))))))............ ( -35.00) >DroWil_CAF1 56 120 + 1 UUAUACUAACUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGAUUCAUUCGACA ......((((......)))).(((.((...((((((((.((((..((((((((((.(...(....)..).))))))))))(((.....))).....))))))))))))..)).))).... ( -39.80) >DroYak_CAF1 5335 117 + 1 U---UCGUACUCACCCGCUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACCGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGACUCCGAGGACA .---..((.(((...(((.....)))....((((((((.((((..((((((((((.((..(....).)).))))))))))(((.....))).....)))))))))))).....))).)). ( -47.80) >DroAna_CAF1 5679 117 + 1 A---AUCAACUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGAGUCAGUUGACA .---.((((((.((.(((.....)))....((((((((.((((..((((((((((.(...(....)..).))))))))))(((.....))).....)))))))))))).)).)))))).. ( -44.70) >consensus U____CUUACUUACCCGUUAAGAGCGAUGCCCUAUCGUGAUCUGUUCUGUGGUGGACAGGGACAUCAGGACCACCACAGAGGCCCUACGCCGAGAGAGAUGCGGUAGGACUCAGUCGACA ...............(((.....)))....((((((((.((((..((((((((((.(...(....)..).))))))))))(((.....))).....))))))))))))............ (-36.96 = -36.98 + 0.03)

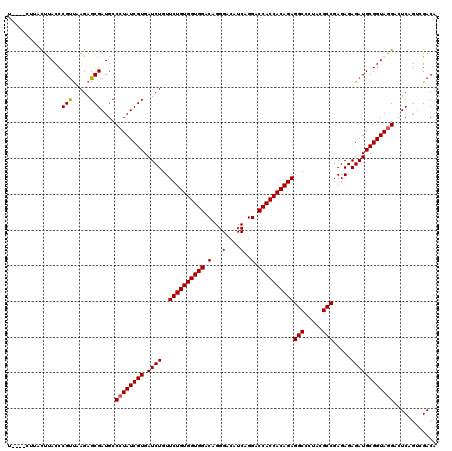
| Location | 7,683,926 – 7,684,046 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -36.74 |
| Consensus MFE | -32.86 |
| Energy contribution | -33.03 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.773034 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7683926 120 - 20766785 AAGCUCCGCACCAUCUCGACUUCGUCUUGCGCCACGACCAUGUCCUCGGAGUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA .......................((((((((((.(((........)))(((...............))))))))))))).((((((((((.(..(......).).))))))))))..... ( -37.46) >DroVir_CAF1 4567 120 - 1 AAGCUCCGUACCAUCUCGACUUCGUCUUGCGCCACGAACAUGUCGACUGAGUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA ...............((((((((((........)))))...)))))(((.(((((((.((..........))))))))).((((((((((.(..(......).).)))))))))).))). ( -39.50) >DroGri_CAF1 5247 120 - 1 AAGCUUCGCACCAUCUCGACUUCGUCUUGCGCCACGAACAUGUCGACUGAAUCUUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA .......................((((((((((..((..(((.((............)))))...))..)))))))))).((((((((((.(..(......).).))))))))))..... ( -35.10) >DroWil_CAF1 96 120 - 1 AAGUUCCGCACCAUCUCGACUUCGUCUUGCGCCACGAACAUGUCGAAUGAAUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA .......((..(((.((((((((((........)))))...))))))))...(((((.((..........))))))))).((((((((((.(..(......).).))))))))))..... ( -36.30) >DroYak_CAF1 5372 120 - 1 AAACUCCGCACCAUCUCGACUUCGUCUUGCGCCACGACCAUGUCCUCGGAGUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCGGUCCACCACAGAACAGA .......................((((((((((.(((........)))(((...............))))))))))))).((((((((((.((.(.....).)).))))))))))..... ( -41.46) >DroAna_CAF1 5716 120 - 1 AAACUCCGCACCAUCUCGACUUCUUCUUGCGCCACGACCAUGUCAACUGACUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA ........................(((((((((..(((...)))....(........)...........)))))))))..((((((((((.(..(......).).))))))))))..... ( -30.60) >consensus AAGCUCCGCACCAUCUCGACUUCGUCUUGCGCCACGAACAUGUCGACUGAGUCCUACCGCAUCUCUCUCGGCGUAGGGCCUCUGUGGUGGUCCUGAUGUCCCUGUCCACCACAGAACAGA .......................((((((((((..((..((((...............))))...))..)))))))))).((((((((((.(...........).))))))))))..... (-32.86 = -33.03 + 0.17)

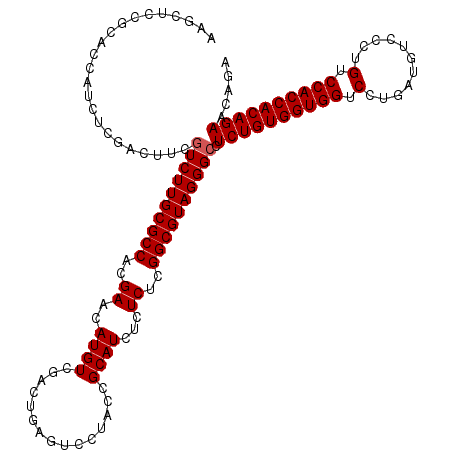
| Location | 7,684,006 – 7,684,099 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.85 |
| Mean single sequence MFE | -29.62 |
| Consensus MFE | -19.93 |
| Energy contribution | -20.27 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.67 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.521597 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7684006 93 + 20766785 UGGUCGUGGCGCAAGACGAAGUCGAGAUGGUGCGGAGCUUGCCGUC---------------------------GGCAAUUCCGCUGUCAUUAAAUUCUCUUCUAACGAAGCCACUACUAA ((((.(((((....((.(((.....((((..((((((.(((((...---------------------------)))))))))))...))))...)))))(((....)))))))).)))). ( -32.70) >DroVir_CAF1 4647 102 + 1 UGUUCGUGGCGCAAGACGAAGUCGAGAUGGUACGGAGCUUGCCAUCGG------------------CACAUCCUGCAACUCCGCUAUCAUUAAAAUCUCUUCUAACGAAGUUACUACUAA ..(((((..(....)))))).....((((((((((((.((((....((------------------.....)).))))))))).))))))).......((((....)))).......... ( -26.10) >DroGri_CAF1 5327 102 + 1 UGUUCGUGGCGCAAGACGAAGUCGAGAUGGUGCGAAGCUUGCUAUCGG------------------AACAUCCAGCUACUCCGCUAUCAUUAUAGUCUCUUCUAACGAAGGUACUACUAA ..(((((..(....)))))).....(((((((((.((...(((...((------------------.....)))))..)).))).)))))).((((..((((....))))..)))).... ( -26.70) >DroSec_CAF1 2977 93 + 1 UGGUCGUGGCGCAAGACGAAGUCGAGAUGGUGCGGAGCUUACCGUC---------------------------GGCAAUUCCGCUGUCAUUAAAUUCUCUUCUAACGAAGCCACUACUAA ((((.(((((....(((........(((((((.......)))))))---------------------------(((......))))))...........(((....)))))))).)))). ( -27.90) >DroWil_CAF1 176 120 + 1 UGUUCGUGGCGCAAGACGAAGUCGAGAUGGUGCGGAACUUGCCAUCGGAGGCCUUAGCUGCAGCAGCACAGGCGGCAAUUCCGCUAUCAUUAAAAUCUCUUCUAACGAUACUACUACUUA .((((((..(....)..((((..((((((((((((((.(((((.......((((..((((...))))..))))))))))))))).))))))....)).))))..)))).))......... ( -35.91) >DroAna_CAF1 5796 93 + 1 UGGUCGUGGCGCAAGAAGAAGUCGAGAUGGUGCGGAGUUUGCCAUC---------------------------GGCAAUACCGCUGUCAUUAAACUCUCUUCUAACGAUACUACUACUAA ((((.((((((..(((((((((...((((..((((...(((((...---------------------------)))))..))))...))))..))).))))))..))...)))).)))). ( -28.40) >consensus UGGUCGUGGCGCAAGACGAAGUCGAGAUGGUGCGGAGCUUGCCAUC___________________________GGCAAUUCCGCUAUCAUUAAAAUCUCUUCUAACGAAGCUACUACUAA .....(((((.......((((..((((((((((((((.(((((..............................))))))))))).))))))....)).)))).......)))))...... (-19.93 = -20.27 + 0.34)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:19:35 2006