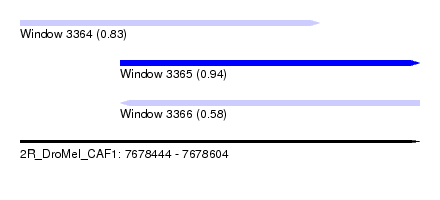
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,678,444 – 7,678,604 |
| Length | 160 |
| Max. P | 0.940731 |

| Location | 7,678,444 – 7,678,564 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.50 |
| Mean single sequence MFE | -58.78 |
| Consensus MFE | -41.66 |
| Energy contribution | -40.26 |
| Covariance contribution | -1.40 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.828627 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7678444 120 + 20766785 CGGAGCCCCCGGUGGCGGAAUCUGCUGAAUUUGGUUGGCCAUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGGGCUGCCCA ....((((((((..(((((....((..(......)..))..)))))..))))).).)).(((.((....)).))).(((((((((((...((((.....))))....))..))))))))) ( -58.50) >DroPse_CAF1 7056 120 + 1 GGGUGCUCCUGGUGGUGGUAUUUGUUGUAUCUGGUUGGCCAUCUGCUGCUGCGCAUGCAAUUGUGCCUGCUGUGCGUGUGCAGCCGCGUAUGCCUGCUGGGCCAUCAAUUGAGCGGCCCA ((((((((.(((((((.((((.....))))(..((.(((.((.(((.((((((((((((...((....))..)))))))))))).))).))))).))..))))))))...)))).)))). ( -58.60) >DroSec_CAF1 8160 120 + 1 CGGAGCACCCGGUGGCGGAAUCUGCUGGAUCUGGUUGGCCAUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGGGCUGCCCA ....((((((((..(((((....((..(......)..))..)))))..)))))..))).(((.((....)).))).(((((((((((...((((.....))))....))..))))))))) ( -58.30) >DroSim_CAF1 7846 120 + 1 CGGAGCACCCGGUGGCGGAAUCUGCUGGAUCUGGUUGGCCAUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGGGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGUGCUGCCCA .((.((((((((..(((((....((..(......)..))..)))))..))))).(..(((((((((((((.((((.((.((....)).)).)))))).))))..)))))))..)))))). ( -59.90) >DroPer_CAF1 7057 120 + 1 GGGUGCUCCUGGUGGUGGUAUUUGUUGUAUCUGGUUGGCCAUCUGCUGCUGCGCAUGCAAUUGUGCCUGCUGUGCGUGUGCAGCCGCGUAUGCCUGCUGGGCCAUCAAUUGAGCGGCCCA ((((((((.(((((((.((((.....))))(..((.(((.((.(((.((((((((((((...((....))..)))))))))))).))).))))).))..))))))))...)))).)))). ( -58.60) >consensus CGGAGCACCCGGUGGCGGAAUCUGCUGGAUCUGGUUGGCCAUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGAGCUGCCCA .(((((((..((((((((......))....(..((.((((...(((.((((..(((((...............)))))..)))).)))..)))).))..)))))))....))))).)).. (-41.66 = -40.26 + -1.40)

| Location | 7,678,484 – 7,678,604 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.67 |
| Mean single sequence MFE | -54.52 |
| Consensus MFE | -40.88 |
| Energy contribution | -41.00 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940731 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7678484 120 + 20766785 AUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGGGCUGCCCACGCAUUUUGUGGUAUCAUGUACGGCGGAAUGGACAUCGUU ......((((.((((.((......)).)))).))))(((((((((((...((((.....))))....))..)))))))))..((((((((.((((...)))).))))))))......... ( -53.00) >DroPse_CAF1 7096 120 + 1 AUCUGCUGCUGCGCAUGCAAUUGUGCCUGCUGUGCGUGUGCAGCCGCGUAUGCCUGCUGGGCCAUCAAUUGAGCGGCCCAUGCAUUUUGUGGUAUCAUGUACGGCGGAAUGGACAUCUUU .((((((((((((((((((...((....))..)))))))))))).((((((((((((((((((.((....))..)))))).)))......))))).))))..))))))............ ( -53.10) >DroSec_CAF1 8200 120 + 1 AUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGGGCUGCCCACGCAUUCUGCGGUAUCAUGUACGGCGGAAUGGACAUCGUU ......((((.((((.((......)).)))).))))(((((((((((...((((.....))))....))..)))))))))..((((((((.((((...)))).))))))))......... ( -57.00) >DroSim_CAF1 7886 120 + 1 AUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGGGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGUGCUGCCCACGCAUUCUGCGGUAUCAUGUACGGCGGAAUGGACAUCGUU .(((...((((((((..(((((((((((((.((((.((.((....)).)).)))))).))))..)))))))..).))))).))(((((((.((((...)))).))))))))))....... ( -56.40) >DroPer_CAF1 7097 120 + 1 AUCUGCUGCUGCGCAUGCAAUUGUGCCUGCUGUGCGUGUGCAGCCGCGUAUGCCUGCUGGGCCAUCAAUUGAGCGGCCCAUGCAUUUUGUGGUAUCAUGUACGGCGGAAUGGACAUCUUU .((((((((((((((((((...((....))..)))))))))))).((((((((((((((((((.((....))..)))))).)))......))))).))))..))))))............ ( -53.10) >consensus AUCUGCUGCUGGGCGUGCAGUUGAGCCUGCUGAGCAUGGGCAGCCGCGUAGGCCUGCUGGGCCAUCAGCUGAGCUGCCCACGCAUUUUGUGGUAUCAUGUACGGCGGAAUGGACAUCGUU ..((((((((.((((.((......)).)))).))))...))))((((((.(((..(((.(((.....))).))).))).))))(((((((.((((...)))).)))))))))........ (-40.88 = -41.00 + 0.12)

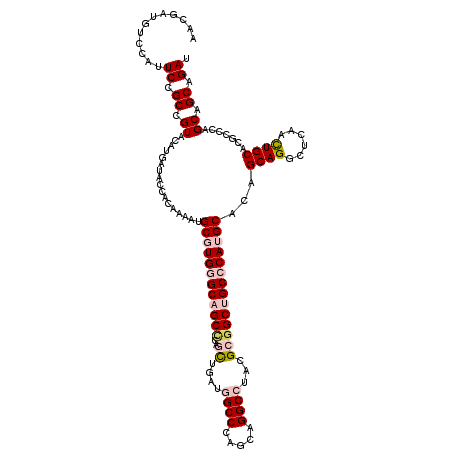
| Location | 7,678,484 – 7,678,604 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.67 |
| Mean single sequence MFE | -43.52 |
| Consensus MFE | -31.56 |
| Energy contribution | -33.12 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581261 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7678484 120 - 20766785 AACGAUGUCCAUUCCGCCGUACAUGAUACCACAAAAUGCGUGGGCAGCCCAGCUGAUGGCCCAGCAGGCCUACGCGGCUGCCCAUGCUCAGCAGGCUCAACUGCACGCCCAGCAGCAGAU ............((.((.((.................((((((((((((..((....((((.....))))...))))))))))))))...((((......)))).......)).)).)). ( -44.60) >DroPse_CAF1 7096 120 - 1 AAAGAUGUCCAUUCCGCCGUACAUGAUACCACAAAAUGCAUGGGCCGCUCAAUUGAUGGCCCAGCAGGCAUACGCGGCUGCACACGCACAGCAGGCACAAUUGCAUGCGCAGCAGCAGAU ...............((((((.....))).......(((.(((((((.((....))))))))))))))).......(((((...((((..((((......)))).))))..))))).... ( -39.50) >DroSec_CAF1 8200 120 - 1 AACGAUGUCCAUUCCGCCGUACAUGAUACCGCAGAAUGCGUGGGCAGCCCAGCUGAUGGCCCAGCAGGCCUACGCGGCUGCCCAUGCUCAGCAGGCUCAACUGCACGCCCAGCAGCAGAU ...((.(((.((((.((.(((.....))).)).))))((((((((((((..((....((((.....))))...))))))))))))))......)))))..((((..((...)).)))).. ( -47.70) >DroSim_CAF1 7886 120 - 1 AACGAUGUCCAUUCCGCCGUACAUGAUACCGCAGAAUGCGUGGGCAGCACAGCUGAUGGCCCAGCAGGCCUACGCGGCUGCCCAUGCCCAGCAGGCUCAACUGCACGCCCAGCAGCAGAU ...((.(((.((((.((.(((.....))).)).))))(((((((((((...((....((((.....))))...)).)))))))))))......)))))..((((..((...)).)))).. ( -46.30) >DroPer_CAF1 7097 120 - 1 AAAGAUGUCCAUUCCGCCGUACAUGAUACCACAAAAUGCAUGGGCCGCUCAAUUGAUGGCCCAGCAGGCAUACGCGGCUGCACACGCACAGCAGGCACAAUUGCAUGCGCAGCAGCAGAU ...............((((((.....))).......(((.(((((((.((....))))))))))))))).......(((((...((((..((((......)))).))))..))))).... ( -39.50) >consensus AACGAUGUCCAUUCCGCCGUACAUGAUACCACAAAAUGCGUGGGCAGCCCAGCUGAUGGCCCAGCAGGCCUACGCGGCUGCCCAUGCACAGCAGGCUCAACUGCACGCCCAGCAGCAGAU ............((.((.((.................((((((((((((..((....((((.....))))...))))))))))))))...((((......)))).......)).)).)). (-31.56 = -33.12 + 1.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:19:26 2006