
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
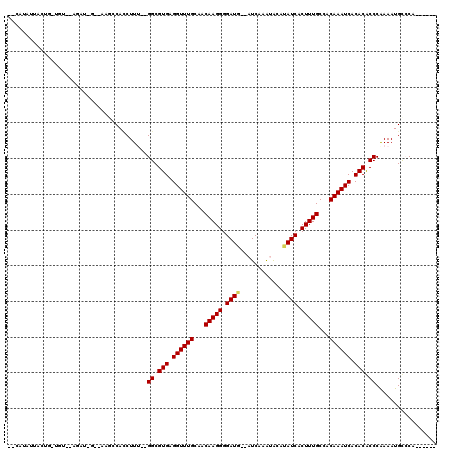
| Location | 7,674,762 – 7,674,866 |
| Length | 104 |
| Max. P | 0.993842 |

| Location | 7,674,762 – 7,674,866 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.79 |
| Mean single sequence MFE | -28.19 |
| Consensus MFE | -16.55 |
| Energy contribution | -16.55 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.870283 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
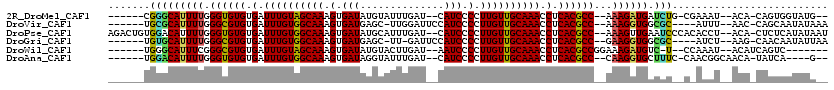
>2R_DroMel_CAF1 7674762 104 + 20766785 --CAUACCACUG-UGU--AUUUCG-CAGAUCAUCUUU--GGCGUGAGGUUUGCAACAAGGGGAUG--AUCAAAUACAUAUCACUUUGCUACAAAUCACACACCCAAAAUGCCCG------ --.......(((-((.--....))-)))..(((.(((--((.(((.((((((...(((((.((((--..........)))).)))))...)))))).)))..))))))))....------ ( -23.20) >DroVir_CAF1 4465 104 + 1 UUUAUAUUGCUG-GUU--AAAU----GCGCCACCUUU--GGCGUGAGGUUUGCAACAAGGGGAUGGAAUCCAA-GCUCAUCACUUUGCCACAAAUCACACGCCCAAAAUGCGCA------ ............-...--...(----((((.......--((((((.((((((...(((((.(((((.......-..))))).)))))...)))))).))))))......)))))------ ( -32.12) >DroPse_CAF1 3154 113 + 1 AUUAUAUGAGAG-UGU--AGGUGUGGGAUUCAACUUU--GGCGUGAGGUUUGCAACAAGGGGAUG--AUCAAAUGCAUAUCACUUUGCCACAAAUCACACACCCAAAAUGUCCACAGUCU .((((((....)-)))--)).((((((.......(((--((.(((.((((((...(((((.((((--..(....)..)))).)))))...)))))).)))..)))))...)))))).... ( -28.10) >DroGri_CAF1 5406 103 + 1 UUAAUAUUGUUG-CUU--AGAU----GCGCCACCUUC--GGCGUGAGGUUUGCAACAAGGGGAUGGAAUC-AA-GCUCAUCACUUUGCCACAAAUCACACGCCCAAAAUGCACA------ ...........(-(..--....----))((.......--((((((.((((((...(((((.(((((....-..-..))))).)))))...)))))).))))))......))...------ ( -28.92) >DroWil_CAF1 4409 100 + 1 -------GACUGAUGU--AUUUGG--A-GACAUCUUUCCGGCGUGAGGUUUGCAACAAGGGGAUU--AUCAAGUACAUAUCACUUUGCUACAAAUCACACGCCCGAAAUGCCCA------ -------.......((--((((((--(-((....)))))((((((.((((((...(((((.(((.--...........))).)))))...)))))).))))))..))))))...------ ( -28.90) >DroAna_CAF1 4457 102 + 1 --C----UGAUA-UGUUGCCGUUG-GAAAGCACCUUG--GGCGUGAGGUUUGCAACAAGGGGAUG--AUCAAAUACCUAUCACUUUGCCACAAAUCACACACCCAAAAUGUCCA------ --.----.((((-(......(((.-...)))...(((--((.(((.((((((...(((((.((((--..........)))).)))))...)))))).))).))))).)))))..------ ( -27.90) >consensus __CAUAUUACUG_UGU__AGAU_G__AAGCCACCUUU__GGCGUGAGGUUUGCAACAAGGGGAUG__AUCAAAUACAUAUCACUUUGCCACAAAUCACACACCCAAAAUGCCCA______ .......................................((.(((.((((((...(((((.((((............)))).)))))...)))))).))).))................. (-16.55 = -16.55 + 0.00)


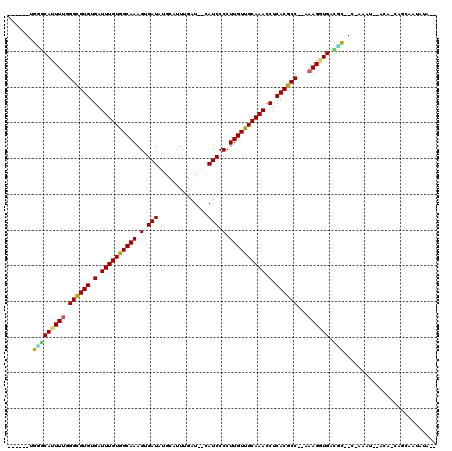
| Location | 7,674,762 – 7,674,866 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.79 |
| Mean single sequence MFE | -34.26 |
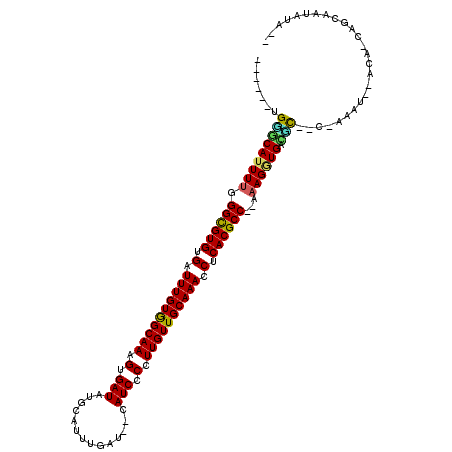
| Consensus MFE | -24.89 |
| Energy contribution | -23.97 |
| Covariance contribution | -0.92 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.73 |
| SVM decision value | 2.43 |
| SVM RNA-class probability | 0.993842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7674762 104 - 20766785 ------CGGGCAUUUUGGGUGUGUGAUUUGUAGCAAAGUGAUAUGUAUUUGAU--CAUCCCCUUGUUGCAAACCUCACGCC--AAAGAUGAUCUG-CGAAAU--ACA-CAGUGGUAUG-- ------((((((((((.((((((.(.((((((((((.(((((.........))--)))....)))))))))).).))))))--.)))))).))))-....((--((.-.....)))).-- ( -30.80) >DroVir_CAF1 4465 104 - 1 ------UGCGCAUUUUGGGCGUGUGAUUUGUGGCAAAGUGAUGAGC-UUGGAUUCCAUCCCCUUGUUGCAAACCUCACGCC--AAAGGUGGCGC----AUUU--AAC-CAGCAAUAUAAA ------((((((((((.((((((.(.((((..((((.(.((((...-........)))).).))))..)))).).))))))--.))))).))))----)...--...-............ ( -36.80) >DroPse_CAF1 3154 113 - 1 AGACUGUGGACAUUUUGGGUGUGUGAUUUGUGGCAAAGUGAUAUGCAUUUGAU--CAUCCCCUUGUUGCAAACCUCACGCC--AAAGUUGAAUCCCACACCU--ACA-CUCUCAUAUAAU .((..(((.......((((((((.(((((.((((...((((..((((......--...........))))....)))))))--).....))))).)))))))--)))-)..))....... ( -28.04) >DroGri_CAF1 5406 103 - 1 ------UGUGCAUUUUGGGCGUGUGAUUUGUGGCAAAGUGAUGAGC-UU-GAUUCCAUCCCCUUGUUGCAAACCUCACGCC--GAAGGUGGCGC----AUCU--AAG-CAACAAUAUUAA ------((((((((((.((((((.(.((((..((((.(.((((...-..-.....)))).).))))..)))).).))))))--.))))).))))----)...--...-............ ( -37.10) >DroWil_CAF1 4409 100 - 1 ------UGGGCAUUUCGGGCGUGUGAUUUGUAGCAAAGUGAUAUGUACUUGAU--AAUCCCCUUGUUGCAAACCUCACGCCGGAAAGAUGUC-U--CCAAAU--ACAUCAGUC------- ------..(((.((((.((((((.(.((((((((((.(.(((.(((.....))--)))).).)))))))))).).)))))).))))(((((.-.--......--))))).)))------- ( -34.10) >DroAna_CAF1 4457 102 - 1 ------UGGACAUUUUGGGUGUGUGAUUUGUGGCAAAGUGAUAGGUAUUUGAU--CAUCCCCUUGUUGCAAACCUCACGCC--CAAGGUGCUUUC-CAACGGCAACA-UAUCA----G-- ------(((((((((((((((((.(.((((..((((.(.(((.(((.....))--)))).).))))..)))).).))))))--)))))))...))-))..(....).-.....----.-- ( -38.70) >consensus ______UGGGCAUUUUGGGCGUGUGAUUUGUGGCAAAGUGAUAUGCAUUUGAU__CAUCCCCUUGUUGCAAACCUCACGCC__AAAGGUGACGC__C_AAAU__ACA_CAGCAAUAUA__ .......(((((((((.((((((.(.((((((((((.(.(((..............))).).)))))))))).).))))))...)))))).))).......................... (-24.89 = -23.97 + -0.92)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:19:20 2006