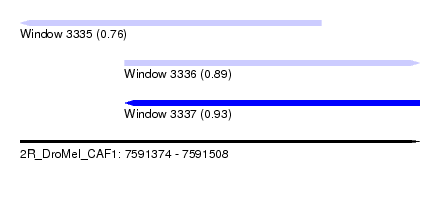
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,591,374 – 7,591,508 |
| Length | 134 |
| Max. P | 0.926203 |

| Location | 7,591,374 – 7,591,475 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 83.08 |
| Mean single sequence MFE | -46.55 |
| Consensus MFE | -27.36 |
| Energy contribution | -30.62 |
| Covariance contribution | 3.26 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.755292 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7591374 101 - 20766785 GCGUAUCCGUA--UCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUUGUGCUGCACUCAACGCGAUGCGCA (((((((((((--(((......)))).....(((((((((.(((((((((......))))))))).))))))((.((....)).)).))).))).))))))). ( -49.00) >DroVir_CAF1 51236 89 - 1 ----AUUGAUAAGACAGCCGGAC-GUGUACUGG----CAUGUG-----CUUGCAUUGUCCUGGCAAUUGCCGGCUGCUUUUCUACUAGCAUGCGCGAUAUGCA ----...((.(((.(((((((((-(((((..((----(....)-----)))))).))))).(((....))))))))))).)).....(((((.....))))). ( -33.50) >DroSec_CAF1 37087 101 - 1 GCGUAUCCGUA--UCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUUGCGCCGCACUCAACGCGAUGCGCA ((((((((((.--...((.(..(((((.(..((.((((((.(((((((((......))))))))).)))))).))...).)))))..))).))).))))))). ( -53.40) >DroSim_CAF1 37876 101 - 1 GCGUAUCCGUA--UCGGACGGACGGUGGACAGGACUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUUGUGCCGCACUCAACGCGAUGCGCA ((((((((((.--.((......))(((((((((.((((((.(((((((((......))))))))).))))))....))))).)))).....))).))))))). ( -52.70) >DroEre_CAF1 36441 101 - 1 CCGUAUCCGUA--UCGGACGGACGGCGGACAGGGAUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUGGUGCCGCACUCAACGCGAUGCGCA .(((((((((.--..(..(((.((.(((.(((...(((((.(((((((((......))))))))).)))))..)))))).)))))..)...))).)))))).. ( -47.30) >DroYak_CAF1 37716 101 - 1 CCGUAUCCGUA--UCGGACGGUCAGUGGACAGGGAUGCAGGUGCCGGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUUGUGCCGCACUCAACGCGAUGCGAA .(((((((((.--..(..(((.((..((.(((...(((((.(((((((((......))))))))).)))))..)))))..)))))..)...))).)))))).. ( -43.40) >consensus GCGUAUCCGUA__UCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUUGCAGGCUGCUUGUGCCGCACUCAACGCGAUGCGCA .(((((((((....((......))(((((((((..(((((.(((((((((......))))))))).))))).....))))).)))).....))).)))))).. (-27.36 = -30.62 + 3.26)

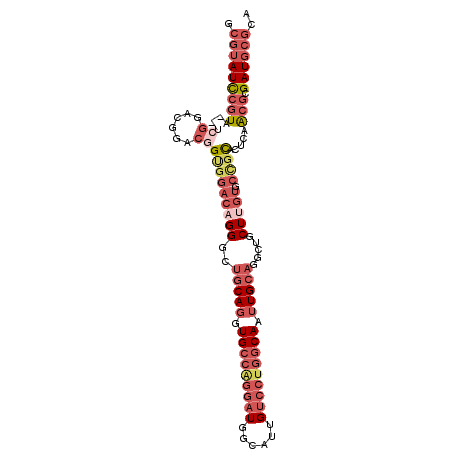
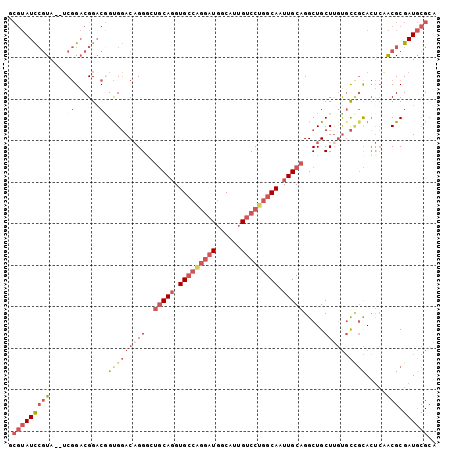
| Location | 7,591,409 – 7,591,508 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 86.06 |
| Mean single sequence MFE | -38.60 |
| Consensus MFE | -27.37 |
| Energy contribution | -29.30 |
| Covariance contribution | 1.93 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.887754 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7591409 99 + 20766785 AAUUGCCAGGACAAUGCCAUCCUGGCACCUGCAGCCCUGUCCACCGUCCGUCCGAUACGGAUACGCA------GUGCCCAGGCACAGGACGAUGGUUACGUGCCA ...((((((((........))))))))......(((.((.(((.((((((((((...))))).....------((((....)))).))))).))).)).).)).. ( -39.20) >DroSec_CAF1 37122 99 + 1 AAUUGCCAGGACAAUGCCAUCCUGGCACCUGCAGCCCUGUCCACCGUCCGUCCGAUACGGAUACGCA------GUGCCCAGGCACAGGACGCCGGUUACGUGCCA ...((((((((........))))))))((((..(((.((..(((.((..(((((...)))))..)).------)))..))))).))))..((((....)).)).. ( -38.10) >DroSim_CAF1 37911 99 + 1 AAUUGCCAGGACAAUGCCAUCCUGGCACCUGCAGUCCUGUCCACCGUCCGUCCGAUACGGAUACGCA------GUGCCCAGGCACAGGACGAUGGUUACGUGCCA ...((((((((........))))))))...(((((.....(((.((((((((((...))))).....------((((....)))).))))).)))..)).))).. ( -39.60) >DroEre_CAF1 36476 94 + 1 AAUUGCCAGGACAAUGCCAUCCUGGCACCUGCAUCCCUGUCCGCCGUCCGUCCGAUACGGAUACGGAUAAG-----------GAUACGCCGACGGUUACGUGCCA ...((((((((........)))))))).(.((((((.((((((..((((((.....)))))).)))))).)-----------)))..)).)..(((.....))). ( -37.30) >DroYak_CAF1 37751 105 + 1 AAUUGCCAGGACAAUGCCAUCCCGGCACCUGCAUCCCUGUCCACUGACCGUCCGAUACGGAUACGGAUACGAGGCGGACGGGCACAGGACGACGGCUACGUGCCA ...((((.(((........))).))))((((....(((((((.((....(((((.........)))))....)).)))))))..)))).....(((.....))). ( -38.80) >consensus AAUUGCCAGGACAAUGCCAUCCUGGCACCUGCAGCCCUGUCCACCGUCCGUCCGAUACGGAUACGCA______GUGCCCAGGCACAGGACGACGGUUACGUGCCA ...((((((((........))))))))...(((.......(((.((((((((((...)))))...........((((....)))).))))).))).....))).. (-27.37 = -29.30 + 1.93)

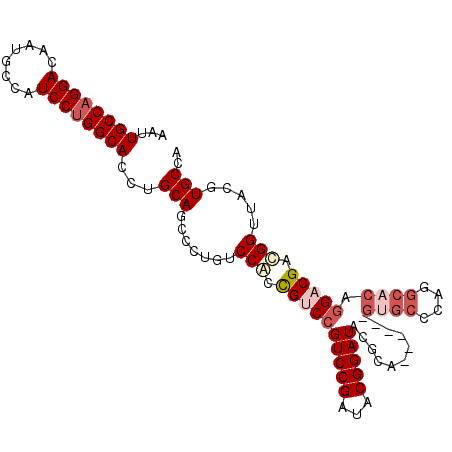
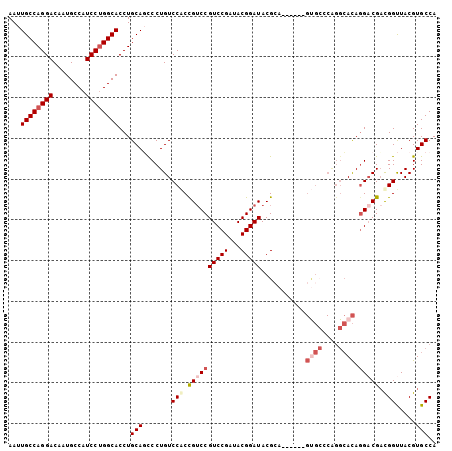
| Location | 7,591,409 – 7,591,508 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 86.06 |
| Mean single sequence MFE | -44.94 |
| Consensus MFE | -32.67 |
| Energy contribution | -33.84 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.926203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
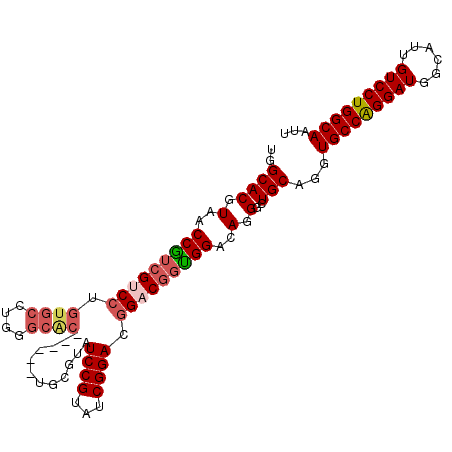
>2R_DroMel_CAF1 7591409 99 - 20766785 UGGCACGUAACCAUCGUCCUGUGCCUGGGCAC------UGCGUAUCCGUAUCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUU ..(((.((..(((((((((.((((....))))------......((((...)))).))))))))).....)))))...(((((((((......)))))))))... ( -45.00) >DroSec_CAF1 37122 99 - 1 UGGCACGUAACCGGCGUCCUGUGCCUGGGCAC------UGCGUAUCCGUAUCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUU ..((.((....))))..((((((((((..(((------((....(((((.....))))))))))..)).))).)))))(((((((((......)))))))))... ( -43.60) >DroSim_CAF1 37911 99 - 1 UGGCACGUAACCAUCGUCCUGUGCCUGGGCAC------UGCGUAUCCGUAUCGGACGGACGGUGGACAGGACUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUU ..((......(((((((((.((((....))))------......((((...)))).))))))))).((((((....((((((.....)))))))))))))).... ( -44.90) >DroEre_CAF1 36476 94 - 1 UGGCACGUAACCGUCGGCGUAUC-----------CUUAUCCGUAUCCGUAUCGGACGGACGGCGGACAGGGAUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUU ..((.((.......))))(((((-----------(((.(((((.(((((.....)))))..))))).))))))))...(((((((((......)))))))))... ( -44.60) >DroYak_CAF1 37751 105 - 1 UGGCACGUAGCCGUCGUCCUGUGCCCGUCCGCCUCGUAUCCGUAUCCGUAUCGGACGGUCAGUGGACAGGGAUGCAGGUGCCGGGAUGGCAUUGUCCUGGCAAUU .(((.....))).....(((((((((((((((.......(((..((((...)))))))...)))))).))).))))))(((((((((......)))))))))... ( -46.60) >consensus UGGCACGUAACCGUCGUCCUGUGCCUGGGCAC______UGCGUAUCCGUAUCGGACGGACGGUGGACAGGGCUGCAGGUGCCAGGAUGGCAUUGUCCUGGCAAUU ..((((.(..(((((((((.((((....))))............((((...)))).)))))))))..).)..)))...(((((((((......)))))))))... (-32.67 = -33.84 + 1.17)

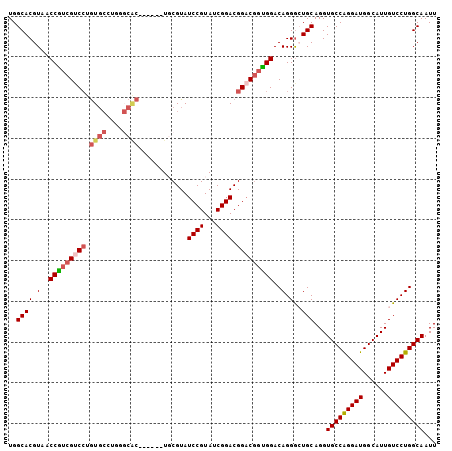
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:18:57 2006