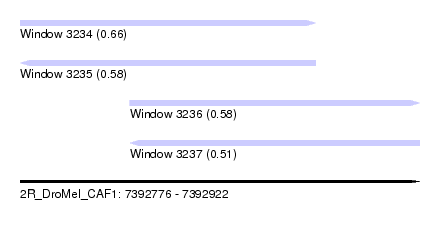
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,392,776 – 7,392,922 |
| Length | 146 |
| Max. P | 0.659046 |

| Location | 7,392,776 – 7,392,884 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 83.33 |
| Mean single sequence MFE | -35.38 |
| Consensus MFE | -24.26 |
| Energy contribution | -24.43 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.659046 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
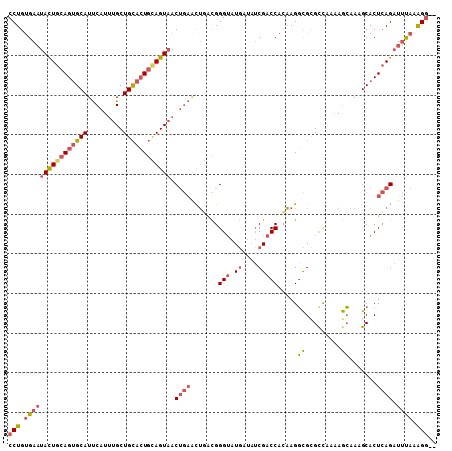
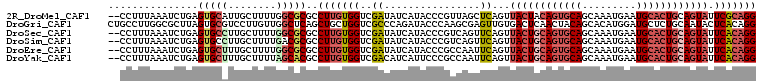
>2R_DroMel_CAF1 7392776 108 + 20766785 UGAUUGGCGUAUUUUUCAGUCACUUGUUUCUUACAGAGGGCCUGCGAAUACUGCAGUGCAUUCAUUUGCUGCACUGUAGUAACUGAGCUAACGGGUAUGAUAUCGACC .((((((........))))))(((((((.(((((((.....)))....((((((((((((.........))))))))))))..))))..)))))))............ ( -30.50) >DroVir_CAF1 8584 108 + 1 UCAUUGCUAUACUUGUCCGUUAUCUUCUGCUCCAGCAGGGCCUGUGAGUAUUGCAGCGCAUCCAGGUGCUUCAGUUGGGUAACAGCUCGCUUGGGUAUCUUGACCAGC ....(((.(((((..(..((.....(((((....)))))))..)..))))).)))((...((.(((((((..(((.((((....)))))))..))))))).))...)) ( -31.00) >DroSec_CAF1 8977 108 + 1 UGAUUGGCGUAUUUUUCAGUCACUUGUUGCUUACAGAGGGCCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACC .(((...((((((..(((((((........((((((.....)))))).((((((((((((.........))))))))))))..)).)))))..))))))..))).... ( -38.90) >DroSim_CAF1 407 108 + 1 UGAUUGGCGUAUUUUUCAGUCACUUGUUGCUUACAGAGGGCCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACC .(((...((((((..(((((((........((((((.....)))))).((((((((((((.........))))))))))))..)).)))))..))))))..))).... ( -38.90) >DroEre_CAF1 8759 108 + 1 UCAUUGGCGUAUUUUUCAGUCACUUGCUGCUUACUGAGGGCCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAAUUGGCGGGUAUGAUAUCGACC ((((...(((....(((((((((..(((.(((...))))))..)))).((((((((((((.........)))))))))))).)))))...)))...))))........ ( -37.00) >DroYak_CAF1 7339 108 + 1 UCAUUGGCGUGUUUUUCAGUUACUUGUUGCUUACUGAGGGCCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAAUUGGCGGGAAUGAUGUCGACC ...(((((((..((((((((............))))))))(((((.((((((((((((((.........))))))))))))......)).)))))....))))))).. ( -36.00) >consensus UCAUUGGCGUAUUUUUCAGUCACUUGUUGCUUACAGAGGGCCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACC .......((((((..((((((((.....((((.....))))..)))).((((((((((((.........))))))))))))......))))..))))))......... (-24.26 = -24.43 + 0.17)

| Location | 7,392,776 – 7,392,884 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 83.33 |
| Mean single sequence MFE | -29.30 |
| Consensus MFE | -19.58 |
| Energy contribution | -20.08 |
| Covariance contribution | 0.51 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.575057 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7392776 108 - 20766785 GGUCGAUAUCAUACCCGUUAGCUCAGUUACUACAGUGCAGCAAAUGAAUGCACUGCAGUAUUCGCAGGCCCUCUGUAAGAAACAAGUGACUGAAAAAUACGCCAAUCA ((.((...........(....)(((((((((.(((((((.........)))))))..((.(((((((.....))))..))))).)))))))))......))))..... ( -26.90) >DroVir_CAF1 8584 108 - 1 GCUGGUCAAGAUACCCAAGCGAGCUGUUACCCAACUGAAGCACCUGGAUGCGCUGCAAUACUCACAGGCCCUGCUGGAGCAGAAGAUAACGGACAAGUAUAGCAAUGA ((((((......)))..)))..(((((.((....(((.....(((((((((...)))....)).))))..((((....)))).......)))....)))))))..... ( -22.30) >DroSec_CAF1 8977 108 - 1 GGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGGCCCUCUGUAAGCAACAAGUGACUGAAAAAUACGCCAAUCA (((.(((.........)))..((((((((((((((((((.........)))))))).......((((.....))))........))))))))))......)))..... ( -31.50) >DroSim_CAF1 407 108 - 1 GGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGGCCCUCUGUAAGCAACAAGUGACUGAAAAAUACGCCAAUCA (((.(((.........)))..((((((((((((((((((.........)))))))).......((((.....))))........))))))))))......)))..... ( -31.50) >DroEre_CAF1 8759 108 - 1 GGUCGAUAUCAUACCCGCCAAUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGGCCCUCAGUAAGCAGCAAGUGACUGAAAAAUACGCCAAUGA ((.((...........(((........((((((((((((.........))))))))))))......)))..(((((..((.....)).)))))......))))..... ( -32.14) >DroYak_CAF1 7339 108 - 1 GGUCGACAUCAUUCCCGCCAAUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGGCCCUCAGUAAGCAACAAGUAACUGAAAAACACGCCAAUGA ((.((...........(((........((((((((((((.........))))))))))))......)))..(((((..((.....)).)))))......))))..... ( -31.44) >consensus GGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGGCCCUCUGUAAGCAACAAGUGACUGAAAAAUACGCCAAUCA (((.(....)...........((((((((((((((((((.........))))))))...........((.........))....))))))))))......)))..... (-19.58 = -20.08 + 0.51)



| Location | 7,392,816 – 7,392,922 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 83.06 |
| Mean single sequence MFE | -35.15 |
| Consensus MFE | -21.03 |
| Energy contribution | -22.15 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581714 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
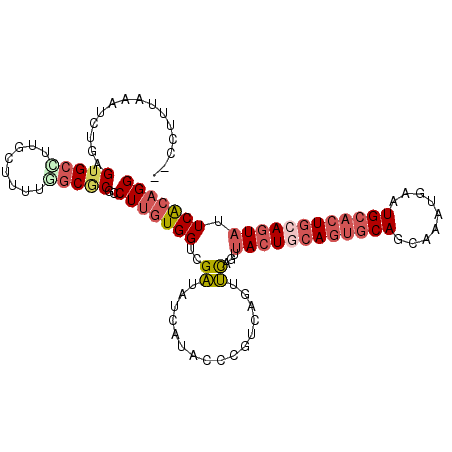
>2R_DroMel_CAF1 7392816 106 + 20766785 CCUGCGAAUACUGCAGUGCAUUCAUUUGCUGCACUGUAGUAACUGAGCUAACGGGUAUGAUAUCGACCACAAGGCGCGCCAAAAGCAAUGCACUCAGAUUUAAAGG-- (((..((((.(((.((((((((((.(((((((...))))))).)))(((....(((.((....)))))....((....))...))).))))))))))))))..)))-- ( -32.20) >DroGri_CAF1 8647 108 + 1 CCUGUGAGUAUUGCAGAGCAUCCAUGUGCUGUAGUUGAGUCACAACUCGCUUGGGUAUCUGGGCGACCAGCAGCUGAGCCAACAAGGACGCACUAAGCGCCAAGGCAG (((((((.((((((((.(((....))).)))))))..).)))).....(((..(.(..((((....)))).).)..)))......((.(((.....))))).)))... ( -34.60) >DroSec_CAF1 9017 106 + 1 CCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACCACAAGGCGCGCCAAAAGCAAGGCACUCAGAUUUAAAGG-- (((.((((((((((((((((.........)))))))))))).((((.((....(((.((....)))))...))....(((........)))..)))).)))).)))-- ( -34.50) >DroSim_CAF1 447 106 + 1 CCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACCACAAGGCGCGUCAAAAGCAAGGCACUCAGAUUUAAAGG-- (((.(((((.(((.(((((.((((.(((((((...))))))).)))).(((((.((.((........))....)).)))))........))))))))))))).)))-- ( -34.70) >DroEre_CAF1 8799 106 + 1 CCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAAUUGGCGGGUAUGAUAUCGACCACAAGGCGCGCCAAAAGCAAAGCACUCAGAUUUAAAGG-- (((.(((((.(((.(((((.((((.(((((((...))))))).))))((((((.((.((........))....)).)))))).......))))))))))))).)))-- ( -38.20) >DroYak_CAF1 7379 106 + 1 CCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAAUUGGCGGGAAUGAUGUCGACCACAAGGCGUGCUAAAAGCAAAGCACUCAGAUUUAAAGG-- (((.((((((((((((((((.........)))))))))))).((((..((((((.(....).))).)))......(((((........))))))))).)))).)))-- ( -36.70) >consensus CCUGUGAAUACUGCAGUGCAUUCAUUUGCUGCACUGCAGUAACUGAACUGACGGGUAUGAUAUCGACCACAAGGCGCGCCAAAAGCAAAGCACUCAGAUUUAAAGG__ (((.((((((((((((((((.........)))))))))))).((((.......(((.((....))))).....((.........)).......)))).)))).))).. (-21.03 = -22.15 + 1.12)

| Location | 7,392,816 – 7,392,922 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 83.06 |
| Mean single sequence MFE | -32.24 |
| Consensus MFE | -21.91 |
| Energy contribution | -23.39 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.68 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513361 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
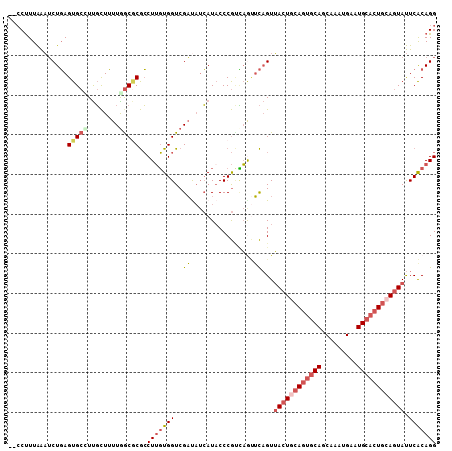
>2R_DroMel_CAF1 7392816 106 - 20766785 --CCUUUAAAUCUGAGUGCAUUGCUUUUGGCGCGCCUUGUGGUCGAUAUCAUACCCGUUAGCUCAGUUACUACAGUGCAGCAAAUGAAUGCACUGCAGUAUUCGCAGG --(((......((((((.....((.....))(((...((((((....))))))..)))..)))))).((((.(((((((.........))))))).)))).....))) ( -26.90) >DroGri_CAF1 8647 108 - 1 CUGCCUUGGCGCUUAGUGCGUCCUUGUUGGCUCAGCUGCUGGUCGCCCAGAUACCCAAGCGAGUUGUGACUCAACUACAGCACAUGGAUGCUCUGCAAUACUCACAGG ..((....))((..((.((((((.((((((((......((((....)))).......))).(((((.....))))).)))))...)))))).))))............ ( -31.62) >DroSec_CAF1 9017 106 - 1 --CCUUUAAAUCUGAGUGCCUUGCUUUUGGCGCGCCUUGUGGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGG --.............(((((........)))))..(((((((..(((.........)))........((((((((((((.........)))))))))))).))))))) ( -35.30) >DroSim_CAF1 447 106 - 1 --CCUUUAAAUCUGAGUGCCUUGCUUUUGACGCGCCUUGUGGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGG --.........(((........(((.((((((.(...((((((....)))))).)))))))...)))((((((((((((.........))))))))))))....))). ( -31.30) >DroEre_CAF1 8799 106 - 1 --CCUUUAAAUCUGAGUGCUUUGCUUUUGGCGCGCCUUGUGGUCGAUAUCAUACCCGCCAAUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGG --(((......((((((((...))...(((((.(...((((((....)))))).)))))))))))).((((((((((((.........)))))))))))).....))) ( -34.50) >DroYak_CAF1 7379 106 - 1 --CCUUUAAAUCUGAGUGCUUUGCUUUUAGCACGCCUUGUGGUCGACAUCAUUCCCGCCAAUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGG --(((......(((((((((........)))))......(((.((..........)))))..)))).((((((((((((.........)))))))))))).....))) ( -33.80) >consensus __CCUUUAAAUCUGAGUGCCUUGCUUUUGGCGCGCCUUGUGGUCGAUAUCAUACCCGUCAGUUCAGUUACUGCAGUGCAGCAAAUGAAUGCACUGCAGUAUUCACAGG ...............(((((........)))))..(((((((..((................))...((((((((((((.........)))))))))))).))))))) (-21.91 = -23.39 + 1.48)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:17:21 2006