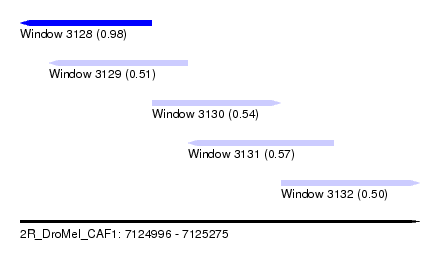
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,124,996 – 7,125,275 |
| Length | 279 |
| Max. P | 0.977554 |

| Location | 7,124,996 – 7,125,088 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 83.10 |
| Mean single sequence MFE | -37.07 |
| Consensus MFE | -30.23 |
| Energy contribution | -30.65 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.24 |
| Mean z-score | -4.71 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977554 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
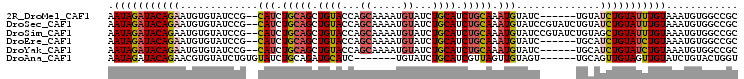
>2R_DroMel_CAF1 7124996 92 - 20766785 CAAGUAUCUGCGAGUGUAUCUAUGUAUCUA--UGCUAGGUGCGUGGGCGUGGCACUCCGCCGCUGCUCAUUAGAUUU----ACUUGAGAUACUUUCCA .((((((((.((((((.(((((.(((((((--...)))))))(((((((((((.....))))).))))))))))).)----))))))))))))).... ( -40.30) >DroSec_CAF1 152568 92 - 1 CAAGUAUCUGCGAGUGUAUCUAUGCAUCUA--UGCUAGGUGCGUGGGCGUGGCACACCGUCGCUGCUCAUUAGAUUU----ACUUGAGAUACUUUCCA .((((((((.((((((.(((((.(((((((--...)))))))(((((((((((.....))))).))))))))))).)----))))))))))))).... ( -39.60) >DroSim_CAF1 151028 92 - 1 CAAGUAUCUGCGAGUGUAUCUAUGCAUCUA--UGCUAGGUGCGUGGGCGUGGCACACCGUCGCUGCUCAUUAGAUUC----ACUUGAGAUACUUUCCA .((((((((.((((((.(((((.(((((((--...)))))))(((((((((((.....))))).))))))))))).)----))))))))))))).... ( -41.30) >DroEre_CAF1 158259 90 - 1 CAAGUAUUUGCGAGUGUAUCUAUGUAUC----UGCUAGAUGCGUGGGCGUGGCAGCCCGCCGCUGCUCAUUAGAUUU----ACUUGAGAUACUUUCCA .((((((((.((((((.(((((.(((((----.....)))))(((((((((((.....))))).))))))))))).)----))))))))))))).... ( -37.20) >DroYak_CAF1 162257 90 - 1 CAAGUAUCUGCGAGUGUAUCUAUGUAUC----UGCUAGAUGCGUGGGCGUGGCACCCCGCCACUGCUCAUUAGAUUU----ACUUGAGAUACUUUCCA .((((((((.((((((.(((((.(((((----.....)))))(((((((((((.....))))).))))))))))).)----))))))))))))).... ( -40.20) >DroAna_CAF1 154648 85 - 1 AAUGUAUCUC----UGUAUCUGUAAAUGUAUGUGGCAGAUGUGUGGGCGCGGUUCC---GCGAUGCUCAUUAGAUUUUGAGACUUGAGAUAC------ ...(((((((----..(((((((...........))))))).(((((((((....)---))...))))))...............)))))))------ ( -23.80) >consensus CAAGUAUCUGCGAGUGUAUCUAUGUAUCUA__UGCUAGAUGCGUGGGCGUGGCACCCCGCCGCUGCUCAUUAGAUUU____ACUUGAGAUACUUUCCA .((((((((.((((((((((((.(((......))))))))))(((((((((((.....))))).))))))...........))))))))))))).... (-30.23 = -30.65 + 0.42)

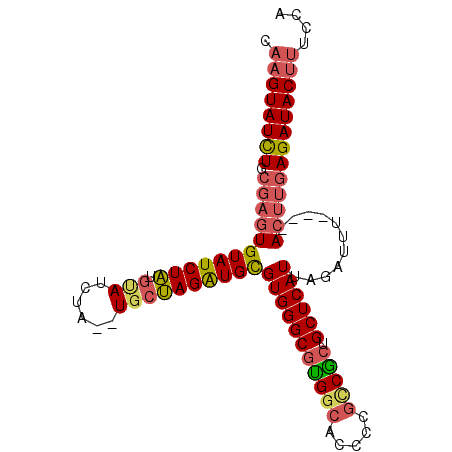
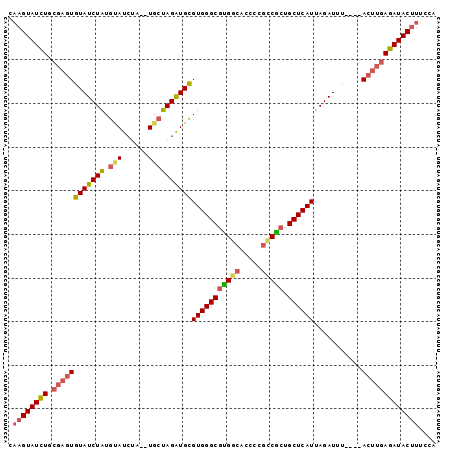
| Location | 7,125,016 – 7,125,113 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.62 |
| Mean single sequence MFE | -32.02 |
| Consensus MFE | -22.94 |
| Energy contribution | -23.33 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.13 |
| Structure conservation index | 0.72 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510495 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7125016 97 - 20766785 GCUGCUCU---------------UGCAUCCGCAAAAUGUACAAGUAUCUGCGAGUGUAUCUAUGUAUCUA--UGCUAGGUGCGUGGGCGUGGCACUCCGCCGCUGCUCAUUAGA ........---------------((((..((((....(((....))).))))..))))((((.(((((((--...)))))))(((((((((((.....))))).)))))))))) ( -30.40) >DroSec_CAF1 152588 97 - 1 GCUGCUCU---------------UGCAUCCGCAAAGUGUACAAGUAUCUGCGAGUGUAUCUAUGCAUCUA--UGCUAGGUGCGUGGGCGUGGCACACCGUCGCUGCUCAUUAGA ((((((..---------------(((((.......)))))..))))...))((((....(((((((((((--...)))))))))))((((((....))).))).))))...... ( -29.90) >DroSim_CAF1 151048 97 - 1 GCUGCUCU---------------UGCAUCCGCAAAGUGUACAAGUAUCUGCGAGUGUAUCUAUGCAUCUA--UGCUAGGUGCGUGGGCGUGGCACACCGUCGCUGCUCAUUAGA ((((((..---------------(((((.......)))))..))))...))((((....(((((((((((--...)))))))))))((((((....))).))).))))...... ( -29.90) >DroEre_CAF1 158279 95 - 1 GCUGCUCU---------------UGCGUCCGCAAAAUGUACAAGUAUUUGCGAGUGUAUCUAUGUAUC----UGCUAGAUGCGUGGGCGUGGCAGCCCGCCGCUGCUCAUUAGA ((((((..---------------.((((((((....((((........))))...(((((((.((...----.)))))))))))))))))))))))..((....))........ ( -31.20) >DroYak_CAF1 162277 94 - 1 GCUGCUCU---------------UGCAUCCGCA-AAUGUACAAGUAUCUGCGAGUGUAUCUAUGUAUC----UGCUAGAUGCGUGGGCGUGGCACCCCGCCACUGCUCAUUAGA ........---------------((((..((((-.(((......))).))))..))))((((.(((((----.....)))))(((((((((((.....))))).)))))))))) ( -31.80) >DroAna_CAF1 154666 106 - 1 GCUGCUGUCACUGGCGCUGCCUGUGCAUCCGCAGA-UACAAAUGUAUCUC----UGUAUCUGUAAAUGUAUGUGGCAGAUGUGUGGGCGCGGUUCC---GCGAUGCUCAUUAGA ((.(((..(((.....((((((((((((..(((((-((((..........----)))))))))..))))))).)))))..)))..)))(((....)---))...))........ ( -38.90) >consensus GCUGCUCU_______________UGCAUCCGCAAAAUGUACAAGUAUCUGCGAGUGUAUCUAUGUAUCUA__UGCUAGAUGCGUGGGCGUGGCACCCCGCCGCUGCUCAUUAGA ...((((................(((....)))....(((........)))))))(((((((.(((......))))))))))(((((((((((.....))))).)))))).... (-22.94 = -23.33 + 0.39)

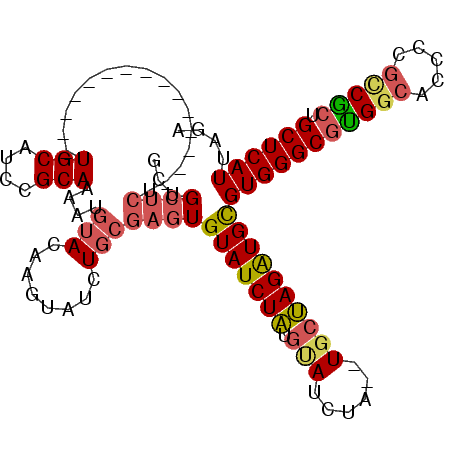
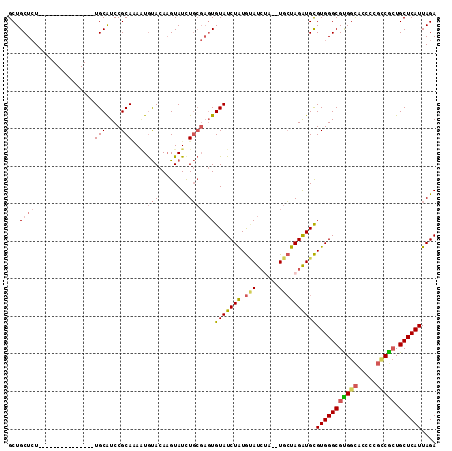
| Location | 7,125,088 – 7,125,178 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 86.43 |
| Mean single sequence MFE | -25.68 |
| Consensus MFE | -20.00 |
| Energy contribution | -20.36 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535793 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7125088 90 + 20766785 UACAUUUUGCGGAUGCA---------------AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUUCC-CAUACGCCGCACAGA .......(((((.(((.---------------...)))((((.(((..(((((.......))))).))))))).................-......))))).... ( -23.60) >DroSec_CAF1 152660 90 + 1 UACACUUUGCGGAUGCA---------------AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUUCC-CAUACGCCGCACAGA ....((.(((((.(((.---------------...)))((((.(((..(((((.......))))).))))))).................-......))))).)). ( -25.40) >DroSim_CAF1 151120 82 + 1 UACACUUUGCGGAUGCA---------------AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUA---------CCGCACAGA ....((.(((((.(((.---------------...)))((((.(((..(((((.......))))).)))))))...............---------))))).)). ( -25.70) >DroEre_CAF1 158349 90 + 1 UACAUUUUGCGGACGCA---------------AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUUCC-CAUACGCCGCACAGA .......(((.(((((.---------------......)))))((((...(((((........))))).(((.(((((.....)))))..-))).))))))).... ( -24.30) >DroYak_CAF1 162347 89 + 1 UACAUU-UGCGGAUGCA---------------AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUUCC-CAUACGCCGCACAGA ......-(((((.(((.---------------...)))((((.(((..(((((.......))))).))))))).................-......))))).... ( -23.60) >DroAna_CAF1 154733 105 + 1 UGUA-UCUGCGGAUGCACAGGCAGCGCCAGUGACAGCAGCGCCAGCUACAGCUCAUCCAGAGCUGAGCCAUGCAAAUUACAACAAUUUCCCCGUACGCCGCACAGA ....-..(((((.((((..(((.((((........)).))))).(((.((((((.....)))))))))..))))...(((............)))..))))).... ( -31.50) >consensus UACAUUUUGCGGAUGCA_______________AGAGCAGCGUCGGCGACAGCUCAUCCAAAGUUGAGCCAUGCAAAUUACAACAAUUUCC_CAUACGCCGCACAGA .......(((((.(((...................)))((((.(((..(((((.......))))).)))))))........................))))).... (-20.00 = -20.36 + 0.36)

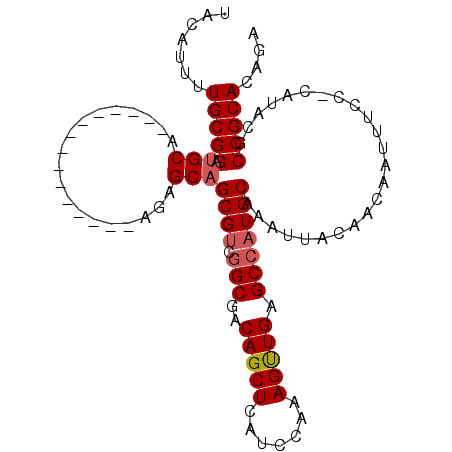

| Location | 7,125,113 – 7,125,215 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 87.17 |
| Mean single sequence MFE | -32.17 |
| Consensus MFE | -25.48 |
| Energy contribution | -24.73 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.570762 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7125113 102 - 20766785 UACAGCUGCAGAUG--CGGAUACACAUUCUGUAUCUAUUUCUGUGCGGCGUAUG-GGAAAUUGUUGUAAUUUGCAUGGCUCAACUUUGGAUGAGCUGUCGCCGAC (((((..(.(((((--((((.......))))))))).)..)))))(((((....-(.((((((...)))))).)((((((((.(....).))))))))))))).. ( -33.20) >DroPse_CAF1 158563 100 - 1 --CAGAUACAGAUA--CAGAUACACAUUCUGUAUCUAUUUCUGUGCGGUGUACG-GGAAAUUGUUGUAAUUUGCAUGGCUUAACGCUGGAUGAGCUGCCGCUGCC --((((...(((((--((((.......)))))))))...)))).(((((.....-(.((((((...)))))).)..((((((.(....).))))))))))).... ( -30.50) >DroSim_CAF1 151145 94 - 1 UACAGCUGCAGAUG--CGGAUACACAUUCUGUAUCUAUUUCUGUGCGG---------UAAUUGUUGUAAUUUGCAUGGCUCAACUUUGGAUGAGCUGUCGCCGAC (((((..(.(((((--((((.......))))))))).)..)))))(((---------(...(((........))).((((((.(....).))))))...)))).. ( -27.90) >DroYak_CAF1 162371 102 - 1 UACAGCUGCAGAUG--CGGAUACACAUUCUGUAUCUAUUUCUGUGCGGCGUAUG-GGAAAUUGUUGUAAUUUGCAUGGCUCAACUUUGGAUGAGCUGUCGCCGAC (((((..(.(((((--((((.......))))))))).)..)))))(((((....-(.((((((...)))))).)((((((((.(....).))))))))))))).. ( -33.20) >DroAna_CAF1 154772 105 - 1 UGCAUCUGCAGAUACACAGAUACACGUUCUGUAUCUAUUUCUGUGCGGCGUACGGGGAAAUUGUUGUAAUUUGCAUGGCUCAGCUCUGGAUGAGCUGUAGCUGGC .((...((((((((((.(((((((.....))))))).(..((((((...))))))..)......))).))))))).((((((((((.....)))))).)))).)) ( -37.70) >DroPer_CAF1 157884 100 - 1 --CAGAUACAGAUA--CAGAUACACAUUCUGUAUCUAUUUCUGUGCGGUGUACG-GGAAAUUGUUGUAAUUUGCAUGGCUUAACGCUGGAUGAGCUGCCGCUGCC --((((...(((((--((((.......)))))))))...)))).(((((.....-(.((((((...)))))).)..((((((.(....).))))))))))).... ( -30.50) >consensus UACAGCUGCAGAUA__CAGAUACACAUUCUGUAUCUAUUUCUGUGCGGCGUACG_GGAAAUUGUUGUAAUUUGCAUGGCUCAACUCUGGAUGAGCUGUCGCCGAC .......(((((.....(((((((.....)))))))...)))))(((((............(((........))).((((((.(....).))))))))))).... (-25.48 = -24.73 + -0.75)
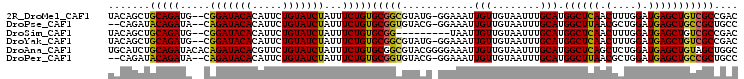
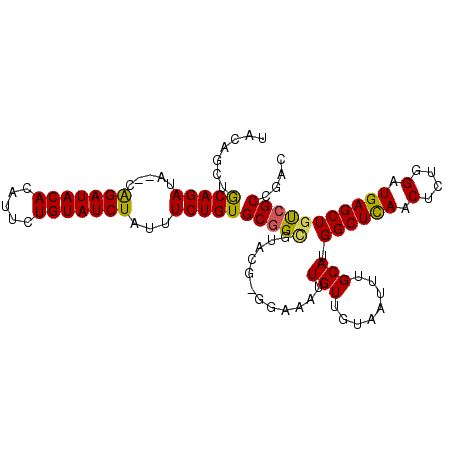
| Location | 7,125,178 – 7,125,275 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 84.99 |
| Mean single sequence MFE | -29.42 |
| Consensus MFE | -18.67 |
| Energy contribution | -19.23 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.93 |
| Structure conservation index | 0.63 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7125178 97 + 20766785 AAUAGAUACAGAAUGUGUAUCCG--CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUC------UGUAUCUGUAUUUGUAAAUGUGGCCGC .((((((((((((((((....))--))).(((((.((((...((.....))...)))).)))))......))------))))))))).................. ( -26.90) >DroSec_CAF1 152750 103 + 1 AAUAGAUACAGAAUGUGUAUCCG--CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUCCGUAUCUGUAUCUGUAUUUGUAAAUGUGGCCGC .((((((((((((((..(....(--(((.(((((.((((...((.....))...)))).))))).)))))..))).))))))))))).................. ( -27.90) >DroSim_CAF1 151202 103 + 1 AAUAGAUACAGAAUGUGUAUCCG--CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUCCGUAUCUGUAGCUGUAUUUGUAAAUGUGGCCGC .((((((((.(((((((....))--))).(((((.((((...((.....))...)))).)))))......)).)))))))).((..((((....))))..))... ( -27.50) >DroEre_CAF1 158439 97 + 1 AAUAGAUACAGAAUGUGUAUCCG--CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUC------UGCAUCUGUAUCUGUAAAUGUGGCCGC .(((((((((((...((((...(--(((.(((((.((((...((.....))...)))).))))).))))...------)))))))))))))))............ ( -30.10) >DroYak_CAF1 162436 97 + 1 AAUAGAUACAGAAUGUGUAUCCG--CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUC------UGCAUCUGUAUCUGUAAAUGUGGCCGC .(((((((((((...((((...(--(((.(((((.((((...((.....))...)))).))))).))))...------)))))))))))))))............ ( -30.10) >DroAna_CAF1 154838 92 + 1 AAUAGAUACAGAACGUGUAUCUGUGUAUCUGCAGAUGCAUC-------UGUAUCUGCAUCGUUAGUUGUAGU------UGCAGUUGUAGUUGUAUCUGUACUGGU .((((((((((.((((((((((((......))))))))))(-------((((.(((((.(....).))))).------)))))..))..))))))))))...... ( -34.00) >consensus AAUAGAUACAGAAUGUGUAUCCG__CAUCUGCAGCUGUACCAGCAAAAUGUAUCUGCAUCUGCAAAUGUAUC______UGCAUCUGUAUUUGUAAAUGUGGCCGC .(((((((((((.............(((.(((((.((((...((.....))...)))).))))).)))..............)))))))))))............ (-18.67 = -19.23 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:40 2006