
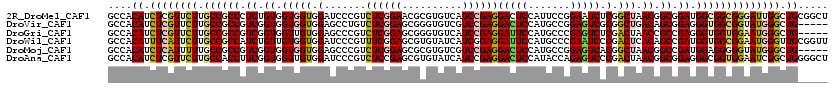
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 7,042,310 – 7,042,430 |
| Length | 120 |
| Max. P | 0.500000 |

| Location | 7,042,310 – 7,042,430 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.63 |
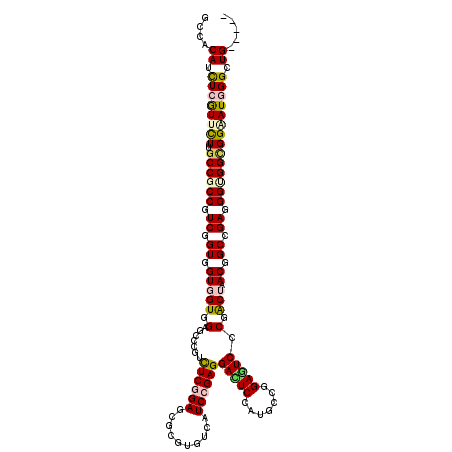
| Mean single sequence MFE | -53.65 |
| Consensus MFE | -35.30 |
| Energy contribution | -35.80 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 7042310 120 + 20766785 GCCACAUCUCGUUCUUGCCGCCCUCUGUGGUGGUGGAUCCCGUCUCGGAACGCGUGUCAUCCGAGGACUCCAUUCCGGAAUCUCGGCUAACGGCGGAUGGCGGCGGGAUUGGCUGCGGCU (((.((.(.((.((((((((((.(((((.(((((.((......((((((((....))..))))))((.(((.....))).)))).))).)).))))).)))))))))).))).)).))). ( -53.40) >DroVir_CAF1 88099 115 + 1 GCCACAUCUCGUUCUUGCCGCCGUCGGUGGUGGUGGAGCCUGUCUCGGAGCGGGUGUCGUCCGAGGACUCCAUGCCGGAGUCGCGGCUGACAGCGGAGGGUGGCGGUAUGGGCUG----- (((.(((..((((((..((((.((((((.((((((..((((((......))))))..)).))...((((((.....)))))))).)))))).))))..)).))))..))))))..----- ( -56.30) >DroGri_CAF1 98792 115 + 1 GCCACAUCUCGUUCUUGCCGCCGUCGGUGGUUGUGGAGCCCGUCUCGGAGCGGGUGUCAUCCGAGGAUUCCAUGCCCGAGUCUCGACUAACCGCCGAGGGUGGUGGAAUGGGCUG----- ....((.((((((((.((((((.(((((((((((.(((.....(((((.(((((.(((.......))).)).)))))))).))).)).))))))))).)))))))))))))).))----- ( -58.60) >DroWil_CAF1 103395 120 + 1 GCCACAUUUCAUUCUUGCCGCCAUCUGUUGUGGUGGAUCCCGUUUCGGAGCGUGUAUCAUCGGAGGAUUCCAUGCCCGAAUCCCGACUCACAGCCGAUGGUGGCGGAAUGGGUUGCGGUU (((.((.((((((((.(((((((((.((((((((((((......((((.(((((.(((.......)))..)))))))))))))..)).)))))).))))))))))))))))).)).))). ( -55.20) >DroMoj_CAF1 101978 115 + 1 GCCACAUCUCAUUUUUGCCGCCGUCGGUGGUGGUGGAGCCCGUCUCGGAGCGCGUGUCGUCCGAGGACUCCAUGCCGGAGUCACGGCUAACGGCCGAUGGAGGGGGUAUGGGCUG----- (((.((((((.......((.((((((((.((.((((((....(((((((.((.....))))))))).))))))((((......))))..)).)))))))).))))).))))))..----- ( -53.51) >DroAna_CAF1 71154 120 + 1 GCCACAUCUCGUUCUUGCCACCUUCGGUGGUUGUGGAUCCCGUCUCCGAGCGUGUAUCAUCCGAGGACUCCAUACCAGAGUCCCGACUAACGGCGGAGGGCGGUGGAAUCGGCUGGGGCU (((.((.((.(((((.(((.(((((.((.((((..(((.((((......))).).)))...((.((((((.......))))))))..)))).)).))))).)))))))).)).)).))). ( -44.90) >consensus GCCACAUCUCGUUCUUGCCGCCGUCGGUGGUGGUGGAGCCCGUCUCGGAGCGCGUGUCAUCCGAGGACUCCAUGCCGGAGUCCCGACUAACGGCCGAGGGUGGCGGAAUGGGCUG_____ ....((.((((((((.((((((.((.((.(((((.(.......((((((..........))))))(((((.......))))).).))).)).)).)).)))))))))))))).))..... (-35.30 = -35.80 + 0.50)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:14 2006