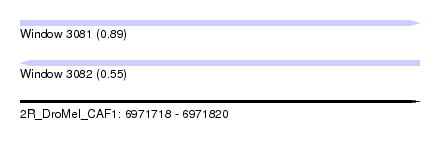
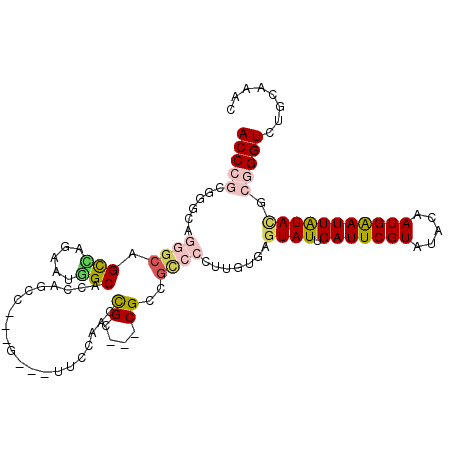
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,971,718 – 6,971,820 |
| Length | 102 |
| Max. P | 0.892490 |

| Location | 6,971,718 – 6,971,820 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 77.01 |
| Mean single sequence MFE | -42.80 |
| Consensus MFE | -14.62 |
| Energy contribution | -16.02 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.34 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892490 |
| Prediction | RNA |
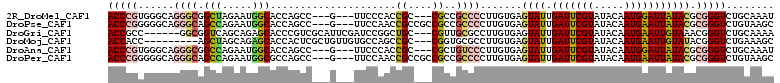
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6971718 102 + 20766785 AUUUGCAGACCCGCGUAUAAUCCAUUGUAUACGAAUCAAUACUCACAAGGGGCGGCG---GCGGUGGGAA---C---GGCUGGUGCCAUUCUAGCCGCCCUGCCCACGGGU ........(((((((((((........))))))................((((((.(---(((((.((((---.---(((....))).)))).)))))))))))).))))) ( -47.00) >DroPse_CAF1 1325 105 + 1 GCUUACAGACCCGCGUAUAAUUCAUUGUAUACGAAUCAAUACUCACAAGGGGCGGCGGCGGCGGUUGGAA---C---GGCUGGUGCCAUUCUGGCUGCCCUGCCCCCGGGU ........((((.((((((........))))))................(((.(((((.((((((..(((---.---(((....))).)))..)))))))))))))))))) ( -49.40) >DroGri_CAF1 1276 102 + 1 UUUUGCAGACCCGUUUACAAUUCAUUGUAUACGAAUCAAUACUCACAAGGCGCAACG---GCAGCCGGAUCGAAUGCGACGGGUGCUCUGCUGACCGCC------GGCGGU ....(((((((((((....((((...((((........))))......(((((....---)).))).....))))..))))))...)))))..((((..------..)))) ( -30.20) >DroMoj_CAF1 1624 99 + 1 GCUUUCAGACCCGUAUACAAUUCAUUGUAUACGAAUCAAUACUCACAAGGCGCACCG---GCGGCUGGCACAACAGCGAGUGGUGCUCUGCUAGCU---------GGUGGU (((((..((..(((((((((....)))))))))..)).........))))).(((((---(((((.(((((.((.....)).)))))..))).)))---------)))).. ( -35.20) >DroAna_CAF1 1290 102 + 1 AUUUGCAGACCCGCGUAUAAUCCAUUGUAUACGAAUCAAUACUCACAAGGGACAGCG---GCGGUGGGAA---C---GGCUGGUGCCAUUCUGGCCGCCCUGCCCACGGGU ........(((((((((((........))))))...............(((.(((.(---(((((.((((---.---(((....))).)))).)))))))))))).))))) ( -46.20) >DroPer_CAF1 1317 105 + 1 GCUUACAGACCCGCGUAUAAUUCAUUGUAUACGAAUCAAUACUCACAAGGGGCGGCGGCGGCGGUUGGAA---C---GGCUGGCGCCAUUCUGGCUGCCCUGCCCCCGGGU ........((((.((((((........))))))................(((.(((((.((((((..(((---.---(((....))).)))..)))))))))))))))))) ( -48.80) >consensus GCUUGCAGACCCGCGUAUAAUUCAUUGUAUACGAAUCAAUACUCACAAGGGGCAGCG___GCGGUUGGAA___C___GGCUGGUGCCAUUCUGGCCGCCCUGCCCACGGGU ........((((((((((((....))))))...........(((....)))...))....((((((((((.......(((....))).)))))))))).........)))) (-14.62 = -16.02 + 1.39)

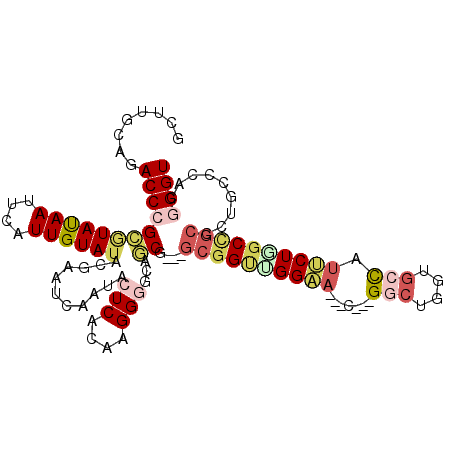

| Location | 6,971,718 – 6,971,820 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 77.01 |
| Mean single sequence MFE | -40.50 |
| Consensus MFE | -14.73 |
| Energy contribution | -15.52 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.36 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.546061 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6971718 102 - 20766785 ACCCGUGGGCAGGGCGGCUAGAAUGGCACCAGCC---G---UUCCCACCGC---CGCCGCCCCUUGUGAGUAUUGAUUCGUAUACAAUGGAUUAUACGCGGGUCUGCAAAU ((((((((((.((((((...(((((((....)))---)---)))...))))---).).)))).......((((.(((((((.....)))))))))))))))))........ ( -44.80) >DroPse_CAF1 1325 105 - 1 ACCCGGGGGCAGGGCAGCCAGAAUGGCACCAGCC---G---UUCCAACCGCCGCCGCCGCCCCUUGUGAGUAUUGAUUCGUAUACAAUGAAUUAUACGCGGGUCUGUAAGC ((((((((((.((((.((..(((((((....)))---)---))).....)).))).).)))))......((((.(((((((.....))))))))))).)))))........ ( -43.10) >DroGri_CAF1 1276 102 - 1 ACCGCC------GGCGGUCAGCAGAGCACCCGUCGCAUUCGAUCCGGCUGC---CGUUGCGCCUUGUGAGUAUUGAUUCGUAUACAAUGAAUUGUAAACGGGUCUGCAAAA ..(((.------((((..((((.(.(((.((((((....)))..))).)))---)))))))))..))).(((..(((((((.(((((....))))).)))))))))).... ( -31.20) >DroMoj_CAF1 1624 99 - 1 ACCACC---------AGCUAGCAGAGCACCACUCGCUGUUGUGCCAGCCGC---CGGUGCGCCUUGUGAGUAUUGAUUCGUAUACAAUGAAUUGUAUACGGGUCUGAAAGC ......---------.(((.((((.(((((....((((......))))...---.)))))...)))).)))...(((((((((((((....)))))))))))))....... ( -33.40) >DroAna_CAF1 1290 102 - 1 ACCCGUGGGCAGGGCGGCCAGAAUGGCACCAGCC---G---UUCCCACCGC---CGCUGUCCCUUGUGAGUAUUGAUUCGUAUACAAUGGAUUAUACGCGGGUCUGCAAAU (((((((((((((((((...(((((((....)))---)---)))...))))---).)))))).......((((.(((((((.....)))))))))))))))))........ ( -46.70) >DroPer_CAF1 1317 105 - 1 ACCCGGGGGCAGGGCAGCCAGAAUGGCGCCAGCC---G---UUCCAACCGCCGCCGCCGCCCCUUGUGAGUAUUGAUUCGUAUACAAUGAAUUAUACGCGGGUCUGUAAGC ((((((((((.((((.((..(((((((....)))---)---))).....)).))).).)))))......((((.(((((((.....))))))))))).)))))........ ( -43.80) >consensus ACCCGCGGGCAGGGCAGCCAGAAUGGCACCAGCC___G___UUCCAACCGC___CGCCGCCCCUUGUGAGUAUUGAUUCGUAUACAAUGAAUUAUACGCGGGUCUGCAAAC (((((......((((.(((.....))).....................((....))..)))).......((((.(((((((.....))))))))))).)))))........ (-14.73 = -15.52 + 0.78)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:14:53 2006