| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,969,782 – 6,969,872 |
| Length | 90 |
| Max. P | 0.974429 |

| Location | 6,969,782 – 6,969,872 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 75.55 |
| Mean single sequence MFE | -40.57 |
| Consensus MFE | -34.02 |
| Energy contribution | -33.88 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.973106 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
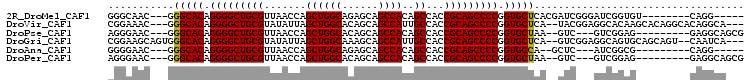
>2R_DroMel_CAF1 6969782 90 + 20766785 GGGCAAC---GGGCACAGGGGCUGCGUUAACCAGCUGGCAGAGCAGCCACAGCCACCGCAGCCCCGGUGCUCACGAUCGGGAUCGGUGU--------CAGG----- .((((..---((((((.(((((((((.......((((((......)))..)))...))))))))).)))))).((((....)))).)))--------)...----- ( -43.70) >DroVir_CAF1 16350 98 + 1 CGGAAAC---GGGCACAGGGGCUGCGUAUAUUAGCUGGCACAGCAGCCAUUGCCACCGCAGCCCCGGUGCUCA--UACGGAGGCACAAGCACAGGCACAGGCA--- .(....)---((((((.(((((((((.....(((.((((......)))))))....))))))))).)))))).--.......((....((....))....)).--- ( -41.60) >DroPse_CAF1 15028 89 + 1 AGGGAAC---GGGCACAGGGGCUGCGUUAACCAGCUGGCACAGCAGCCACAGCCACCGCAGCCCCGGUGCUAA--GUC---GUCGGAG---------GAGGCAGCG ...((..---.(((((.(((((((((.......((((((......)))..)))...))))))))).)))))..--.))---(((....---------..))).... ( -38.90) >DroGri_CAF1 18298 99 + 1 CGGAAGCAGUGGGCACAGGGGCUGCGUAUAUUAGCUGGCAAAGCAGCCAUUGCCACCGCAGCCCCGGUGCUCA--GUCGGAGGCAGUGCAGCAGU--CAAUCA--- .((..((..(((((((.((((((((((......))((((((........))))))..)))))))).)))))))--((.....))......))..)--).....--- ( -41.40) >DroAna_CAF1 24348 84 + 1 GGGGAAC---GGGCACAGGGGCUGCGUUAACCAGCUGGCAGAGCAGCCACAGCCACCGCAGCCCCGGUGCCA--GCUC---AUCGGCG---------CAGG----- .(((...---.(((((.(((((((((.......((((((......)))..)))...))))))))).))))).--.)))---.......---------....----- ( -38.90) >DroPer_CAF1 14982 89 + 1 AGGGAAC---GGGCACAGGGGCUGCGUUAACCAGCUGGCACAGCAGCCACAGCCACCGCAGCCCCGGUGCUAA--GUC---GUCGGAG---------GAGGCAGCG ...((..---.(((((.(((((((((.......((((((......)))..)))...))))))))).)))))..--.))---(((....---------..))).... ( -38.90) >consensus AGGGAAC___GGGCACAGGGGCUGCGUUAACCAGCUGGCACAGCAGCCACAGCCACCGCAGCCCCGGUGCUAA__GUC___GUCGGAG_________CAGGCA___ ...........(((((.(((((((((.......((((((......))))..))...))))))))).)))))................................... (-34.02 = -33.88 + -0.14)

| Location | 6,969,782 – 6,969,872 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 75.55 |
| Mean single sequence MFE | -41.70 |
| Consensus MFE | -36.29 |
| Energy contribution | -36.07 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.73 |
| SVM RNA-class probability | 0.974429 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
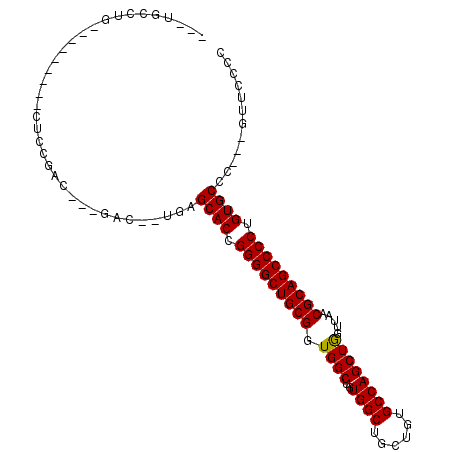
>2R_DroMel_CAF1 6969782 90 - 20766785 -----CCUG--------ACACCGAUCCCGAUCGUGAGCACCGGGGCUGCGGUGGCUGUGGCUGCUCUGCCAGCUGGUUAACGCAGCCCCUGUGCCC---GUUGCCC -----...(--------((..((((....)))).(.((((.(((((((((.((((((((((......)))).).))))).))))))))).)))).)---))).... ( -42.10) >DroVir_CAF1 16350 98 - 1 ---UGCCUGUGCCUGUGCUUGUGCCUCCGUA--UGAGCACCGGGGCUGCGGUGGCAAUGGCUGCUGUGCCAGCUAAUAUACGCAGCCCCUGUGCCC---GUUUCCG ---.....(..(..(((((..(((....)))--..))))).(((((((((.((((..((((......)))))))).....))))))))).)..)..---....... ( -44.70) >DroPse_CAF1 15028 89 - 1 CGCUGCCUC---------CUCCGAC---GAC--UUAGCACCGGGGCUGCGGUGGCUGUGGCUGCUGUGCCAGCUGGUUAACGCAGCCCCUGUGCCC---GUUCCCU .........---------....(((---(..--...((((.(((((((((.((((((((((......)))).).))))).))))))))).)))).)---))).... ( -38.80) >DroGri_CAF1 18298 99 - 1 ---UGAUUG--ACUGCUGCACUGCCUCCGAC--UGAGCACCGGGGCUGCGGUGGCAAUGGCUGCUUUGCCAGCUAAUAUACGCAGCCCCUGUGCCCACUGCUUCCG ---...(((--...((......))...))).--((.((((.(((((((((.((((..((((......)))))))).....))))))))).)))).))......... ( -39.60) >DroAna_CAF1 24348 84 - 1 -----CCUG---------CGCCGAU---GAGC--UGGCACCGGGGCUGCGGUGGCUGUGGCUGCUCUGCCAGCUGGUUAACGCAGCCCCUGUGCCC---GUUCCCC -----....---------.......---((((--.(((((.(((((((((.((((((((((......)))).).))))).))))))))).))))).---))))... ( -46.20) >DroPer_CAF1 14982 89 - 1 CGCUGCCUC---------CUCCGAC---GAC--UUAGCACCGGGGCUGCGGUGGCUGUGGCUGCUGUGCCAGCUGGUUAACGCAGCCCCUGUGCCC---GUUCCCU .........---------....(((---(..--...((((.(((((((((.((((((((((......)))).).))))).))))))))).)))).)---))).... ( -38.80) >consensus ___UGCCUG_________CUCCGAC___GAC__UGAGCACCGGGGCUGCGGUGGCUGUGGCUGCUGUGCCAGCUGGUUAACGCAGCCCCUGUGCCC___GUUCCCC ....................................((((.(((((((((.((((..((((......)))))))).....))))))))).))))............ (-36.29 = -36.07 + -0.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:14:50 2006