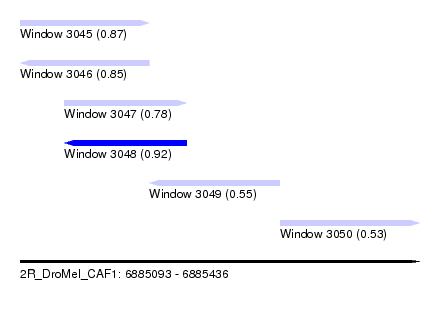
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,885,093 – 6,885,436 |
| Length | 343 |
| Max. P | 0.923661 |

| Location | 6,885,093 – 6,885,204 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.69 |
| Mean single sequence MFE | -51.08 |
| Consensus MFE | -31.20 |
| Energy contribution | -30.92 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.32 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874275 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885093 111 + 20766785 CCCAUCUCACCCGCAUCCUCA--GUCGCUGCCGCAGCUGCUGCUG-----UCAGC-AAGGCCAAUGCAAAUGCCCUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGC .....(((((..((......(--(((((((..(((((....))))-----)))))-.((((((..((....))..))))))....))))..(((...(((....))).)))))-))))). ( -44.50) >DroVir_CAF1 613 112 + 1 CCCGCCUUGCCG-----CUCAGUGCCGCUGGCAAGGCUUCGGUGGCGGCCUCGGUUAGUGCCAACGCCGCGGCGCUGGCGACGCUAGCUAG---UCUGCCCAUGGGCAUUGGCGGUCUGC .(((((.(((((-----(.(((((((((.(((.(((((.(....).))))).(((....)))...))))))))))))))(((........)---))........))))..)))))..... ( -56.80) >DroSec_CAF1 640 111 + 1 CCCAUCUCACCCGCAUCCUCA--GCCGCUGCCGCAGCUGCUGCUG-----UCAGC-AAGGCCAAUGCAAAUGCCCUGGCCUCACUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGC .....(((((..((......(--(((((((..(((((....))))-----)))))-.((((((..((....))..))))))....))))..(((...(((....))).)))))-))))). ( -47.20) >DroSim_CAF1 4753 111 + 1 CCCAUCUCACCCGCAUCCUCA--GCCGCUGCCGCAGCUGCUGCUG-----UCAGC-AAGGCCAAUGCAAAUGCGCUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGC .....(((((..((......(--(((((((..(((((....))))-----)))))-.((((((.(((....))).))))))....))))..(((...(((....))).)))))-))))). ( -48.80) >DroEre_CAF1 6086 111 + 1 CCCAUCUCACCCGCAUCCUCG--GCCGCUGCCGCAGCUGCUGCCG-----UCAGC-AAGGCCAAUGCAAAUGCACUGGCCUCACUGGCUAACCAUCUGCCCAUGGGCUUGGGU-GUGGGC .........(((((((((..(--((.((.((....)).)).)))(-----((((.-.((((((.(((....))).))))))..))))).........(((....)))..))))-))))). ( -53.70) >DroYak_CAF1 6035 111 + 1 CCCAUCUCGCCCGCAUCCUCA--GCCGCUGCCGCAGCUGCUGCCG-----UCAGC-AAGGCCAAUGCAAAUGCACUGGCCUCUUUGGCUAACCAUCUGCCCAUGGGCUUGGGU-GUGGGC ........((((((((((..(--((((((((.((((...)))).)-----.))))-.((((((.(((....))).))))))....))))........(((....)))..))))-)))))) ( -55.50) >consensus CCCAUCUCACCCGCAUCCUCA__GCCGCUGCCGCAGCUGCUGCUG_____UCAGC_AAGGCCAAUGCAAAUGCACUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC_GUGAGC .................((((.((((((.((.((....)).))........(((...((((((.(((....))).))))))..))))).........(((....)))...)))).)))). (-31.20 = -30.92 + -0.28)
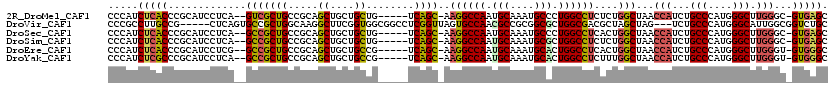
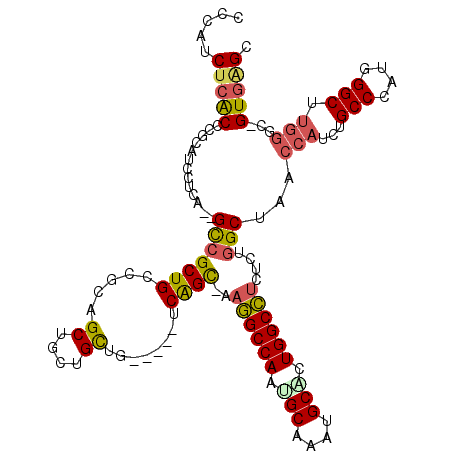
| Location | 6,885,093 – 6,885,204 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.69 |
| Mean single sequence MFE | -55.70 |
| Consensus MFE | -34.91 |
| Energy contribution | -36.32 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.11 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.79 |
| SVM RNA-class probability | 0.852212 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885093 111 - 20766785 GCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGGGCAUUUGCAUUGGCCUU-GCUGA-----CAGCAGCAGCUGCGGCAGCGAC--UGAGGAUGCGGGUGAGAUGGG .(((((-(((((......))))).....((((((....((((((((.((....)).))))))))-)))))-----).(((.(((.(((....))).)--))....)))..)))))..... ( -48.10) >DroVir_CAF1 613 112 - 1 GCAGACCGCCAAUGCCCAUGGGCAGA---CUAGCUAGCGUCGCCAGCGCCGCGGCGUUGGCACUAACCGAGGCCGCCACCGAAGCCUUGCCAGCGGCACUGAG-----CGGCAAGGCGGG .....(((((..((((....))))..---.........((((((((.((((((((((((....)))).(((((((....))..)))))))).))))).))).)-----))))..))))). ( -55.00) >DroSec_CAF1 640 111 - 1 GCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGUGAGGCCAGGGCAUUUGCAUUGGCCUU-GCUGA-----CAGCAGCAGCUGCGGCAGCGGC--UGAGGAUGCGGGUGAGAUGGG .(((((-(((((......))))).........(.(((..(((((((.((....)).))))))).-.))).-----).(((.(((((((....)))))--))....)))..)))))..... ( -55.40) >DroSim_CAF1 4753 111 - 1 GCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGCGCAUUUGCAUUGGCCUU-GCUGA-----CAGCAGCAGCUGCGGCAGCGGC--UGAGGAUGCGGGUGAGAUGGG .(((((-(((((......))))).....((((((....((((((((.((....)).))))))))-)))))-----).(((.(((((((....)))))--))....)))..)))))..... ( -54.70) >DroEre_CAF1 6086 111 - 1 GCCCAC-ACCCAAGCCCAUGGGCAGAUGGUUAGCCAGUGAGGCCAGUGCAUUUGCAUUGGCCUU-GCUGA-----CGGCAGCAGCUGCGGCAGCGGC--CGAGGAUGCGGGUGAGAUGGG .(((((-((((..(((....))).........(((((..((((((((((....)))))))))).-.))).-----((((.((.((....)).)).))--)).....)))))))...)))) ( -60.50) >DroYak_CAF1 6035 111 - 1 GCCCAC-ACCCAAGCCCAUGGGCAGAUGGUUAGCCAAAGAGGCCAGUGCAUUUGCAUUGGCCUU-GCUGA-----CGGCAGCAGCUGCGGCAGCGGC--UGAGGAUGCGGGCGAGAUGGG ((((.(-(.((..(((....)))......((((((...(((((((((((....)))))))))))-((((.-----(.((((...)))).))))))))--))))).)).))))........ ( -60.50) >consensus GCUCAC_GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGCGCAUUUGCAUUGGCCUU_GCUGA_____CAGCAGCAGCUGCGGCAGCGGC__UGAGGAUGCGGGUGAGAUGGG .((((..((((..(((....))).....((...((...((((((((.((....)).)))))))).((((......))))....(((((....))))).....))..))))))....)))) (-34.91 = -36.32 + 1.40)

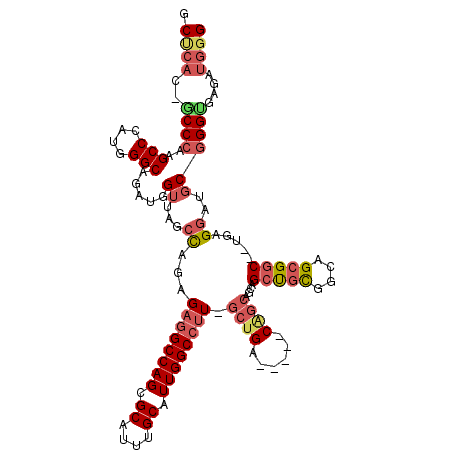
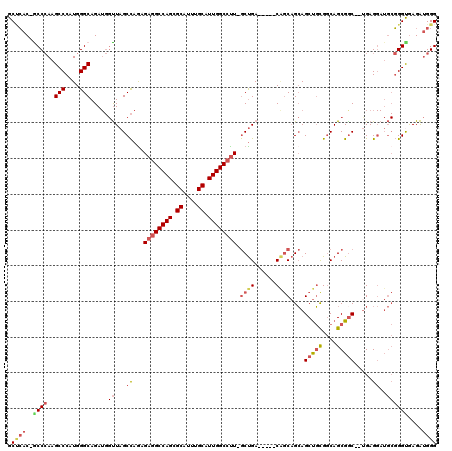
| Location | 6,885,131 – 6,885,236 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.12 |
| Mean single sequence MFE | -48.58 |
| Consensus MFE | -30.67 |
| Energy contribution | -31.48 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.775433 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885131 105 + 20766785 UGCUG-----UCAGC-AAGGCCAAUGCAAAUGCCCUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGCGGACUG--------GCCCAGCAGAUGUUCCCGCUAGCCAC .((((-----((((.-.((((((..((....))..))))))..)))))....(((((((...((((((.(.((-....))...).)--------))))))))))))........)))... ( -46.80) >DroVir_CAF1 648 117 + 1 GGUGGCGGCCUCGGUUAGUGCCAACGCCGCGGCGCUGGCGACGCUAGCUAG---UCUGCCCAUGGGCAUUGGCGGUCUGCCGGCUGCAGCGGCUGCUGCGCAAAUGUUUCCACUGGCCAC (((....)))..((((((((..((((((((.(((((((((((....(((((---..((((....))))))))).))).)))))).)).)))))(((...)))...)))..)))))))).. ( -55.50) >DroSec_CAF1 678 105 + 1 UGCUG-----UCAGC-AAGGCCAAUGCAAAUGCCCUGGCCUCACUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGCGGACUG--------GCCCAGCAGAUGUUCCCGCUAGCCAC .((((-----((((.-.((((((..((....))..))))))..)))))....(((((((...((((((.(.((-....))...).)--------))))))))))))........)))... ( -46.90) >DroSim_CAF1 4791 105 + 1 UGCUG-----UCAGC-AAGGCCAAUGCAAAUGCGCUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC-GUGAGCGGACUG--------GCCCAGCAGAUGUUCCCGCUGGCCAC ....(-----(((((-.((((((.(((....))).))))))....((.....(((((((...((((((.(.((-....))...).)--------))))))))))))...))))))))... ( -48.90) >DroEre_CAF1 6124 105 + 1 UGCCG-----UCAGC-AAGGCCAAUGCAAAUGCACUGGCCUCACUGGCUAACCAUCUGCCCAUGGGCUUGGGU-GUGGGCGGAUUG--------GCCCAGCAGAUGUUCCCGCUGGCCAC .((((-----((((.-.((((((.(((....))).))))))..))))).....((((((((((..((....))-)))))))))).)--------))(((((.(......).))))).... ( -49.70) >DroYak_CAF1 6073 105 + 1 UGCCG-----UCAGC-AAGGCCAAUGCAAAUGCACUGGCCUCUUUGGCUAACCAUCUGCCCAUGGGCUUGGGU-GUGGGCGGAUUG--------GCCCAGCAGAUGUUCCCGCUAGCCAC (((..-----...))-)((((((.(((....))).))))))...((((((.(((((((((((......)))..-.(((((......--------)))))))))))).....).)))))). ( -43.70) >consensus UGCUG_____UCAGC_AAGGCCAAUGCAAAUGCACUGGCCUCUCUGGCUAACCAUCUGCCCAUGGGCUUGGGC_GUGAGCGGACUG________GCCCAGCAGAUGUUCCCGCUAGCCAC .................((((((.(((....))).))))))...((((((.((((((((...(((((...........................)))))))))))).....).)))))). (-30.67 = -31.48 + 0.81)

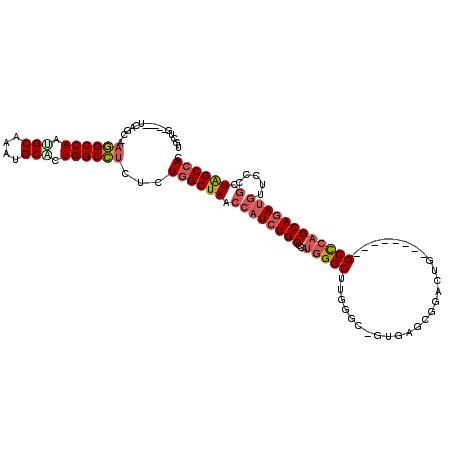
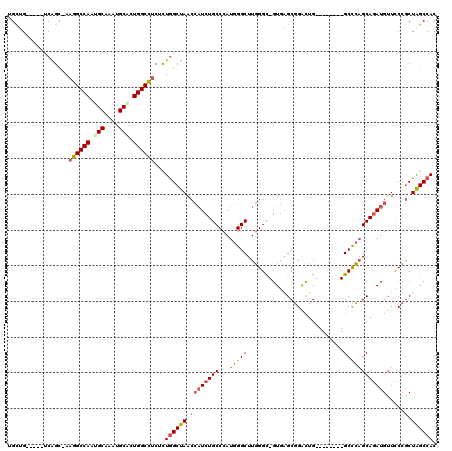
| Location | 6,885,131 – 6,885,236 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.12 |
| Mean single sequence MFE | -52.02 |
| Consensus MFE | -35.03 |
| Energy contribution | -36.00 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.923661 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885131 105 - 20766785 GUGGCUAGCGGGAACAUCUGCUGGGC--------CAGUCCGCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGGGCAUUUGCAUUGGCCUU-GCUGA-----CAGCA .((((((((((......))))).)))--------))....(((...-(((((......))))).....((((((....((((((((.((....)).))))))))-)))))-----)))). ( -47.40) >DroVir_CAF1 648 117 - 1 GUGGCCAGUGGAAACAUUUGCGCAGCAGCCGCUGCAGCCGGCAGACCGCCAAUGCCCAUGGGCAGA---CUAGCUAGCGUCGCCAGCGCCGCGGCGUUGGCACUAACCGAGGCCGCCACC ((((((((((....)))).(((((((....)))))(((.(((.....)))..((((....))))..---...))).))...((((((((....)))))))).........)))))).... ( -57.40) >DroSec_CAF1 678 105 - 1 GUGGCUAGCGGGAACAUCUGCUGGGC--------CAGUCCGCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGUGAGGCCAGGGCAUUUGCAUUGGCCUU-GCUGA-----CAGCA (((((((((.....((((((((((((--------..(..((....)-)..)..))))...)))))))))))))))((..(((((((.((....)).))))))).-.))..-----..)). ( -49.10) >DroSim_CAF1 4791 105 - 1 GUGGCCAGCGGGAACAUCUGCUGGGC--------CAGUCCGCUCAC-GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGCGCAUUUGCAUUGGCCUU-GCUGA-----CAGCA .((((((((((......))))).)))--------))....(((...-(((((......))))).....((((((....((((((((.((....)).))))))))-)))))-----)))). ( -49.70) >DroEre_CAF1 6124 105 - 1 GUGGCCAGCGGGAACAUCUGCUGGGC--------CAAUCCGCCCAC-ACCCAAGCCCAUGGGCAGAUGGUUAGCCAGUGAGGCCAGUGCAUUUGCAUUGGCCUU-GCUGA-----CGGCA ...(((.((.....((((((((((((--------......))))).-..(((......))))))))))....))(((..((((((((((....)))))))))).-.))).-----.))). ( -56.30) >DroYak_CAF1 6073 105 - 1 GUGGCUAGCGGGAACAUCUGCUGGGC--------CAAUCCGCCCAC-ACCCAAGCCCAUGGGCAGAUGGUUAGCCAAAGAGGCCAGUGCAUUUGCAUUGGCCUU-GCUGA-----CGGCA .((((((((.....((((((((((((--------......))))).-..(((......))))))))))))))))))..(((((((((((....)))))))))))-((...-----..)). ( -52.20) >consensus GUGGCCAGCGGGAACAUCUGCUGGGC________CAGUCCGCUCAC_GCCCAAGCCCAUGGGCAGAUGGUUAGCCAGAGAGGCCAGCGCAUUUGCAUUGGCCUU_GCUGA_____CAGCA .((((.((((((.......((.((((..........))))))......)))..(((....))).....))).))))..((((((((.((....)).)))))))).((((......)))). (-35.03 = -36.00 + 0.98)

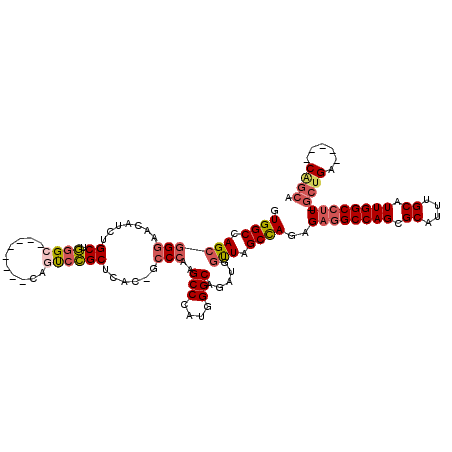
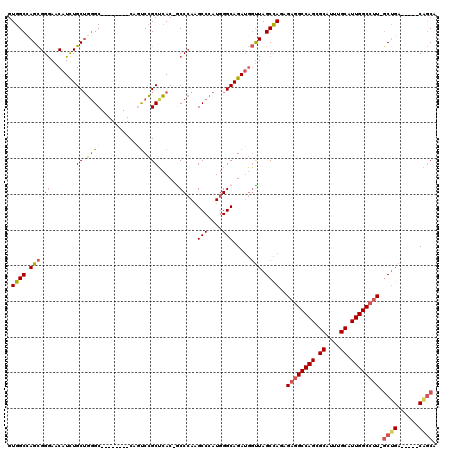
| Location | 6,885,204 – 6,885,316 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.63 |
| Mean single sequence MFE | -52.95 |
| Consensus MFE | -34.96 |
| Energy contribution | -37.47 |
| Covariance contribution | 2.50 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.548759 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885204 112 - 20766785 UUGCGCUGCCGGUGCUGCAACCAGGCGGCGUGAUCGUAGGUGGCAGGGGAAGACCAGCAGCCACUGCCGCCGCCUUGAGCGUGGCUAGCGGGAACAUCUGCUGGGC--------CAGUCC (..((((((((((......))).)))))))..)(((.(((((((.((......)).((((...)))).))))))))))((.((((((((((......))))).)))--------)))).. ( -54.10) >DroVir_CAF1 725 102 - 1 C---------CGUGCUGCAGUUGGG---------CGUGGACGUCAGCGGCAGUCCAGCGGCAGCUGCGGCCGCUUUCAGCGUGGCCAGUGGAAACAUUUGCGCAGCAGCCGCUGCAGCCG .---------((.((((((((...(---------(.(((((((.....)).)))))))(((.(((((((((((.......))))))((((....))))...))))).))))))))))))) ( -57.00) >DroSec_CAF1 751 112 - 1 UUGCGCUUCCGGUGCUGCAACCAGGCGGCGUGAUCGUGGGUGGCAGGGGAAGACCAGCAGCCACUGCCGCCGCCUUGAGCGUGGCUAGCGGGAACAUCUGCUGGGC--------CAGUCC ..((((((..(((......)))((((((((.(...((((.((.(.((......)).))).))))..))))))))).))))))(((((((((......))))).)))--------)..... ( -51.30) >DroSim_CAF1 4864 112 - 1 UUGCGCUUCCGGUGCUGCAACCAGGCGGCGUGAUCGUGGGUGGCAGGGGAAGACCAGCAGCCACUGCCGCCGCCUUGAGCGUGGCCAGCGGGAACAUCUGCUGGGC--------CAGUCC ..((((((..(((......)))((((((((.(...((((.((.(.((......)).))).))))..))))))))).))))))(((((((((......))))).)))--------)..... ( -53.60) >DroEre_CAF1 6197 112 - 1 CUGCGCUUCCGGUGCUGCAACCAGGCGGCGUAAUCGUAGGUGGCAGAGGUAGACCAGCAGCCACUGCCGCCGCCUUGAGCGUGGCCAGCGGGAACAUCUGCUGGGC--------CAAUCC ..((((((..((((((((......))))).........((((((((.(((.(.....).))).)))))))))))..))))))(((((((((......))))).)))--------)..... ( -54.80) >DroYak_CAF1 6146 112 - 1 UUACGCUUCCGGUGCUGCAACCAGGCGGCGUGAUCGUAGGUGGCAGAGGAAGACCAGCAGCCACUGCCGCAGCCUUGAGCGUGGCUAGCGGGAACAUCUGCUGGGC--------CAAUCC .....(((((....((((.(((..((((.....)))).))).)))).))))).(((((((....(.((((((((........)))).)))).)....)))))))..--------...... ( -46.90) >consensus UUGCGCUUCCGGUGCUGCAACCAGGCGGCGUGAUCGUAGGUGGCAGGGGAAGACCAGCAGCCACUGCCGCCGCCUUGAGCGUGGCCAGCGGGAACAUCUGCUGGGC________CAGUCC ..((((((..((((((((......))))).........((((((((.((..(.....)..)).)))))))))))..))))))(.((((((((....)))))))).).............. (-34.96 = -37.47 + 2.50)

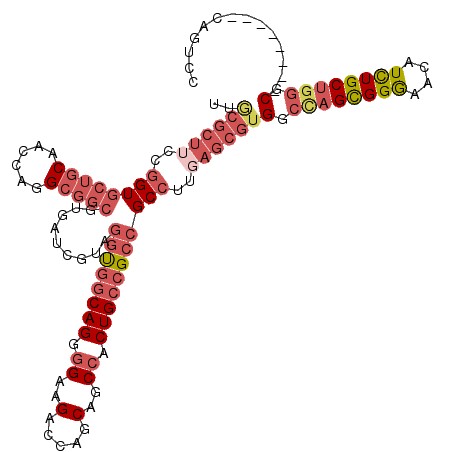
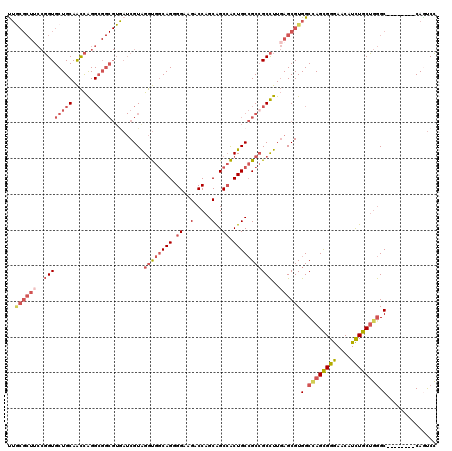
| Location | 6,885,316 – 6,885,436 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.00 |
| Mean single sequence MFE | -44.90 |
| Consensus MFE | -39.22 |
| Energy contribution | -39.45 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.534579 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6885316 120 + 20766785 CCAACAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCGGACAACUUCAAGAAGGAACUGACAGCGGCUGCGGCAGCUGGAGCACCUUUGGGAUUGUUCCAGAACC ....((((..(((((((((((((........((((((......))))))..)))))..(((....))).))))))))...)))).((...((((((((((....))..))))))))..)) ( -47.90) >DroVir_CAF1 827 117 + 1 CCAA---UAAUGUGGGCUGUUUGAUUAUCGAGCCGCAUCUGUUUGCCGCCGCGGACAACUUCAAGAAGGAGCUGAGCGCAGCUGCAGCGGCCGGUGCGCCGCUGGGUCUGUUCCAGAAUU ....---.......(((.(((((.....))))).(((((.....(((((.((......(((....))).(((((....))))))).))))).)))))))).(((((.....))))).... ( -39.60) >DroSec_CAF1 863 120 + 1 CCAAUAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCCGACAACUUUAAGAAGGAACUGACAGCGGCUGCGGCUGCUGGAGCACCUCUGGGAUUGUUCCAGAACC ......((..((((((((.((........))((((((......))))))))))))))..................(((((((....)))))))..))...(((((((...)))))))... ( -44.80) >DroSim_CAF1 4976 120 + 1 CCAACAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCGGACAACUUCAAGAAGGAACUGACAGCGGCUGCGGCUGCUGGAGCACCUCUGGGAUUGUUCCAGAACC ....((((..(((((((((((((........((((((......))))))..)))))..(((....))).))))))))...)))).((...((((((((((....))..))))))))..)) ( -47.90) >DroEre_CAF1 6309 120 + 1 CCAACAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCGGACAACUUCAAGAAGGAACUGACAGCGGCUGCGGCUGCUGGAGCACCACUGGGAUUGUUUCAGAACC ....((((..(((((((((((((........((((((......))))))..)))))..(((....))).))))))))...)))).((...((((((((((....))..))))))))..)) ( -44.60) >DroYak_CAF1 6258 120 + 1 CCAACAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCGGACAACUUCAAGAAGGAACUGACAGCGGCUGCGGCUGCUGGAGCACCACUGGGAUUGUUUCAGAACC ....((((..(((((((((((((........((((((......))))))..)))))..(((....))).))))))))...)))).((...((((((((((....))..))))))))..)) ( -44.60) >consensus CCAACAGCAAUGUCGGUUGUCUGAUCAUCGAGCCGCAUCUCUUUGCGGCGGCGGACAACUUCAAGAAGGAACUGACAGCGGCUGCGGCUGCUGGAGCACCUCUGGGAUUGUUCCAGAACC ....((((..(((((((((((((........((((((......))))))..)))))..(((....))).))))))))...)))).((...((((((((((....))..))))))))..)) (-39.22 = -39.45 + 0.23)

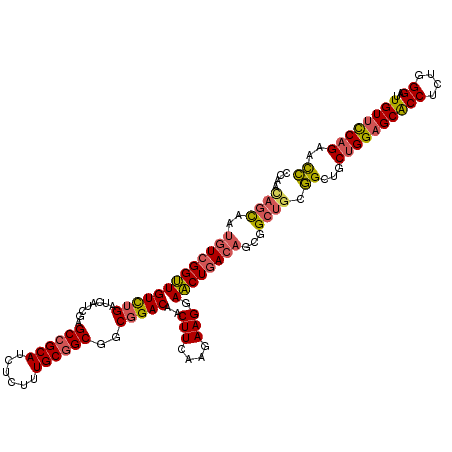
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:14:23 2006