
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,827,425 – 6,827,595 |
| Length | 170 |
| Max. P | 0.804866 |

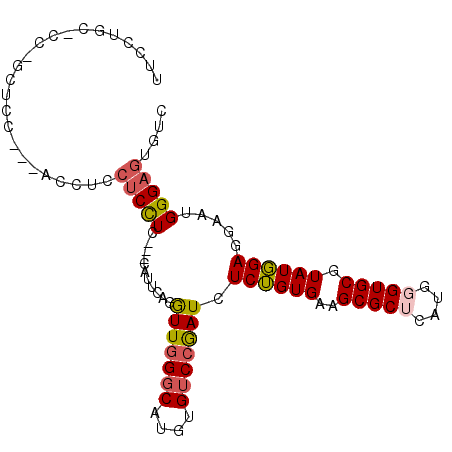
| Location | 6,827,425 – 6,827,521 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 76.34 |
| Mean single sequence MFE | -35.77 |
| Consensus MFE | -19.24 |
| Energy contribution | -19.55 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.804866 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6827425 96 + 20766785 UUCCUGCCGCCGCUCC---ACCUCCUCCUC---CAUUCAGGUUGGGCAUAUGUCCGAUCUCUGUGAAGCGCUCAUGGGUGCGUAUGGAGGAAUGGGAGUGUC ..........((((((---...((((((..---(((..(((((((((....)))))))))..)))..(((((....)))))....))))))...)))))).. ( -42.70) >DroPse_CAF1 1591 84 + 1 CGCCU---------------CCACCUCCUC---CAGUCAUGUUGGGCAUGUGUCCGAUCUCUGUGAAGCGCUCGUGUGUGCGUAUGGAAGAGUGGGAGUGGC (((..---------------((((.((.((---((.(((((((((((....)))))))....)))).((((......))))...)))).))))))..))).. ( -32.90) >DroSec_CAF1 364 99 + 1 UUCCUGCUCCACCUCC---ACCUCCUCCUCCACCAUUCAGGUUGGGCAUAUGUCCGAUCUCUGUGAAGCGCUCAUGGGUGCGUAUGGAGGAAUGGGAGUGUC .........(((..((---(..((((((.....(((..(((((((((....)))))))))..)))..(((((....)))))....)))))).)))..))).. ( -40.60) >DroYak_CAF1 923 96 + 1 UUCCAGCCCCUGCUCC---ACCUCCUCCUC---CAUUCAGGUUGGGCAUGUGUCCGAUCUCUGUGAAGCGCUCAUGGGUGCGUAUGGACGAAUGGGAGUGUC ...........(((((---...((.(((..---(((..(((((((((....)))))))))..)))..(((((....)))))....))).))...)))))... ( -34.30) >DroMoj_CAF1 298 93 + 1 UUACAGAUCCAGUUCCAGUUGCUCCACUUC---------CAUUUGGCAUGUGGCCAAUUUCGGUGAAGCGCUCGUGGGUGCGUAUCGAGGAGUGGGAAUGAC ...........(((((....(((((.....---------...(((((.....)))))..((((((..(((((....))))).))))))))))).)))))... ( -31.20) >DroPer_CAF1 944 84 + 1 CGCCU---------------CCACCUCCUC---CAGUCAUGUUGGGCAUGUGUCCGAUCUCUGUGAAGCGCUCGUGUGUGCGUAUGGAAGAGUGGGAGUGGC (((..---------------((((.((.((---((.(((((((((((....)))))))....)))).((((......))))...)))).))))))..))).. ( -32.90) >consensus UUCCUGC_CC_GCUCC___ACCUCCUCCUC___CAUUCAGGUUGGGCAUGUGUCCGAUCUCUGUGAAGCGCUCAUGGGUGCGUAUGGAGGAAUGGGAGUGUC ........................(((((...........(((((((....))))))).((((((..(((((....))))).)))))).....))))).... (-19.24 = -19.55 + 0.31)

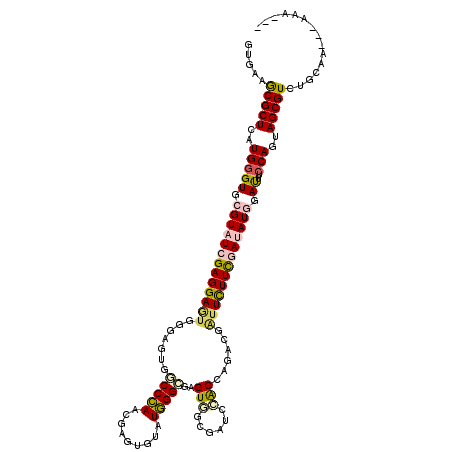
| Location | 6,827,481 – 6,827,595 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.52 |
| Mean single sequence MFE | -39.57 |
| Consensus MFE | -27.88 |
| Energy contribution | -28.30 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.632880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
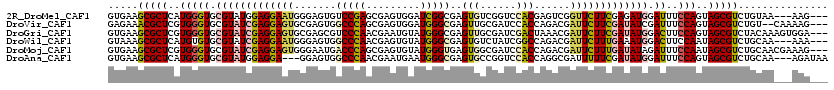
>2R_DroMel_CAF1 6827481 114 + 20766785 GUGAAGCGCUCAUGGGUGCGUAUGGAGGAAUGGGAGUGUCCGAGCGAGUGGAUCGGCGAGUGUCGGUCCACGAGUCGGUUCUUCGAGAUGGAUUUCCAGUAGCGUCUGUAA---AAG--- .....(((((....))))).((((((.(....((((...((((.(..((((((((((....))))))))))..)))))..))))....(((....)))....).)))))).---...--- ( -42.70) >DroVir_CAF1 362 115 + 1 GAGAAACGCUCGUGGGUGCGUAUCGAGGAGUGCGAGUGGCCCAGCGAGUGGAUGGGCGAGUUGCGAUCCACCAGACGAUUCUUCGAUAUCGAUUUCCAGUAGCGUCUGU--CAAAAG--- ((.(.(((((..((((..(((((((((((((((((.(.(((((.(.....).))))).).))))(.((.....))).))))))))))).))...))))..))))).).)--).....--- ( -44.10) >DroGri_CAF1 390 117 + 1 GUGAAGCGCUCGUGGGUGCGUAUCGAGGAGUGCGAGCGUCCCAACGAAUGUAUGGGCGAGUUGCGAUCGACUAAACGAUUCUUCGAUAUGGACUUCCAGUAGCGUCUACAAAGUGGA--- (((..(((((((..(((.(((((((((((.(((((.((.((((((....)).))))))..)))))((((......))))))))))))))).)))..).).))))).)))........--- ( -41.80) >DroWil_CAF1 2047 114 + 1 GUAAAGCGCUCAUGUGUGCGUAUCGAGGAAUGGGAGUGGCCCAACGAGUGUAUGGGCGAGUGUCUAUCGGCCAGACGAUUCUUUGAAAUGGACUUCCAAUAGCGUCUGCAA---AAA--- (((..(((((..((.((.(((.((((((((((((.....)))......(((.(((.(((.......))).))).)))))))))))).))).))...))..))))).)))..---...--- ( -35.20) >DroMoj_CAF1 351 117 + 1 GUGAAGCGCUCGUGGGUGCGUAUCGAGGAGUGGGAAUGACCCAGCGAGUGUAUGGGUGAGUGGCGAUCCACCAGACGAUUCUUUGAUAUAGAUUUCCAAUAGCGUCUGCAACGAAAG--- ((..((((((..((((..(((((((((((((.(...(((((((((....)).)))))..((((....))))))..).)))))))))))).)...))))..)))).))...)).....--- ( -38.00) >DroAna_CAF1 549 114 + 1 GUGAAGCGCUCAUGGGUGCGUAUGGAGGA---GGAGUGGCCCAACGAAUGAAUGGGCGAGUGCCGGUCCACCAGGCGAUUUUUCGAUAUGGAUUUCCAGUAGCGUCUGCAA---AGAUAA .((.((((((..(((((.(((((.(((((---(.(.(.(((((.........))))).).)(((((....)).)))..)))))).))))).))..)))..)))).)).)).---...... ( -35.60) >consensus GUGAAGCGCUCAUGGGUGCGUAUCGAGGAGUGGGAGUGGCCCAACGAGUGUAUGGGCGAGUGGCGAUCCACCAGACGAUUCUUCGAUAUGGAUUUCCAGUAGCGUCUGCAA___AAA___ .....(((((..(((((.(((((((((((((.......(((((.........)))))..(((......)))......))))))))))))).))..)))..)))))............... (-27.88 = -28.30 + 0.42)
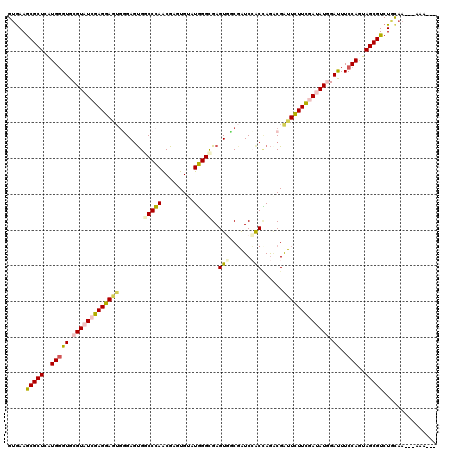
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:14:04 2006