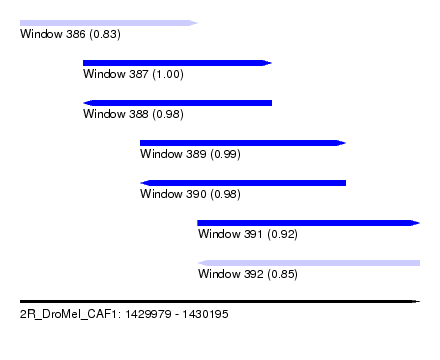
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,429,979 – 1,430,195 |
| Length | 216 |
| Max. P | 0.995704 |

| Location | 1,429,979 – 1,430,075 |
|---|---|
| Length | 96 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.74 |
| Mean single sequence MFE | -29.42 |
| Consensus MFE | -13.20 |
| Energy contribution | -14.12 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.45 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.830022 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1429979 96 + 20766785 UUUGACGAGGCAUCAUACCCGGCUUAUGGCAUAU-------GGCAUAUGCUUUUAUGUGUG----UC----CAUACAUACAUAUAU---------AGUAUGUGUGUUGCAGCUGCAUAUG .............((((..(((((....(((...-------.(((((((((..((((((((----(.----...)))))))))...---------)))))))))..))))))))..)))) ( -28.90) >DroSec_CAF1 48396 104 + 1 UUUGACGAGGCAUCAUACCCGGCUUAUGGCAUAU-------GGCAUAUGAUUGCAUGUGUGUGUGUCCCUACAUACAUACAUAUAU---------AGUGUGUGUGUUGCAGCUGCAUAUG .............((((..(((((...(((((((-------(.((((((....))))))))))))))...((((((((((......---------.))))))))))...)))))..)))) ( -30.80) >DroSim_CAF1 53483 120 + 1 UUUGACGAGGCAUCAUACCCGGCUUAUGGCAUAUGGCAUAUGGCAUAUGCUUGCAUGUGUGUGUGUCCCUACAUACAUACAUAUAUAGUAUGUAUAGUGUGUGUGUUGCAGCUGCAUAUG ......((.(((.(((((...((....((((((((.(....).)))))))).))..)))))))).))...((((((((((.(((((.....)))))))))))))))(((....))).... ( -40.40) >DroEre_CAF1 46510 92 + 1 UUUGACGAGGCAUCAGACCCGGUUUAUGGCAUGU-------GGCAUAUGGAUAUAUGUGUGUGUGUC------------UAUAUAU---------AGCAUGUGUGUUGCAGCUGCAUAUG ........(.(((.((((...))))))).)(((.-------.((((((((((((((....)))))))------------)))))..---------.(((.......))).))..)))... ( -25.30) >DroYak_CAF1 51368 86 + 1 UUUGACGAGGCAUCGGACCCAGUUUAUGGCAUGU-------GGCAUAUGGAUAU------GUCUGUU------------UAUAUAU---------AGUUUGUAUGUUGCAGCUGCAAAUG .....(((....)))......((((..(((.((.-------.(((((..(((((------((.....------------...))))---------.)))..)))))..)))))..)))). ( -21.70) >consensus UUUGACGAGGCAUCAUACCCGGCUUAUGGCAUAU_______GGCAUAUGCUUAUAUGUGUGUGUGUC____CAUACAUACAUAUAU_________AGUAUGUGUGUUGCAGCUGCAUAUG .(((.(((.(((.((((((((.....))).............(((((((....)))))))....................................)))))))).))))))......... (-13.20 = -14.12 + 0.92)

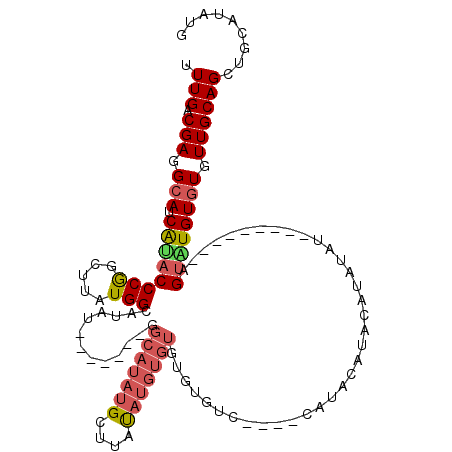
| Location | 1,430,013 – 1,430,115 |
|---|---|
| Length | 102 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.98 |
| Mean single sequence MFE | -32.50 |
| Consensus MFE | -21.03 |
| Energy contribution | -23.18 |
| Covariance contribution | 2.15 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.13 |
| Structure conservation index | 0.65 |
| SVM decision value | 2.61 |
| SVM RNA-class probability | 0.995704 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430013 102 + 20766785 -GGCAUAUGCUUUUAUGUGUG----UC----CAUACAUACAUAUAU---------AGUAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU -.(((((((((..((((((((----(.----...)))))))))...---------)))))))))..(((....)))..((((((((((((..........))))))))...))))..... ( -32.20) >DroSec_CAF1 48430 110 + 1 -GGCAUAUGAUUGCAUGUGUGUGUGUCCCUACAUACAUACAUAUAU---------AGUGUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU -.(((......)))((((((((((((....))))))))))))....---------((..((((.((((.((((((..........))).))).)))).))))..)).............. ( -36.10) >DroSim_CAF1 53523 120 + 1 UGGCAUAUGCUUGCAUGUGUGUGUGUCCCUACAUACAUACAUAUAUAGUAUGUAUAGUGUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU (((((((((((...((((((((((((....))))))))))))....)))))))..((..((((.((((.((((((..........))).))).)))).))))..)).....))))..... ( -40.30) >DroEre_CAF1 46544 98 + 1 -GGCAUAUGGAUAUAUGUGUGUGUGUC------------UAUAUAU---------AGCAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU -(((((((((((((((....)))))))------------)))))..---------((((((((.((((.((((((..........))).))).)))).)))))))).....)))...... ( -34.00) >DroYak_CAF1 51402 92 + 1 -GGCAUAUGGAUAU------GUCUGUU------------UAUAUAU---------AGUUUGUAUGUUGCAGCUGCAAAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU -.(((((..(((((------((.....------------...))))---------.)))..)))))(((....)))..((((((((((((..........))))))))...))))..... ( -19.90) >consensus _GGCAUAUGCUUAUAUGUGUGUGUGUC____CAUACAUACAUAUAU_________AGUAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAU .(((((.....((((((((((..............))))))))))..........((((((((.((((.((((((..........))).))).)))).))))))))...)))))...... (-21.03 = -23.18 + 2.15)

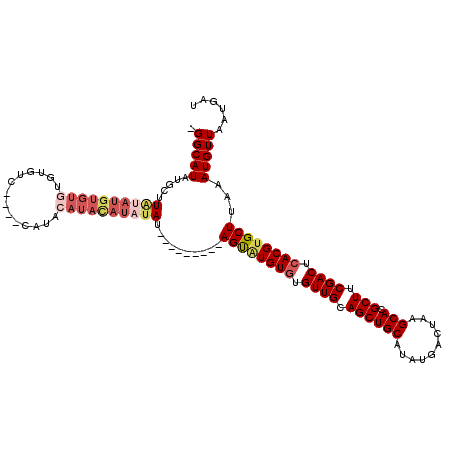
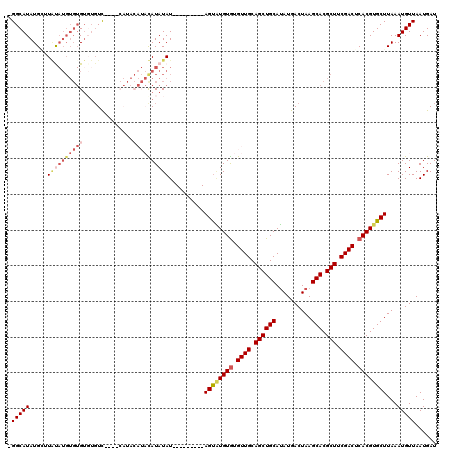
| Location | 1,430,013 – 1,430,115 |
|---|---|
| Length | 102 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.98 |
| Mean single sequence MFE | -25.48 |
| Consensus MFE | -15.28 |
| Energy contribution | -15.28 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982192 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430013 102 - 20766785 AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACAUACU---------AUAUAUGUAUGUAUG----GA----CACACAUAAAAGCAUAUGCC- ...........((((((((((........)))))))))).(((((((...........((((((.---------......))))))(((----..----....)))....)))))))..- ( -28.00) >DroSec_CAF1 48430 110 - 1 AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACU---------AUAUAUGUAUGUAUGUAGGGACACACACACAUGCAAUCAUAUGCC- ...........((((((((((........)))))))))).....(((....)))...........---------.((((((..(((((((..(......)..)))))))..))))))..- ( -27.10) >DroSim_CAF1 53523 120 - 1 AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACUAUACAUACUAUAUAUGUAUGUAUGUAGGGACACACACACAUGCAAGCAUAUGCCA ............(((((((((........)))))))))(.(((((((...((((...........((((((((.......))))))))((..(......)..)).)))).))))))).). ( -33.10) >DroEre_CAF1 46544 98 - 1 AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACAUGCU---------AUAUAUA------------GACACACACACAUAUAUCCAUAUGCC- ............(((((((((........)))))))))((((((((.(((((.......)).)))---------.))))).------------))).......(((((....)))))..- ( -22.50) >DroYak_CAF1 51402 92 - 1 AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUUUGCAGCUGCAACAUACAAACU---------AUAUAUA------------AACAGAC------AUAUCCAUAUGCC- ............(((((((((........)))))))))(((..((((....)))).(((.....)---------)).....------------....)))------.............- ( -16.70) >consensus AUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACU_________AUAUAUGUAUGUAUG____GACACACACACAUACAACCAUAUGCC_ ...........((((((((((........)))))))))).....(((....))).................................................................. (-15.28 = -15.28 + 0.00)

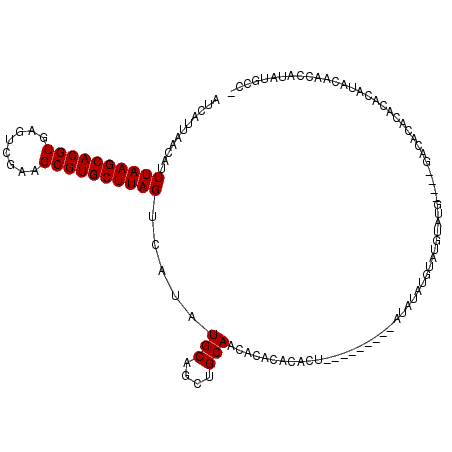
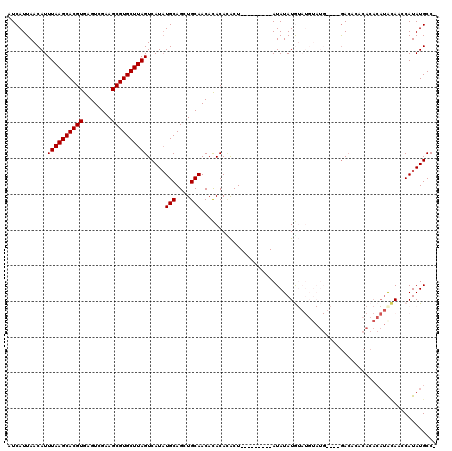
| Location | 1,430,044 – 1,430,155 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.76 |
| Mean single sequence MFE | -29.95 |
| Consensus MFE | -26.54 |
| Energy contribution | -26.62 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993747 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430044 111 + 20766785 AUAUAU---------AGUAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUC ((((..---------((((((((.((((.((((((..........))).))).)))).))))))))...))))..(((((((((.....((......))........))))).))))... ( -29.42) >DroSec_CAF1 48469 111 + 1 AUAUAU---------AGUGUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUC ..((((---------((((((((.((.....((((...((((((((((((..........))))))))...))))....(((((....).)))).))))....)).)))))))).)))). ( -28.70) >DroSim_CAF1 53563 120 + 1 AUAUAUAGUAUGUAUAGUGUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUC ..((((((((((((.(((((((.(((.(((...(((((((((((((((((..........))))))))...)))....))))))...))).))).)))....)))))))))))).)))). ( -34.10) >DroEre_CAF1 46571 111 + 1 AUAUAU---------AGCAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUC ((((..---------((((((((.((((.((((((..........))).))).)))).))))))))...))))..(((((((((.....((......))........))))).))))... ( -31.42) >DroYak_CAF1 51423 111 + 1 AUAUAU---------AGUUUGUAUGUUGCAGCUGCAAAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCUACAAGCGGAAUUACUUGCAUACUCAUAUC ..((((---------(((.((((.((.....((((...((((((((((((..........))))))))...))))....(((((....)).))).))))....)).)))).))).)))). ( -26.10) >consensus AUAUAU_________AGUAUGUGUGUUGCAGCUGCAUAUGACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUC ...............((((((((.((.....((((...((((((((((((..........))))))))...))))....(((((....)).))).))))....)).))))))))...... (-26.54 = -26.62 + 0.08)

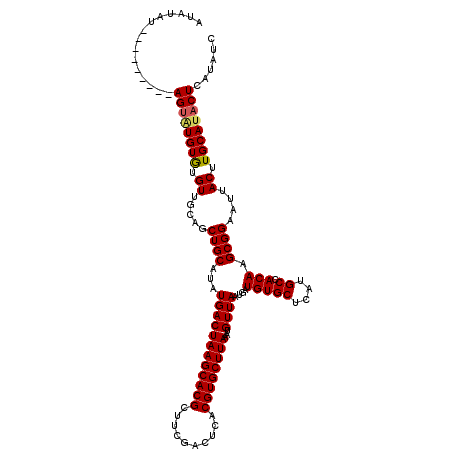
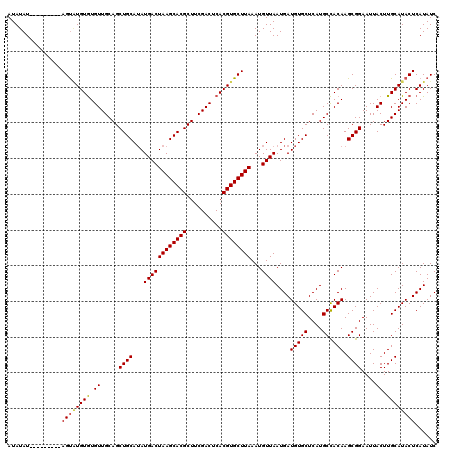
| Location | 1,430,044 – 1,430,155 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.76 |
| Mean single sequence MFE | -28.54 |
| Consensus MFE | -25.68 |
| Energy contribution | -25.68 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.979279 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430044 111 - 20766785 GAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACAUACU---------AUAUAU .((((((((((((((((......)))))((((((((.....)))).......(((((((((........)))))))))))))..(((....))).....))))))---------.))))) ( -29.50) >DroSec_CAF1 48469 111 - 1 GAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACU---------AUAUAU ....((.((.((((.((......((.((((((((((.....)))).......(((((((((........))))))))))))))).)).)))))))).))......---------...... ( -26.00) >DroSim_CAF1 53563 120 - 1 GAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACUAUACAUACUAUAUAU .(((((((((((..(((.........((((((((((.....)))).......(((((((((........)))))))))))))))(((....)))........)))...)))))).))))) ( -29.90) >DroEre_CAF1 46571 111 - 1 GAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACAUGCU---------AUAUAU .((((((((((((((((......)))))((((((((.....)))).......(((((((((........)))))))))))))..(((....))).....))))))---------.))))) ( -29.50) >DroYak_CAF1 51423 111 - 1 GAUAUGAGUAUGCAAGUAAUUCCGCUUGUAGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUUUGCAGCUGCAACAUACAAACU---------AUAUAU .......(((((.((((......))))(((((((((.....))))......((((((((((........)))))))))).........)))))..))))).....---------...... ( -27.80) >consensus GAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGUCAUAUGCAGCUGCAACACACACACU_________AUAUAU (((.((..(((((((((......))))...)))))..)).)))........((((((((((........)))))))))).....(((....))).......................... (-25.68 = -25.68 + -0.00)

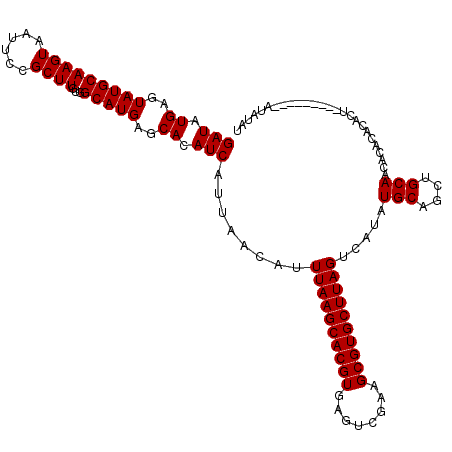
| Location | 1,430,075 – 1,430,195 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.25 |
| Mean single sequence MFE | -24.21 |
| Consensus MFE | -21.62 |
| Energy contribution | -21.72 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.922006 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430075 120 + 20766785 ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUCAUACAGACGCACUCACAGAUGCAACAUAACAUAUGUAAAG ..((((((((..........)))))))).((((((((((((((...((((...(((........)))))))...))))))))......(((........)))....))))))........ ( -25.90) >DroSec_CAF1 48500 120 + 1 ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUCAUACAGACGCACUCACAGACGCAACAUAACAUAUGUAAAG ..((((((((..........)))))))).((((((((((((((...((((...(((........)))))))...))))))))......((..........))....))))))........ ( -24.30) >DroSim_CAF1 53603 120 + 1 ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUCAUACAGACGCACUCACAGACGCAACAUAACAUAUGUAAAG ..((((((((..........)))))))).((((((((((((((...((((...(((........)))))))...))))))))......((..........))....))))))........ ( -24.30) >DroEre_CAF1 46602 120 + 1 ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUCAUACAGACCCACUCACAGACGCCACCUAACAUAUGUAAAC ..((((((((..........)))))))).((((((((((((((...((((...(((........)))))))...))))))))...((.....))............))))))........ ( -22.60) >DroYak_CAF1 51454 108 + 1 ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCUACAAGCGGAAUUACUUGCAUACUCAUAUCAUGCA------------GACGCCACCUAACAUAUGUAAAC ..((((((((..........)))))))).((((((((((((((...((((.................))))...))))))))((.------------...))....))))))........ ( -23.93) >consensus ACUAAGCACGCUUCGACUCACGUGCUUAAAUGUUAAUGAUGUGCUCAUGCCACAAGCGGAAUUACUUGCAUACUCAUAUCAUACAGACGCACUCACAGACGCAACAUAACAUAUGUAAAG ..((((((((..........)))))))).((((((((((((((...((((...(((........)))))))...))))))))...((.....))............))))))........ (-21.62 = -21.72 + 0.10)

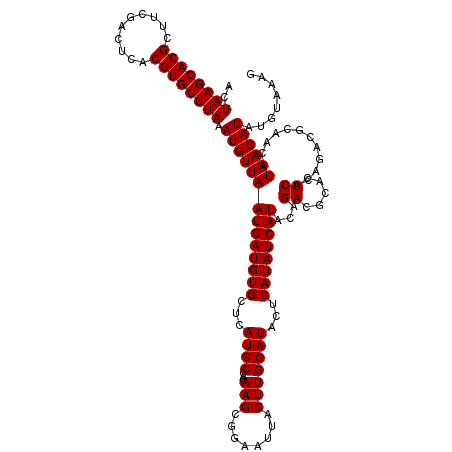
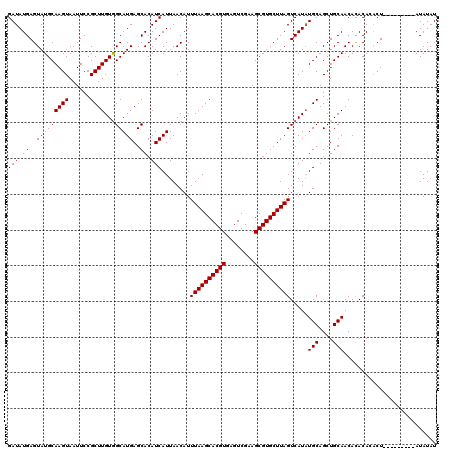
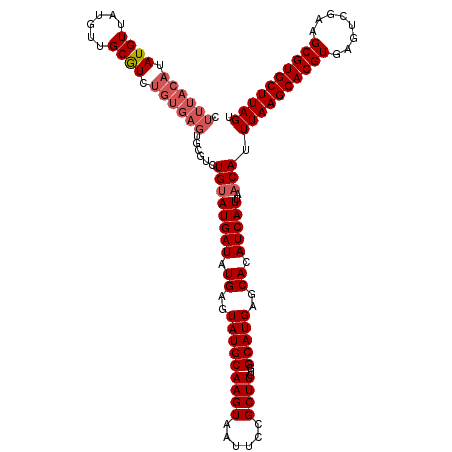
| Location | 1,430,075 – 1,430,195 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.25 |
| Mean single sequence MFE | -33.22 |
| Consensus MFE | -28.74 |
| Energy contribution | -29.98 |
| Covariance contribution | 1.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.46 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.79 |
| SVM RNA-class probability | 0.851834 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1430075 120 - 20766785 CUUUACAUAUGUUAUGUUGCAUCUGUGAGUGCGUCUGUAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU ..(((((.((((......)))).)))))(((((((.....)))(((....(((((((......))))))).)))..))))...........((((((((((........)))))))))). ( -33.60) >DroSec_CAF1 48500 120 - 1 CUUUACAUAUGUUAUGUUGCGUCUGUGAGUGCGUCUGUAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU ..(((((.((((......)))).)))))(((((((.....)))(((....(((((((......))))))).)))..))))...........((((((((((........)))))))))). ( -33.20) >DroSim_CAF1 53603 120 - 1 CUUUACAUAUGUUAUGUUGCGUCUGUGAGUGCGUCUGUAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU ..(((((.((((......)))).)))))(((((((.....)))(((....(((((((......))))))).)))..))))...........((((((((((........)))))))))). ( -33.20) >DroEre_CAF1 46602 120 - 1 GUUUACAUAUGUUAGGUGGCGUCUGUGAGUGGGUCUGUAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU ........((((((((((((..(((..((((((.(((((((.......))))).))....))))))..))).....)).))))..))))))((((((((((........)))))))))). ( -35.30) >DroYak_CAF1 51454 108 - 1 GUUUACAUAUGUUAGGUGGCGUC------------UGCAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUAGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU ........(((((((((((((((------------.....)))(((....(((((((......))))))).)))..)).))))..))))))((((((((((........)))))))))). ( -30.80) >consensus CUUUACAUAUGUUAUGUUGCGUCUGUGAGUGCGUCUGUAUGAUAUGAGUAUGCAAGUAAUUCCGCUUGUGGCAUGAGCACAUCAUUAACAUUUAAGCACGUGAGUCGAAGCGUGCUUAGU .((((((.((((......)))).))))))......((((((((.((..(((((((((......))))...)))))..)).)))))..))).((((((((((........)))))))))). (-28.74 = -29.98 + 1.24)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:31:09 2006